Matrix-unterstützte Laser-Desorption/Ionisation
Matrix-unterstützte Laser-Desorption/Ionisation (englisch Matrix-assisted Laser Desorption/Ionization, MALDI) ist ein Verfahren zur Ionisation von Molekülen. Es erwies sich seit seiner Entwicklung durch Franz Hillenkamp und Michael Karas[1] in den 1980er-Jahren als besonders effektiv für die Massenspektrometrie von großen Molekülen und Polymeren sowie Biopolymeren (z. B. Proteine), wird aber auch für die Detektion von Lipiden und Pigmenten genutzt. Der Laser wirkt nicht direkt auf die großen Moleküle (die bei direkter Einwirkung zerbrechen würden), sondern über eine Matrix, in die sie eingebettet sind.
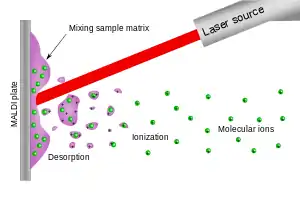
Hauptanwendungsbereich für MALDI-MS ist zumeist der Bereich der medizinischen Forschung.[2][3] Zusätzlich findet die Technik Anwendung für biologische Fragestellungen[4], wie die Untersuchung von Polymerisationsreaktionen in komplexen Gemischen ohne Probenaufbereitung in schneller, zeitlicher Abfolge[5]. In den vergangenen Jahren zudem auch in den Umweltwissenschaften. Letzteres besonders durch die Möglichkeit, hochauflösende Daten über das Paläoklima zu erhalten.[6]
Meist wird der Begriff MALDI auch als Kurzform für die MALDI-Massenspektrometrie (MALDI-MS; MALDI-TOF-MS) benutzt. Durch die hohe Massengenauigkeit und die Anwendbarkeit auf eine riesige Menge unterschiedlicher Moleküle ist die MALDI-MS in vielen Bereichen einsetzbar. Durch die eingesetzte Technik ist es zudem möglich MALDI-MS als bildgebende Methode für Gewebeschnitte einzusetzen. Hierbei lassen sich unterschiedliche Biomarker in Gewebeschnitten unterscheiden.
Das Verfahren wurde von Franz Hillenkamp und Michael Karas 1985 entwickelt. Für ein ähnliches Verfahren (Soft Laser Desorption, SLD), um die gleiche Zeit entwickelt aber erst 1987 bekanntgegeben, erhielt Koichi Tanaka 2002 den Nobelpreis. Ausschlaggebend dabei war, dass Tanaka es erstmals auf Proteine anwandte.
Funktionsweise
MALDI beruht auf der Kokristallisation von Matrix und Analyt mit einem 100- bis 100.000-fachen molaren Überschuss an Matrixmolekülen. Analytmoleküle müssen in die Kristalle der MALDI-Matrix „eingebaut“ werden, während sich die Kristalle bilden. Typischerweise erfordert die erfolgreiche Kokristallisation ein Matrix-zu-Analyt-Verhältnis von etwa 5000:1 (mol/mol). Als Matrixsubstanzen werden kleine organische Moleküle gewählt, die bei der verwendeten Laserwellenlänge (z. B. Stickstofflaser bei einer Wellenlänge von 337,1 nm) Energie stark absorbieren (z. B. Sinapinsäure, 2,5-Dihydroxybenzoesäure, α-Cyanohydroxyzimtsäure, 2,4,6-Trihydroxyacetophenon[7]). Mit kurzen, hochenergetischen Laserpulsen von 2–5 ns Pulsdauer erfolgt die Anregung, die nach Relaxation im Kristallgitter zu explosionsartigen Teilchenablösungen an der Oberfläche des Kristalls führt. Gemeinsam mit der Matrix werden dabei die eingeschlossenen Analytmoleküle mit in das Vakuum des Massenspektrometers überführt und so der massenspektrometrischen Analyse zugänglich.
Wesentlich für eine MALDI-Messung ist die Probenpräparation und das Auftragen auf den Probenteller. Hierfür gibt es verschiedene Möglichkeiten, wie zum Beispiel die Dried-Droplet-Methode (=Vermischen der Analyt- und der Matrixlösung, mit anschließendem Verdampfen der verwendeten Lösungsmittel) oder die Dünnschichtpräparation.
Der Ionisationsmechanismus bei MALDI ist noch nicht vollständig verstanden. Es gibt zurzeit zwei favorisierte Möglichkeiten:
- Ausgangspunkt für die Messung ist der Analyt in seiner durch den pH-Wert bestimmten Form in der Matrix (wahrscheinlich in einer Lösungsmitteltasche im Kristall). Durch einen Laserpuls werden Cluster aus der Oberfläche herausgelöst. Die Cluster enthalten den Analyten sowie entsprechende Gegenionen und einen Überschuss an Matrix. Im weiteren Verlauf kommt es zur Verdampfung der Matrix und zur Desolvatisierung des Analyten. Dabei werden entsprechende Gegenionen neutralisiert und verdampfen ebenfalls. Anschließend liegt ein mehrfach geladenes Analyt-Ion vor, das durch Elektroneneinfang weitere Ladungen verliert und zum Schluss nur noch einfach geladen detektiert wird. Diese Theorie der Ladungstrennung lässt sich zurzeit noch nicht nachweisen.
- Eine Photoionisation würde zu den gleichen Ergebnissen führen, wobei es darüber in der Literatur Diskussionen gibt, ob die Energie dafür ausreichen würde.
Einzelnachweise
- Michael. Karas, Franz. Hillenkamp: Laser desorption ionization of proteins with molecular masses exceeding 10,000 daltons. In: Analytical Chemistry. Band 60, Nr. 20, 15. Oktober 1988, ISSN 0003-2700, S. 2299–2301, doi:10.1021/ac00171a028.
- Pierre Chaurand, Markus Stoeckli, Richard M. Caprioli: Direct Profiling of Proteins in Biological Tissue Sections by MALDI Mass Spectrometry. In: Analytical Chemistry. Band 71, Nr. 23, Dezember 1999, ISSN 0003-2700, S. 5263–5270, doi:10.1021/ac990781q.
- Markus Stoeckli, Pierre Chaurand, Dennis E. Hallahan, Richard M. Caprioli: Nature Medicine. Band 7, Nr. 4, S. 493–496, doi:10.1038/86573.
- Astrid Vieler, Christian Wilhelm, Reimund Goss, Rosmarie Süß, Jürgen Schiller: The lipid composition of the unicellular green alga Chlamydomonas reinhardtii and the diatom Cyclotella meneghiniana investigated by MALDI-TOF MS and TLC. In: Chemistry and Physics of Lipids. Band 150, Nr. 2, Dezember 2007, S. 143–155, doi:10.1016/j.chemphyslip.2007.06.224.
- Ch. Krösche, M.G. Peter: Detection of Melanochromes by MALDI-TOF Mass Spectrometry. In: Tetrahedron. Band 52, Nr. 11, 1996, S. 3947–3952.
- Lars Wörmer, Marcus Elvert, Jens Fuchser, Julius Sebastian Lipp, Pier Luigi Buttigieg: Ultra-high-resolution paleoenvironmental records via direct laser-based analysis of lipid biomarkers in sediment core samples. In: Proceedings of the National Academy of Sciences. Band 111, Nr. 44, 4. November 2014, S. 15669–15674, doi:10.1073/pnas.1405237111, PMID 25331871, PMC 4226096 (freier Volltext).
- Maldi MS: A Practical Guide to Instrumentation, Methods and Applications. John Wiley & Sons, 2007, ISBN 978-3-527-61047-1 (books.google.de).