Kerngerüst-/Kernmatrixanheftungsregionen
Der Begriff Kerngerüst-/Kernmatrixanheftungsregionen (engl. scaffold/matrix attachment region, S/MAR), auch scaffold-attachment region (SAR) oder matrix-associated element (MAR) genannt, beschreibt DNA-Sequenzen von eukaryotischen Chromosomen bzw. der 30-nm-Faser oder des Chromatins, an der sich die Kernmatrix anheftet. Man kann S/MARs als architektonische DNA-Komponenten beschreiben die sowohl einen organisatorischen Einfluss auf das Genom haben als auch die Aktivierung und Inaktivierung des eukaryotischen Genomabschnittes beeinflussen und ebenfalls die strukturelle Organisation vermitteln, d. h. die Kondensation, des Chromatins innerhalb des Zellkerns. Diese Elemente bilden Ankerpunkte der DNA für das Chromatingerüst und dienen dazu das Chromatin in Strukturdomänen anzuordnen. Studien über individuelle Gene führten zu der Schlussfolgerung, dass die durch S/MARs vermittelte, komplexe und dynamische Organisation des Chromatins bei der Regulation der Genexpression eine wichtige Rolle spielt.
Überblick
Es ist seit vielen Jahren bekannt, dass ein polymeres Geflecht, eine sogenannte Kernmatrix oder Kerngerüst, eine essentielle Komponente von eukaryotischen Zellkernen ist. Das nukleare Skelett agiert als dynamische Unterstützung für zahlreiche spezifische Ereignisse bezüglich der Transkription der sich auszubreitenden genetischen Information.
S/MARs erfassen keine kartographisch zufälligen Abschnitte im Genom. Sie treten an den Seiten von transkribierten Regionen, in 5'-Introns und an Genbruchpunkt-Clusterregionen (gene breakpoint cluster regions - BCRs) auf. Als Verbindungspunkte für gemeinsame Kernstrukturproteine (z. B. die Lamine der Lamina oder Matrine wie ARBP/meCP2,[1] HMG 1, HMG 2,[2] Nukleolin oder Histone), sind S/MARs erforderlich für eine authentische und effiziente Replikation, Transkription, Rekombination und Kondensation der Chromosomen. S/MARs haben keine offensichtliche Consensussequenz. Obwohl Urtypelemente aus Längen mit mehreren hundert A-T reichen Basenpaaren bestehen, ist die Gesamtzusammensetzung definitiv nicht die primäre Variable, die maßgebend für ihre Aktivität ist. Stattdessen erfordert ihre Funktion ein Muster aus AT-Bereichen, das lokale Strangtrennungen unter Torsionsspannungen ermöglicht. Die Ankerpunkte bzw. Kontaktpunkte werden durch MAR-Proteine vermittelt. Diese Stellen der DNA haben eine Länge von 100 bis 1000 Basenpaaren und einen A-T-Gehalt von 70 %. Außerdem sind sie aufgrund der A-T-Basenpaarung mit nur zwei Wasserstoffbrücken leicht gebogen, sodass die Verbindung der DNA und der Matrix unterstützt wird.[3]
Bioinformatische Ansätze untermauern die These, dass S/MARs nicht nur eine bestimmte transkriptionelle Einheit (Chromatindomäne bzw. DNA-Abschnitt der mittels Nukleosomen kondensiert vorliegt) von deren Nachbarn trennen, sondern auch Plattformen bieten für das Präzipitieren von Faktoren die transkriptionelle Reaktionen innerhalb einer gegebenen Domäne ermöglichen. Die verschärfte Neigung zur Trennung der DNA-Stränge (das sogenannte: ‘stress induced duplex destabilization’ potential, SIDD[4]) kann der Bildung von Sekundärstrukturen wie z. B. Cruciformen oder Schlupfstrukturen dienen, die maßgebliche Merkmale für eine Gruppe von Enzymen (DNAsen, Topoisomerase, Poly(ADP-ribosyl) Polymerase und Enzyme des Histon-acetylierungs und DNA-Methylierungs Komplexes) sind. S/MARs sind entweder als konstitutiv (agierend als permanente Domänbegrenzung in allen Zelltypen) oder als fakultativ (zelltyp- oder funktionsbezogen) kategorisiert worden, abhängig von ihren dynamischen Eigenschaften.
Während die Anzahl der S/MARs im menschlichen Genom pauschal auf 64.000 geschätzt wurde (Chromatindomänen) plus ergänzend 10.000 (Replikations Fokusse), hatte in 2007 immer noch nur ein kleiner Bruchteil (559 für alle Eukaryoten) die Norm für die Kriterien zur Anmerkung in den S/MARt-Datenbanken[5] erfüllt.
Umstandsabhängige Eigenschaften von S/MARs
Derzeitige Ansichten über die Kernmatrix beschreiben sie als dynamisches Objekt, das seine Eigenschaften den entsprechenden Erfordernissen des Zellkerns anpasst – genauso wie das Cytoskelett passt sie ihre Struktur und Funktion externen Signalen an. Rückblickend sind zwei Herangehensweisen bei der Entdeckung der S/MARs von Bedeutung:
- die Beschreibung der SARs (scaffold-attachment elements) von Laemmli &Co., von denen angenommen wurde, dass sie eine gegebene Chromatindomäne abgrenzen[6]
- die Charakterisierung der MARs (matrix-associated regions) die ersten Beispiele die die Immunoglobulin-kappa-chain-Enhancer je nach dessen Besetzung mit Transkriptionsfaktoren unterstützen[7]
Nachfolgende Arbeiten haben demonstriert, dass beide Funktionen der Elemente – die constitutive (SAR-ähnlich) und die fakultative (MAR-ähnlich) – abhängig vom Zusammenhang sind. Wohingegen konstitutive S/MARs in allen Zelltypen mit DNAse I hypersensitive sites[8] verbunden gefunden wurden (unabhängig davon ob die anliegende Domäne transkribiert wurde oder nicht), hängt die DNAse I Hypersensivität des fakultativen Typs vom Transkriptionsstatus ab.[9] Der auffälligste Unterschied zwischen diesen beiden funktionellen Arten der S/MARs ist ihre Größe: die konstitutiven Elemente dürften sich über mehrere kbp erstrecken, wohingegen die fakultativen die kleineren sind, ca. um die 300 bp.
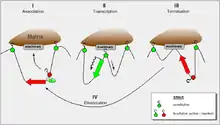
Die Abbildung zeigt das derzeitige Verständnis von diesen Eigenschaften und enthält folgende Erkenntnisse:
- die dynamischen Eigenschaften der S/MAR-scaffold Kontakte, abgeleitet von der Forschung mittels haloFISH (halo-Fluoreszenz-in-situ-Hybridisierung)[10]
- die Tatsache, dass während der Transkription DNA durch RNA-Polymerase gespult wird, die seinerseits eine feste Komponente der Kernmatrix ist[11]
- die Tatsache, dass bestimmte Domän-intrinsische S/MARs die Unterstützung eines anliegenden Transkriptionsfaktors erfordern, um aktiv zu werden[9]
Weiterführend
Tetko stellte in Arabidopsis thaliana eine Beziehung zwischen intragenischen S/MARs und spatiotemporaler Genexpression (Ort und Zeitpunkt spezifische Expression bezogen auf Gewebsregionen innerhalb eines Organismus während einer Entwicklung) fest.[12] Auf einer Genkarte, die gewebs- und organspezifische Genexpression sowie die der Entwicklung betont, sind S/MARs beinhaltende Muster gefunden wurden. Vor allem beinhalten die Gene der Transkriptionsfaktoren eine signifikant höhere Menge an S/MARs. Der auffallende Unterschied der Expressionscharakteristika der S/MARs beinhaltenden Gene unterstreicht die Wichtigkeit ihre funktionellen Bedeutung und die der struktur-chromosomalen Charakteristika für die Genregulation sowohl in Pflanzen als auch in anderen Eukaryoten.
Einzelnachweise
- J. M. Weitzel, H. Buhrmester, W. H. Strätling: Chicken MAR-binding protein ARBP is homologous to rat methyl-CpG-binding protein MeCP2. In: Molecular and cellular biology. Band 17, Nummer 9, September 1997, S. 5656–5666, PMID 9271441, PMC 232414 (freier Volltext).
- HMGB2 in der englischsprachigen Wikipedia
- David P. Clark, Nanette J. Pazdernik: Molekulare Biotechnologie – Grundlagen und Anwendungen. Spektrum Akad. Verlag, 2009, ISBN 978-3-8274-2128-9, S. 7.
- SIDD in der englischsprachigen Wikipedia
- SMARtDB
- J. Mirkovitch, M. E. Mirault, U. K. Laemmli: Organization of the higher-order chromatin loop: specific DNA attachment sites on nuclear scaffold. In: Cell. Band 39, Nr. 1, November 1984, S. 223–332, doi:10.1016/0092-8674(84)90208-3, PMID 6091913.
- P. N. Cockerill, W. T. Garrard: Chromosomal loop anchorage of the kappa immunoglobulin gene occurs next to the enhancer in a region containing topoisomerase II sites. In: Cell. Band 44, Nr. 2, Januar 1986, S. 273–82, doi:10.1016/0092-8674(86)90761-0, PMID 3002631.
- DNAse I hypersensitive site in der englischsprachigen Wikipedia
- M. Klar, E. Stellamanns, P. Ak, A. Gluch, J. Bode: Dominant genomic structures: detection and potential signal functions in the interferon-beta domain. In: Gene. Band 364, Dezember 2005, S. 79–89, doi:10.1016/j.gene.2005.07.023, PMID 16185826.
- H. H. Heng, S. Goetze, C. J. Ye u. a.: Chromatin loops are selectively anchored using scaffold/matrix-attachment regions. In: J. Cell. Sci. Band 117, Pt 7, März 2004, S. 999–1008, doi:10.1242/jcs.00976, PMID 14996931.
- D. A. Jackson, A. Dolle, G. Robertson, P. R. Cook: The attachments of chromatin loops to the nucleoskeleton. In: Cell Biol. Int. Rep. Band 16, Nr. 8, August 1992, S. 687–96, doi:10.1016/s0309-1651(05)80013-x, PMID 1446346.
- Igor V. Tetko, Georg Haberer, Stephen Rudd, Blake Meyers, Hans-Werner Mewes, Klaus F. X. Mayer: Spatiotemporal Expression Control Correlates with Intragenic Scaffold Matrix Attachment Regions (S/MARs) in Arabidopsis thaliana. In: PLoS Computational Biology. 2 (2006), S. 136–145. plosjournals.org (Memento des Originals vom 10. Juli 2012 im Webarchiv archive.today) Info: Der Archivlink wurde automatisch eingesetzt und noch nicht geprüft. Bitte prüfe Original- und Archivlink gemäß Anleitung und entferne dann diesen Hinweis., doi:10.1371/journal.pcbi.0020021.