F430
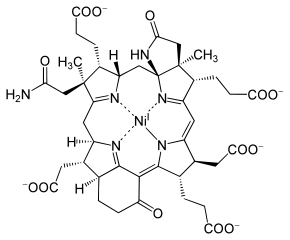
Der Kofaktor F430, auch F430, ist die prosthetische Gruppe des Enzyms Methyl-Coenzym-M-Reduktase. Seinem Absorptionsmaximum bei λmax = 430 nm verdankt er den Namen. Der Kofaktor kommt ausschließlich in methanogenen Archaeen vor.
| Strukturformel | |||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
 | |||||||||||||
| Allgemeines | |||||||||||||
| Name | F430 | ||||||||||||
| Andere Namen |
| ||||||||||||
| Summenformel | C42H51N6NiO13− | ||||||||||||
| Externe Identifikatoren/Datenbanken | |||||||||||||
| |||||||||||||
| Eigenschaften | |||||||||||||
| Molare Masse | 906,6 g·mol−1 | ||||||||||||
| Sicherheitshinweise | |||||||||||||
| |||||||||||||
| Soweit möglich und gebräuchlich, werden SI-Einheiten verwendet. Wenn nicht anders vermerkt, gelten die angegebenen Daten bei Standardbedingungen. | |||||||||||||
Entdeckung
F430 wurde 1977 von Jean LeGall als gelbe Komponente in Zellextrakten von Methanobacterium thermoautotrophicum entdeckt. Wegen seiner starken Absorption bei λmax = 430 nm wurde er Faktor F430 bezeichnet.[2] Die 1980 von Thauer und Mitarbeitern vorgeschlagene Tetrapyrrol-ähnliche Struktur wurde schließlich durch NMR-Studien verifiziert.[3]
Vorkommen
Obwohl der Kofaktor F430 in seinem Aufbau anderen Tetrapyrrolen ähnelt, wurde er nur in methanogenen Archaeen nachgewiesen.[4] In jenen dient er ausschließlich als prosthetische Gruppe des Enzyms Methyl-Coenzym-M-Reduktase (MCR).
Chemische Eigenschaften
F430 erinnert seiner Struktur nach einem Tetrapyrrolring und ähnelt den Porphyrinen bzw. Corrinen. Der Chromophor ist ein Tetrahydro-Derivat des sogenannten Corphins. Das Ringsystem in F430 hat insgesamt nur fünf Doppelbindungen und ist damit der am stärksten reduzierte Tetrapyrrolring in der Natur. Durch den Mangel an konjugierten Doppelbindungen ist es im Gegensatz zum ungesättigteren Tetrapyrrolen (beispielsweise dem roten Häm) gelb. Darüber hinaus ist das Ringsystem durch zwei angeknüpfte Ringe vergrößert.
Bislang ist es der einzige Abkömmling eines Tetrapyrrolsystems, das ein Nickelion enthält. Dieses liegt als Ni(I) vor und ist paramagnetisch. Der Kofaktor kann durch Denaturieren der MCR mittels Säuren isoliert werden. Isoliertes F430 ist thermisch instabil und Sauerstoff-sensitiv.[5]

Es wurde eine Variante des Cofaktors F430 entdeckt, die am C172-Atom durch eine Methylthio-Gruppe modifiziert ist (siehe Bild).[6] Diese Variante hat aber anscheinend keine Auswirkung auf das Nickelatom im Zentrum, das für die Funktion des Cofaktors essentiell ist. Warum diese Variante so modifiziert ist, steht noch zur Diskussion. Weitere Varianten und Modifikationen hat man in anderen methanogenen und anaeroben methanotrophen Archaeen nachgewiesen.[7]
Bedeutung
Der Kofaktor ist die prosthetische Gruppe des Enzyms Methyl-Coenzym-M-Reduktase (MCR). Jedes Enzym enthält zwei nicht kovalent gebundene F430. MCR katalysiert den letzten Schritt der Methanogenese, bei der Methan freigesetzt wird und ein Disulfidkomplex aus Coenzym M (CoM) und Coenzym B (CoB) entsteht:
Der genaue Mechanismus ist noch nicht aufgeklärt. Es ist dabei auch unklar, ob infolge der Katalyse Ni(III) gebildet werden kann.[8][9]
Biosynthese
Die Biosynthese des Kofaktors beginnt wie bei allen anderen natürlichen Tetrapyrrolen beim gemeinsamen Vorläufermolekül Uroporphyrinogen III.[10] Dieses wird zunächst zu Sirohydrochlorin umgesetzt.[11] In Sirohydrochlorin wird anschließend mittels einer Typ 2 Chelatase (CfbA) das Nickelatom eingebaut, so dass daraus Ni(II)-Sirohydrochlorin entsteht. Eine Amidase (CfbE) setzt zwei der Acetat-Seitenketten unter ATP- und N-Verbrauch zu Acetamid um, was infolgedessen zur Bildung von Ni(II)-Sirohydrochlorin-a,c-diamid führt. Dieses wird durch einen Enzymkomplex CfbC/CfbD zu einem Intermediat reduziert: Ni(I)-Hexahydrosirohydrochlorin-a,c-diamid. Bei diesem Prozess werden insgesamt sechs Elektronen und sieben Protonen übertragen. Ob die folgende Ringschließung zu seco-F430 spontan oder enzymatisch erfolgt, wird noch untersucht. Aus seco-F430 entsteht schließlich unter ATP-Verbrauch F430, indem sich der carbozyklische Ring F (mit der Ketogruppe) unter Wasserabspaltung bildet. Dies katalysiert eine Ligase (CfbB).[12]
Die für die Biosynthese nötige N-Quelle stellen entweder Glutamin oder freies Ammonium dar.
Siehe auch
- Methanogenese
- Coenzym M
- Coenzym B
Literatur
- A. A. DiMarco et al.: Unusual coenzymes of methanogenesis. In: Annu Rev Biochem., 59, 1990, S. 355–394, PMID 2115763.
- R. P. Hausinger: Nickel utilization by microorganisms. In: Microbiol Rev., 51, (1) 1987, S. 22–42 (englisch); PMID 3104749, mmbr.asm.org
Einzelnachweise
- Dieser Stoff wurde in Bezug auf seine Gefährlichkeit entweder noch nicht eingestuft oder eine verlässliche und zitierfähige Quelle hierzu wurde noch nicht gefunden.
- R. P. Gunsalus, R. S. Wolfe: Chromophoric factors F342 and F430 of Methanobacterium thermoautotrophicum. In: FEMS Microbiol. Lett. 1978, 3 (4), S. 191–193, doi:10.1111/j.1574-6968.1978.tb01916.x
- D. A. Livingston et al.: Zur Kenntnis des Faktors F430 aus methanogenen Bakterien: Struktur des proteinfreien Faktors. In: Helv. Chim. Acta. 1984, 67 (1), S. 334–351, doi:10.1002/hlca.19840670141
- G. Diekert et al.: Nickel requirement and factor F430 content of methanogenic bacteria. In: J Bacteriol. 1981, 148 (2), S. 459–464, PMID 7298577, PDF (freier Volltextzugriff, engl.)
- A. Ghosh et al.: Deconstructing F430: quantum chemical perspectives of biological methanogenesis. In: Curr Opin Chem Biol. 2001, 5 (6), S. 744–750, PMID 11738187.
- S. Mayr et al.: Structure of an F430 variant from archaea associated with anaerobic oxidation of methane. In: J. Am. Chem. Soc. 2008, 130 (32), S. 10758–10767, PMID 18642902, doi:10.1021/ja802929z.
- KD. Allen et al.: Discovery of multiple modified F(430) coenzymes in methanogens and anaerobic methanotrophic archaea suggests possible new roles for F(430) in nature. In: Appl Environ Microbiol., 2014, 80(20), S. 6403–6412. doi:10.1128/AEM.02202-14; PMID 25107965
- U. Ermler: On the mechanism of methyl-coenzyme M reductase. In: J. Chem. Soc., Dalton Trans. 2005, 21, S. 345–348, PMID 16234924, PDF (freier Volltextzugriff, engl.)
- S. W. Ragsdale: Nickel and the carbon cycle. In: J. Inorg. Biochem. 2007, 101 (11–12), S. 1657–1666, PMID 17716738, PMC 2100024 (freier Volltext).
- Simon J. Moore, Sven T. Sowa, Christopher Schuchardt, Evelyne Deery, Andrew D. Lawrence, José Vazquez Ramos, Susan Billig, Claudia Birkemeyer, Peter T. Chivers: Elucidation of the biosynthesis of the methane catalyst coenzyme F430. In: Nature. 543, Nr. 7643, 2. März 2017, ISSN 0028-0836, S. 78–82. doi:10.1038/nature21427.
- Helmut Mucha, Eberhard Keller, Hans Weber, Franz Lingens, Walter Trösch: Sirohydrochlorin, a precursor of factor F430 biosynthesis in Methanobacterium thermoautotrophicum. In: FEBS Letters. 190, Nr. 1, 7. Oktober 1985, S. 169–171. doi:10.1016/0014-5793(85)80451-8.
- Zheng K, Ngo PD, Owens VL, Yang XP, Mansoorabadi SO: The biosynthetic pathway of coenzyme F430 in methanogenic and methanotrophic archaea. In: Science. 354, Nr. 6310, Oktober 2016, S. 339–342. doi:10.1126/science.aag2947. PMID 27846569.