Chrysochromulina-ericina-Virus
Chrysochromulina-ericina-Virus 01B oder einfach Chrysochromulina-ericina-Virus (CeV-01B, CeV, alias Haptolina-ericina-Virus, HeV)[3][4] ist ein Riesenvirus aus der erweiterten Familie Mimiviridae (Ordnung Imitervirales), das Haptolina ericina (früher der Gattung Chrysochromulina zugeordnet[5]) infiziert, eine marine Mikroalge aus der Gruppe der Haptophyta. CeV ist wie alle Mitglieder der erweiterten Mimiviridae (bzw. „Imitervirales“) ein dsDNA-Virus[6] und gehört mit diesen zum Phylum Nucleocytoviricota (veraltet Nucleocytoplasmic large DNA viruses, NCLDV).
| Chrysochromulina-ericina-Virus | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
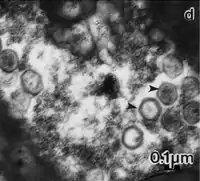
TME-Aufnahme von Dünnschnitten einer mit | ||||||||||||||||||
| Systematik | ||||||||||||||||||
| ||||||||||||||||||
| Taxonomische Merkmale | ||||||||||||||||||
| ||||||||||||||||||
| Wissenschaftlicher Name | ||||||||||||||||||
| „Chrysochromulina ericina virus 01B“ | ||||||||||||||||||
| Kurzbezeichnung | ||||||||||||||||||
| CeV-01B | ||||||||||||||||||
| Links | ||||||||||||||||||
|
Geschichte und Systematik
CeV wurde 1998 in den norwegischen Küstengewässern entdeckt, isoliert und charakterisiert.[6] Es wurde damals angenommen, dass es zusammen mit allen anderen bekannten Viren, die Algen infizieren – der Gruppe der „OLPG“ (englisch Organic Lake Phycodna(virus) Group) – zu den Phycodnaviridae gehört.[6][7] Die Entdeckung von Acanthamoeba polyphaga Mimivirus zeigte, dass es marine Mimiviren gibt, die Mikroalgen infizieren könnten.[8] Ein CeV-Stamm wurde später 2013 im Golf von Maine gefunden,[9] und die phylogenetische Analyse einiger spezifischer Marker bestätigte die Nähe zu den Mimiviren.[7][9] Im Jahr 2015 wurde CeV vollständig sequenziert, um es als Mitglied der erweiterten Mimiviridae zu klassifizieren.[10]
Inzwischen wurde vorgeschlagen, die Familie Mimiviridae um einige früher zu den Phycodnaviridae gestellte Vertreter die Mikroalgen parasitieren zu erweitern. Für die so erweiterten Mimiviridae wurde vom ICTV im März 2020 mit der neu geschaffenen Ordnung Imitervirales eine Heimat gefunden. Für die Erweiterung selbst (zumindest die Klade mit „OLPG“) wurde der Rang einer Familie „Mesomimiviridae“ vorgeschlagen[2] — innerhalb einer erweiterten Familie Mimiviridae war für diese Gruppe früher bereits der Rang einer Unterfamilie „Mesomimiviridae“ vorgeschlagen worden. Zu diesen gehören außer CeV auch die Kandidaten[11][12][13]
- „Organic Lake Phycodnavirus 1“ und „2“ (OLPV1, OLPV2),[14]
- „Phaeocystis globosa Virus 12“, „14“, „16“ (PgV-12T, PgV-14T, PgV-16T, infizieren Haptophyta),[12][15][16]
- „Phaeocystis pouchetii virus 01“ (PpV),[17]
- „Yellowstone Lake Mimivirus“[18] alias „Yellowstone lake giant virus“ (YLGV) bzw. ursprünglich „Yellowstone Lake Phycodnavirus 4“ (YSLPV4)
- „Prymnesium kappa virus RF01“ und „RF02“ (PkV-RF01, PkV-RF02)[19][20][4]
als „OLPG“-Mitglieder; sowie eventuell
- „Pyramimonas orientalis virus 01“ (PoV)[21][22]
- „Tetraselmis virus“ (TetV-1),[23][21][22] und
- „Aureococcus anophagefferens virus“ (AaV, englisch brown tide virus, infiziert Stramenopile)[24][15]
sollten diese nicht basal in den erweiterten Mimivirdae (d. h. Imitervirales) stehen.
Damit wären die „Mesomimiviridae“ die Schwestergruppe zu den herkömmlichen Mimiviridae.[25][26][27]
Aufbau
Die Viruspartikel/Virionen von CeV haben einen Durchmesser von 160 nm. Sie haben eine ikosaedrische Struktur und keine äußere Membran.[6]
Genom

Das Genom von CeV (Isolat CeV-01B) hat 473.558 bp und einen niedrigen GC-Gehalt von 25 %. Es wird vorausgesagt, dass 512 ORFs (englisch Open Reading Frame) vorhanden sind (vorhergesagte Anzahl der kodierten Proteine).[10][28] CeV besitzt eine große Anzahl von Kerngenen wie das Major capsid protein VP1 und die DNA-Polymerasen B mit Ähnlichkeit zu den jeweiligen Genen von PgV.
Das Vorhandensein der Sequenzen von MutS7 und einer DNA-Reparatur-Nuklease vom Typ ERCC4, die an der DNA-Reparatur beteiligt sind, legt nahe, dass CeV die Fähigkeit besitzen könnte, seine DNA zu reparieren. Das zuletzt genannte Enzym wird typischerweise zur Reparatur von DNA-Schäden verwendet, die durch UV-Licht verursacht werden. Das entspricht dem Lebensraum eines Mimivirus, das einen photosynthetischen Wirt infiziert. CeV hat darüber hinaus 305 Gene, zu denen die keine Übereinstimmung in den öffentlichen Datenbanken gefunden werden konnte, und die daher möglicherweise spezifisch für dieses Virus sind.[11]
Vermehrungszyklus
Über den Vermehrungszyklus von CeV ist wenig bekannt. Es repliziert im Zytoplasma des Wirts und sein Lysezyklus dauert 14 bis 19 Stunden.[6] CeV besitzt in seinem Genom eine Sequenz, die für eine DNA-Polymerase und zwei DNA-abhängige RNA-Polymerase II kodiert. Es hat auch zwölf tRNAs, was auf eine bedeutende Maschinerie für eine relative unabhängige Replikation und Virionbildung hinweist, wie es für Mimiviridea charakteristisch ist.[11]
Einzelnachweise
- ICTV: ICTV Master Species List 2019.v1, New MSL including all taxa updates since the 2018b release, March 2020 (MSL #35)
- Jonathan Filée: Giant viruses and their mobile genetic elements: the molecular symbiosis hypothesis, in: Current Opinion in Virology, Band 33, Dezember 2018, S. 81–88; bioRxiv: 2018/04/11/299784 (Preprint-Volltext)
-
Romain Blanc-Mathieu, Håkon Dahle, Antje Hofgaard, David Brandt, Hiroki Ban, Jörn Kalinowski, Hiroyuki Ogata, Ruth-Anne Sandaa: A Persistent Giant Algal Virus, with a Unique Morphology, Encodes an Unprecedented Number of Genes Involved in Energy Metabolism, in: ASM Journals: Journal of Virology, Band 95, Nr. 8, 25. März 2021, doi:10.1128/JVI.02446-20 .
PrePrint: bioRxiv, doi:10.1101/2020.07.30.228163, CSH, 13. Januar 2021 - Torill Vik Johannessen, Gunnar Bratbak, Aud Larsen, Hiroyuki Ogatac, Elianne S. Egged, Bente Edvardsen, Wenche Eikremd, Ruth-Anne Sandaa: Characterisation of three novel giant viruses reveals huge diversity among viruses infecting Prymnesiales (Haptophyta), in: Virology, Band 476, Februar 2015, S. 180-188, doi:10.1016/j.virol.2014.12.014, PMID 25546253, insbes. Fig. 4 und Fig. S2
- Bente Edvardsen, Wenche Eikrem, Jahn Throndsen, Alberto G. Sáez, Ian Probert, Linda K. Medlin: Ribosomal DNA phylogenies and a morphological revision provide the basis for a revised taxonomy of the Prymnesiales (Haptophyta). In: European Journal of Phycology. 46, Nr. 3, August 2011, S. 202–228. doi:10.1080/09670262.2011.594095.
- Ruth-Anne Sandaa, Mikal Heldal, Tonje Castberg, Runar Thyrhaug, Gunnar Bratbak: Isolation and Characterization of Two Viruses with Large Genome Size Infecting Chrysochromulina ericina (Prymnesiophyceae) and Pyramimonas orientalis (Prasinophyceae). In: Virology. 290, Nr. 2, November 2001, S. 272–280. doi:10.1006/viro.2001.1161. PMID 11883191.
- J. B. Larsen, A. Larsen, G. Bratbak, R.-A. Sandaa: Phylogenetic Analysis of Members of the Phycodnaviridae Virus Family, Using Amplified Fragments of the Major Capsid Protein Gene. In: Applied and Environmental Microbiology. 74, Nr. 10, 21. März 2008, S. 3048–3057. doi:10.1128/AEM.02548-07. PMID 18359826. PMC 2394928 (freier Volltext).
- Adam Monier, Jens B. Larsen, Ruth-Anne Sandaa, Gunnar Bratbak, Jean-Michel Claverie, Hiroyuki Ogata: Marine mimivirus relatives are probably large algal viruses. In: Virology Journal. 5, Nr. 1, 2008, S. 12. doi:10.1186/1743-422X-5-12. PMID 18215256. PMC 2245910 (freier Volltext).
- William H. Wilson, Ilana C. Gilg, Amy Duarte, Hiroyuki Ogata: Development of DNA mismatch repair gene, MutS, as a diagnostic marker for detection and phylogenetic analysis of algal Megaviruses. In: Virology. 466-467, Oktober 2014, S. 123–128. doi:10.1016/j.virol.2014.07.001. PMID 25063474.
- Lucie Gallot-Lavallée, António Pagarete, Matthieu Legendre, Sebastien Santini, Ruth-Anne Sandaa, Heinz Himmelbauer, Hiroyuki Ogata, Gunnar Bratbak, Jean-Michel Claverie: The 474-Kilobase-Pair Complete Genome Sequence of CeV-01B, a Virus Infecting Haptolina (Chrysochromulina) ericina (Prymnesiophyceae). In: Genome Announcements. 3, Nr. 6, 3. Dezember 2015. doi:10.1128/genomeA.01413-15. PMID 26634761. PMC 4669402 (freier Volltext).
- Lucie Gallot-Lavallée, Guillaume Blanc, Jean-Michel Claverie, Grant McFadden: Comparative Genomics of Chrysochromulina ericina virus and Other Microalga-Infecting Large DNA Viruses Highlights Their Intricate Evolutionary Relationship with the Established Mimiviridae Family. In: Journal of Virology. 91, Nr. 14, 15. Juli 2017. doi:10.1128/JVI.00230-17. PMID 28446675. PMC 5487555 (freier Volltext).
- Jean-Michel Claverie, Chantal Abergel: Mimiviridae: An Expanding Family of Highly Diverse Large dsDNA Viruses Infecting a Wide Phylogenetic Range of Aquatic Eukaryotes. In: Viruses. 2018 Sep; 10(9), 18. September 2018, S. 506, doi:10.3390/v10090506, PMC 6163669 (freier Volltext), PMID 30231528
- William H Wilson, Ilana C Gilg, Mohammad Moniruzzaman, Erin K Field, Sergey Koren, Gary R LeCleir, Joaquín Martínez Martínez, Nicole J Poulton, Brandon K Swan, Ramunas Stepanauskas, Steven W Wilhelm: Genomic exploration of individual giant ocean viruses, in: ISME Journal 11(8), August 2017, S. 1736–1745, doi:10.1038/ismej.2017.61, PMC 5520044 (freier Volltext), PMID 28498373
- Weijia Zhang, Jinglie Zhou, Taigang Liu, Yongxin Yu, Yingjie Pan, Shuling Yan, Yongjie Wang et al.: Four novel algal virus genomes discovered from Yellowstone Lake metagenomes. In: Scientific Reports. 5, Nr. 1, 13. Oktober 2015, S. 15131. doi:10.1038/srep15131. PMC 4602308 (freier Volltext).
- Sailen Barik: A Family of Novel Cyclophilins, Conserved in the Mimivirus Genus of the Giant DNA Viruses, in: Computational and Structural Biotechnology Journal, Band 16, Juli 2018, S. 231–236, doi:10.1016/j.csbj.2018.07.001
- Jônatas Abrahão, Lorena Silva, Ludmila Santos Silva, Jacques Yaacoub Bou Khalil, Rodrigo Rodrigues, Thalita Arantes, Felipe Assis, Paulo Boratto, Miguel Andrade, Erna Geessien Kroon, Bergmann Ribeiro, Ivan Bergier, Herve Seligmann, Eric Ghigo, Philippe Colson, Anthony Levasseur, Guido Kroemer, Didier Raoult, Bernard La Scola: Tailed giant Tupanvirus possesses the most complete translational apparatus of the known virosphere. In: Nature Communications. 9, Nr. 1, 27. Februar 2018. doi:10.1038/s41467-018-03168-1.
- Natalya Yutin, Philippe Colson, Didier Raoult, Eugene V Koonin: Mimiviridae: clusters of orthologous genes, reconstruction of gene repertoire evolution and proposed expansion of the giant virus family, in: Virol J. 2013; 10: 106, 4. April 2013, doi:10.1186/1743-422X-10-106, PMC 3620924 (freier Volltext), PMID 23557328
- NCBI: Yellowstone lake mimivirus (species)
- NCBI: Prymnesium kappa virus (species)
- Lucie Gallot-Lavallee, Guillaume Blanc, Jean-Michel Claverie: Comparative genomics of Chrysochromulina Ericina Virus (CeV) and other microalgae-infecting large DNA viruses highlight their intricate evolutionary relationship with the established Mimiviridae family, in: J. Virol., 26 April 2017, doi:10.1128/JVI.00230-17
- Christopher R. Schvarcz, Grieg F. Steward: A giant virus infecting green algae encodes key fermentation genes. Virology, 2018; 518: 423 doi:10.1016/j.virol.2018.03.010
- Christoph M. Deeg, Cheryl-Emiliane T. Chow, Curtis A. Suttle: The kinetoplastid-infecting Bodo saltans virus (BsV), a window into the most abundant giant viruses in the sea…, in: eLife Sciences 7, März 2018, doi:10.7554/eLife.33014
- A new giant virus found in the waters of Oahu, Hawaii, ScienceDaily, 3. Mai 2018
- David M. Needham, Susumu Yoshizawa, Toshiaki Hosaka, Camille Poirier, Chang Jae Choi, Elisabeth Hehenberger, Nicholas A. T. Irwin, Susanne Wilken, Cheuk-Man Yung, Charles Bachy, Rika Kurihara, Yu Nakajima, Keiichi Kojima, Tomomi Kimura-Someya, Guy Leonard, Rex R. Malmstrom, Daniel R. Mende, Daniel K. Olson, Yuki Sudo, Sebastian Sudek, Thomas A. Richards, Edward F. DeLong, Patrick J. Keeling, Alyson E. Santoro, Mikako Shirouzu, Wataru Iwasaki, Alexandra Z. Worden: A distinct lineage of giant viruses brings a rhodopsin photosystem to unicellular marine predators, in: PNAS, 23. September 2019, doi:10.1073/pnas.1907517116, ISSN 0027-8424
- Natalya Yutin et al.: Origin of giant viruses from smaller DNA viruses not from a fourth domain of cellular life. In: Virology, Volumes 466–467, Oktober 2014, S. 38–52, doi:10.1016/j.virol.2014.06.032
- Carolina Reyes, Kenneth Stedman: Are Phaeocystis globosa viruses (OLPG) and Organic Lake phycodnavirus a part of the Phycodnaviridae or Mimiviridae?, Blog auf ResearchGate, 8. Januar 2016
- Frederik Schulz, Lauren Alteio, Danielle Goudeau, Elizabeth M. Ryan, Feiqiao B. Yu, Rex R. Malmstrom, Jeffrey Blanchard, Tanja Woyke: Hidden diversity of soil giant viruses, in: Nature Communicationsvolume 9, Article number: 4881 (2018) vom 19. November 2018, doi:10.1038/s41467-018-07335-2
- David M. Needham, Alexandra Z. Worden et al.: A distinct lineage of giant viruses brings a rhodopsin photosystem to unicellular marine predators, in: PNAS, 23. September 2019, doi:10.1073/pnas.1907517116, ISSN 0027-8424, hier: Supplement 1 (xlsx)