Haptolina
Haptolina ist eine Gattung von Haptophyten, die zur Familie der Prymnesiaceae gehört.[1] Die Gattung wurde von Chrysochromulina ausgegliedert.[2]
| Haptolina | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|
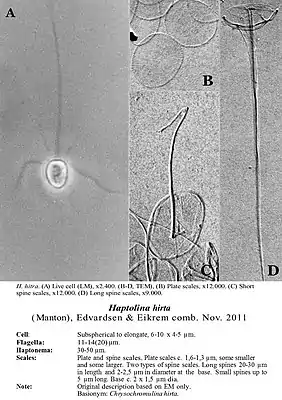
H. hirta von der Ostsee, Schuppen und Dornen | ||||||||||||
| Systematik | ||||||||||||
| ||||||||||||
| Wissenschaftlicher Name | ||||||||||||
| Haptolina | ||||||||||||
| Edvardsen & Eikrem, 2011 |
Die Gattung hat eine kosmopolitische Verbreitung.[1] Einige Arten können Algenblüten (mit-)
Arten
Die Systematik der Gattung Haptolina ist wie folgt:[4][1][5][6]
Gattung Haptolina Edvardsen & Eikrem, 2011
- Haptolina brevifila (Parke & Manton) Edvardsen & Eikrem, 2011[7] – Typus; früher Chrysochromulina brevifilum
- Haptolina ericina (Parke & Manton) Edvardsen & Eikrem, 2011[8] – früher C. ericina
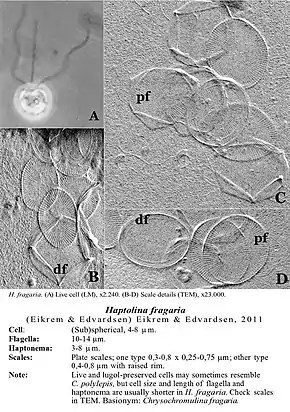
- Haptolina fragaria (Eikrem & Edvardsen) Eikrem & Edvardsen, 2011[9] – früher C. fragaria
- Haptolina herdlensis (B.Leadbeater) Edvardsen & Eikrem, 2011[10] – früher C. herdlensis
- Haptolina hirta (Manton) Edvardsen & Eikrem, 2011[11] – früher C. hirta
- „Haptolina sp. RCC2300“ – früher „Chrysochromulina sp. RCC2300“ (NCBI)[12]
Algenblüten
Kattegat-Algenblüte 1992: Von April bis Mai 1992 gab es im südlichen Kattegat eine große Blüte, die aus vielen Phytoplanktonarten bestand, wobei über 90 % der Biomasse Chrysochromulina-Arten waren (viele aber inzwischen der Gattung Haptolina zugeordnet).[3] Die am häufigsten vorkommenden Arten in der Blüte waren:[3]
- Haptolina hirta (früher Chrysochromulina hirta),
- H. ericina (früher C. ericina),
- H. brevifila (früher C. brevifilum),
- Chrysochromulina spinifera, sowie
- eine unbeschriebene Art (Ø. Moestrup).
H. hirta, H. ericina und C. spinifera zeichnen sich durch relativ kleine Zellen mit langen Dornen aus, die den Gesamtorganismen einen Durchmesser von 25–76 μm verleihen, was für die vorhandenen Ciliaten (als potentielle Fressfeinde) zu groß ist, um sie zu verschlingen; wahrscheinlich ein Grund für den „Erfolg“ der Blüte war.[3]
Es gab zwar keine Beweise, die diese Blüte oder die vorhandenen Arten direkt mit der Produktion von Toxinen (wie bei der C. polylepis-Blüte 1988) in Verbindung brachten.[13][3] Es wurden jedoch etwa 50 Tonnen toter Regenbogenforellen (Oncorhynchus mykiss) in Verbindung mit der Blüte gebracht.[3]
Viren
Es wurde über mehrere Virenspezies-Kandidaten berichtet, insbesondere über:
- „Chrysochromulina-ericina-Virus 01B“ (CeV-01B)[14] alias „Haptolina-ericina-Virus 01B“ (HeV-01B), Klasse Megaviricetes
- „Haptolina ericina virus RF02B“ (HeV-RF02)
Derzeit (Stand Juli 2021) ist allerdings noch kein Haptolina-Virus vom International Committee on Taxonomy of Viruses (ICTV) offiziell bestätigt.
CeV-01B/HeV-01B
CeV-01B (HeV-01B) wurde erstmals 1998 aus Küstengewässern Norwegens isoliert. Es handelt sich um ein ikosaedrisches DNA-Doppelstrangvirus mit einer Genomgröße von 474 kbp (Kilobasenpaaren). CeV-01B/HeV-01B gehört zur Subklade „OLPG“ der erweiterten Familie Mimiviridae (auch Megaviridae) in der Ordnung Imitervirales.[14]
HeV-RF02
HeV-RF02 wurde isoliert auf dem Stamm Haptolina ericina UIO028 aus dem Raunefjord (Provinz Vestland, Norwegen). Torill Vik Johannessen und Kollegen berichteten 2015 über die Entdeckung dreier neuer lytischer Doppelstrang-DNA-Viren aus dem Raunefjord. Diese stehen mutmaßlich der Familie Mimiviridae nahe oder gehören zu ihr. Neben „Haptolina ericina virus RF02“ (HeV-RF02) waren die beiden anderen Kandidaten „Prymnesium kappa virus RF01“ (PkV-RF01)[15][16][17] und „Prymnesium kappa virus RF02“ (PkV-RF02). Jedes dieser neuen Haptophytenviren stellte zum Zeitpunkt der Entdeckung die gängigen Vorstellungen von Algenviren in Bezug auf Wirtsspektrum, phylogenetische Zugehörigkeit und Größe in Frage. HeV-RF02 war (mit einem Kapsiddurchmesser von ca. 310 nm) das bis dato größte bekannte Algenvirus. Die Ergebnisse zeigten eine große Variation unter Viren, die eng verwandte Mikroalgen infizieren, und stellen damit die gängige Vorstellung in Frage, dass Algenviren einen engen Wirtsbereich haben der die Phylogenie ihre Wirtszugehörigkeit widerspiegelt.[18] HeV-RF02 und PkV-RF02 stehen CeV-01B/HeV-01B, „Phaeocystis globosa virus 12“ (PgV-12T) und „Phaeocystis pouchetii virus 01“ (PpV01) nahe, alle in der Gruppe mit der provisorischen Bezeichnung „OLPG“. Es gibt derzeit (20. Juli 2021) noch keine offizielle Taxonomie seitens des ICTVs für diese Funde; von den meisten Autoren werden auch diese den erweiterten Mimiviridae (en. extended Mimiviridae), Ordnung Imitervirales zugerechnet.[18]
Einzelnachweise
- Haptolina Edvardsen & Eikrem, 2011 (en) In: www.gbif.org. Abgerufen am 25. April 2021.
- B. Edvardsen, W. Eikrem, J. Throndsen, A. G. Sáez, I. Probert, L. K. Medlin: Ribosomal DNA phylogenies and a morphological revision provide the basis for a revised taxonomy of the Prymnesiales (Haptophyta). European Journal of Phycology, Band 46, Nr. 3, 2011, S. 202–228
- Per Juel Hansen, Torkel Gissel Nielsen, Hanne Kaas: Distribution and Growth of Protists and Mesozooplankton during a Bloom of Chrysochromulina Spp. (Prymnesiophyceae, Prymnesiales). In: Phycologia. 34, Nr. 5, 1995, S. 409–416. doi:10.2216/i0031-8884-34-5-409.1. Epub 22. Apr 2019. Freier Volltext (ResearchGate)
- AlgaeBase: AlgaeBase: Haptolina Edvardsen & Eikrem, 2011
- NCBI: Haptolina Edvardsen & Eikrem (genus); graphisch: Haptolina, auf: Lifemap, NCBI Version.
- WoRMS: Haptolina Edvardsen & Eikrem, 2011
- Nordic Microalgae: Chrysochromulina brevifilum, auf: Nordic Microalgae and aquatic protozoa, Swedish Meterological and Hydrological Institute (SMHI)
- Nordic Microalgae: Chrysochromulina ericina, auf: Nordic Microalgae and aquatic protozoa, Swedish Meterological and Hydrological Institute (SMHI)
- Nordic Microalgae: Chrysochromulina fragaria, auf: Nordic Microalgae and aquatic protozoa, Swedish Meterological and Hydrological Institute (SMHI)
- Nordic Microalgae: Chrysochromulina herdlensis, auf: Nordic Microalgae and aquatic protozoa, Swedish Meterological and Hydrological Institute (SMHI)
- Nordic Microalgae: Chrysochromulina hirta, auf: Nordic Microalgae and aquatic protozoa, Swedish Meterological and Hydrological Institute (SMHI)
- Sergio Balzano, Dominique Marie, Priscillia Gourvil, Daniel Vaulot: Composition of the summer photosynthetic pico and nanoplankton communities in the Beaufort Sea assessed by T-RFLP and sequences of the 18S rRNA gene from flow cytometry sorted samples, in: ISME J, Band 6, Nr. 8, 2012, S. 1480–1498, PMID 22278671, PMC 3400408 (freier Volltext)
- Torkel Nielsen, Thomas Kiørboe, Peter Bjørnsen: Effects of a Chrysochromulina polylepis subsurface bloom on the planktonic community. In: Marine Ecology Progress Series. 62, Nr. 1/2, 1990, S. 21–35. bibcode:1990MEPS...62...21N. doi:10.3354/meps062021.
- Lucie Gallot-Lavallée, António Pagarete, Matthieu Legendre, Sebastien Santini, Ruth-Anne Sandaa, Heinz Himmelbauer, Hiroyuki Ogata, Gunnar Bratbak, Jean-Michel Claverie: The 474-Kilobase-Pair Complete Genome Sequence of CeV-01B, a Virus Infecting Haptolina (Chrysochromulina) ericina (Prymnesiophyceae). In: Genome Announcements. 3, Nr. 6, 3. Dezember 2015. doi:10.1128/genomeA.01413-15. PMID 26634761. PMC 4669402 (freier Volltext).
-
Romain Blanc-Mathieu, Håkon Dahle, Antje Hofgaard, David Brandt, Hiroki Ban, Jörn Kalinowski, Hiroyuki Ogata, Ruth-Anne Sandaa: A Persistent Giant Algal Virus, with a Unique Morphology, Encodes an Unprecedented Number of Genes Involved in Energy Metabolism, in: ASM Journals: Journal of Virology, Band 95, Nr. 8, 25. März 2021, doi:10.1128/JVI.02446-20 .
PrePrint: bioRxiv, doi:10.1101/2020.07.30.228163, CSH, 13. Januar 2021 - NCBI: Prymnesium kappa virus (species) – unclassified Phycodnaviridae
- Lucie Gallot-Lavallee, Guillaume Blanc, Jean-Michel Claverie: Comparative genomics of Chrysochromulina Ericina Virus (CeV) and other microalgae-infecting large DNA viruses highlight their intricate evolutionary relationship with the established Mimiviridae family (PDF) in: J. Virol., 26 April 2017, doi:10.1128/JVI.00230-17
- Torill Vik Johannessen, Gunnar Bratbak, Aud Larsen, Hiroyuki Ogatac, Elianne S. Egged, Bente Edvardsen, Wenche Eikremd, Ruth-Anne Sandaa: Characterisation of three novel giant viruses reveals huge diversity among viruses infecting Prymnesiales (Haptophyta), in: Virology, Band 476, Februar 2015, S. 180–188, doi:10.1016/j.virol.2014.12.014, PMID 25546253, insbes. Fig. 4 und Fig. S2