ADAM-Metalloproteasen
Die ADAM-Metalloproteasen, (engl. A Disintegrin And Metalloproteinase = „ein Disintegrin und Metalloproteinase“), sind eine Gruppe von Enzymen, die zu den Metzinkinen – einer Unterklasse der Metalloproteinasen – gehören. Als Kofaktor wird ein Zink-Ion benötigt. Bisher sind über 30 orthologe ADAMs bekannt. Dazu kommen noch Spleißvarianten.[1][2]
Aufbau
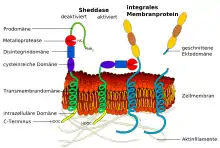
ADAM-Metalloproteasen sind integrale Transmembranproteine vom Typ I (single pass). Sie bestehen in der Regel aus 800 bis 1200 Aminosäuren. Der C-Terminus befindet sich im intrazellulären Raum. Ihm folgt der transmembrane Bereich, dem sich im extrazellulären Raum bei einigen ADAMs eine EGF-ähnliche Domäne anschließt. Der nachfolgenden cysteinreichen Domäne folgt die Disintegrin-Domäne. Danach kommt die Metalloprotease-Domäne. Eine Prodomäne mit N-Terminus schließt das ADAM-Protein ab. Die Prodomäne muss für die Aktivierung des Enzyms abgespalten werden.[3] Die Prodomäne deaktiviert die Metalloprotease-Domäne mittels eines Cystein-Schalters (cysteine switch).[4] Konservierte Cystein-Reste in der Prodomäne koordinieren zusätzlich das für die Funktion des Enzyms notwendige Zink-Ion (Zn2+) innerhalb der Metalloprotease-Domäne. Dadurch kommt die Metalloprotease-Domäne in eine inaktive Konformation, wodurch das gesamte Enzym deaktiviert ist. Durch Furin oder andere Proprotein-Convertasen kann die Prodomäne abgespalten werden, wodurch das Zink-Ion nur noch von der Metalloproteasen-Domäne koordiniert wird und so das Enzym aktiviert ist.[5]
Durch stärkere Komplexbildner, wie beispielsweise EDTA, wird das Zink gebunden und die ADAM-Protease (reversibel) deaktiviert.[3]
Die Disintegrin-Domäne trägt ihren Namen wegen der hohen Strukturanalogie zu den Disintegrinen in Schlangengiften (snake venom metalloprotease, SVMP).
Funktion
An einer Reihe von zellbiologischen Vorgängen sind die ADAM-Metalloproteasen unmittelbar beteiligt. So spielen sie beispielsweise bei der Membranfusion, der Entwicklung der Muskeln, der Zelldifferenzierung, der Zellmigration und bei Entzündungsreaktionen eine wichtige Rolle.[5][3] Beim Schneiden anderer Transmembranproteine, dem sogenannten Ectodomain-Shedding, sind die ADAMs ebenfalls beteiligt.[6] Beispielsweise wird das Amyloid-Precursor-Protein (APP) durch die ADAM-Metalloproteasen ADAM9, ADAM10 und ADAM17 – die zu den α-Sekretasen gehören – „richtig“ geschnitten.[7][8]
Von den bekannten humanen ADAM-Proteasen wird zwölf eine proteolytische Aktivität vorhergesagt. Bisher wurde nur in der Hälfte davon auch die proteolytische Aktivität gemessen. Die Selektivität beziehungsweise Spezifität der ADAM-Proteasen ist noch nicht gesichert.[5] Einige ADAMs gehören zu den Sheddasen.
Vorkommen
In allen Wirbeltieren finden sich die ADAM-Metalloproteasen. Auch Zellen der Modellorganismen Caenorhabditis elegans, Drosophila und Xenopus exprimieren ADAMs. Dagegen finden sie sich nicht auf den Zellmembranen von Escherichia coli und Saccharomyces cerevisiae. Auch bei Pflanzen sind sie nicht vorhanden. Die einzelnen ADAMs werden je nach Zelltyp unterschiedlich stark exprimiert.[5]
Einzelnachweise
- Familie M12 bei MEROPS
- EC 3.4.24.-
- F. Scholz: Einfluss der Disintegrin-ähnlichen Metalloproteinase ADAM10 auf die proteolytische Spaltung von transmembranen Proteinen der Haut. Dissertation, Christian-Albrechts-Universität zu Kiel, 2006. urn:nbn:de:gbv:8-diss-18259
- H. E. Van Wart und H. Birkedal-Hansen: The cysteine switch: A principle of regulation of metalloproteinase activity with potential applicability to the entire matrix metalloproteinase gene family. In: PNAS 87, 1990, S. 5578–5582. PMID 2164689
- D. F. Seals und S. A. Courtneidge: The ADAMs family of metalloproteases: multidomain proteins with multiple functions. In: Genes Dev 17, 2003, S. 7–30. PMID 12514095 (Review)
- C. P. Blobel: Functional and biochemical characterization of ADAMs and their predicted role in protein ectodomain shedding. In: Inflamm Res 51, 2002, S. 83–84. PMID 11926318 (Review)
- S. F. Lichtenthaler: Ectodomain shedding of the amyloid precursor protein: cellular control mechanisms and novel modifiers. In: Neurodegener Dis 3, 2006, S. 262–269. PMID 17047366 (Review)
- Gregor Larbig: Studien zur Identifizierung & Optimierung potentieller Wirkstoffe für die Behandlung von Morbus Alzheimer. Dissertation, TU Darmstadt, 2007
Literatur
Review-Artikel
- P. Primakoff und D. G. Myles: The ADAM gene family: surface proteins with adhesion and protease activity. In: Trends Genet 16, 2000, S. 83–87. PMID 10652535
- S. Higashiyama und D. Nanba: ADAM-mediated ectodomain shedding of HB-EGF in receptor cross-talk. In: Biochim Biophys Acta 1751, 2005, S. 110–117. PMID 16054021
- C. P. Blobel: ADAMs: key components in EGFR signalling and development. In: Nat Rev Mol Cell Biol 6, 2005, S. 32–43. PMID 15688065
- L. C. Bridges und R. D. Bowditch: ADAM-Integrin Interactions: potential integrin regulated ectodomain shedding activity. In: Curr Pharm Des 11, 2005, S. 837–847. PMID 15777238
- J. Schlöndorff und C. P. Blobel: Metalloprotease-disintegrins: modular proteins capable of promoting cell-cell interactions and triggering signals by protein-ectodomain shedding. In: J Cell Sci 112, 1999, S. 3603–3617. PMID 10523497
- D. R. Edwards u. a.: The ADAM metalloproteinases. In: Mol Aspects Med 29, 2008, S. 258–289. PMID 18762209
- T. G. Wolfsberg u. a.: ADAM, a novel family of membrane proteins containing A Disintegrin And Metalloprotease domain: multipotential functions in cell-cell and cell-matrix interactions. In: J Cell Biol 131, 1995, S. 275–278. PMID 7593158
Fachbücher
- N. M. Hooper und U. Lendeckel: The Adam Family of Proteases. Verlag Birkhäuser, 2005, ISBN 0-387-25149-9