Ectodomain-Shedding
Unter dem Vorgang des Ectodomain-Sheddings, das auch mit Abspaltung der Ektodomäne umschrieben wird, versteht man die proteolytische Abtrennung der extrazellulären Domänen (Ektodomänen) vieler Membranproteine.
Beschreibung
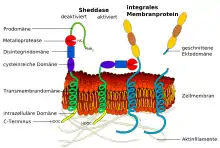
Beim Ectodomain-Shedding werden eine Reihe von Membranproteinen, wie beispielsweise Wachstumsfaktoren, Zytokine, Rezeptoren oder Zelladhäsionsmoleküle (CAMs), durch bestimmte Metalloproteasen[1] – diese Enzyme werden auch Sheddasen genannt – gespalten. Zu diesen Sheddasen gehören unter anderem die ADAMs (A Disintegrin And Metalloprotease) ADAM9, ADAM10 und ADAM17 aus der Metazincin-Familie.[2][3] Die Abspaltung der Membranproteine hat erhebliche Veränderungen an der Oberfläche der Zellen zur Folge, die beispielsweise auf die Kommunikation der Zelle oder aber auch auf therapeutische Maßnahmen großen Einfluss haben kann. Zudem entstehen beim Ectodomain-Shedding im extrazellulären Raum freie gelöste Regulatorproteine.[4][5]
Das Ectodomain-Shedding ist in einer Reihe von pathophysiologischen Vorgängen wie Entzündungen, Zelldegeneration, der Apoptose und bei der Entstehung von Krebs (Onkogenese) involviert.[6]
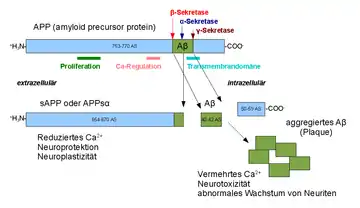
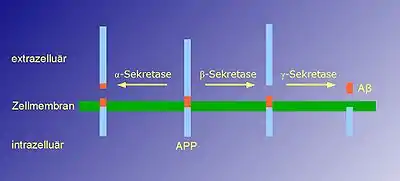
Auch im Fall der Alzheimer-Krankheit spielt das Ectodomain-Shedding eine wichtige Rolle. So schneiden die α-Sekretasen, beispielsweise ADAM 9, ADAM 10 und ADAM 17, das integrale Membranprotein APP (Amyloid-Precursor-Protein) an der „richtigen“ Stelle (nahe der Zellmembran), während die β-Sekretase (BACE1) das APP an der „falschen“ Stelle (weiter im extrazellulären Raum) schneidet. Die γ-Sekretase schneidet dann – unabhängig davon, ob zuvor die α- oder β-Sekretase das APP geschnitten hat – den APP-Rest innerhalb der Zellmembran. Der im Fall des ersten Schnittes entstehende größere Abschnitt (38, 40 oder 42 Aminosäuren), Aβ genannt, führt zu den Amyloidplaques, während das im Fall der α-Sekretase entstehende Fragment, P3 genannt, keine Plaques bildet.[7][8]
Das Antigen-Shedding ist eine spezielle Form des Ectodomain-Sheddings.[9]
Einzelnachweise
- J. Schlöndorff und C. P. Blobel: Metalloprotease-disintegrins: modular proteins capable of promoting cell-cell interactions and triggering signals by protein-ectodomain shedding. In: J Cell Sci 112, 1999, S. 3603–3617, PMID 10523497 (Review).
- L. C. Bridges und R. D. Bowditch: ADAM-Integrin Interactions: potential integrin regulated ectodomain shedding activity. In: Curr Pharm Des 11, 2005, S. 837–847. PMID 15777238 (Review)
- J. Mullberg u. a.: The importance of shedding of membrane proteins for cytokine biology. In: Eur Cytokine Netw 11, 2000, S. 27–38, PMID 10705296 (Review).
- C. P. Blobel: Remarkable roles of proteolysis on and beyond the cell surface. In: Curr Opin Cell Biol 12, 2000, S. 606–612. PMID 10978897 (Review)
- J. Sy: Die Disintegrin- und Metalloproteinasen-Familie „ADAM“ im humanen hämatopoetischen System. Dissertation, Eberhard-Karls-Universität zu Tübingen, 2002.
- P. Dello Sbarba und E. Rovida: Transmodulation of cell surface regulatory molecules via ectodomain shedding. In: Biol Chem 383, 2002, S. 69–83, PMID 11928824 (Review).
- S. F. Lichtenthaler: Ectodomain shedding of the amyloid precursor protein: cellular control mechanisms and novel modifiers. In: Neurodegener Dis 3, 2006, S. 262–269. PMID 17047366 (Review).
- Gregor Larbig: Studien zur Identifizierung & Optimierung potentieller Wirkstoffe für die Behandlung von Morbus Alzheimer. Dissertation, TU Darmstadt, 2007.
- Y. Zhang und I. Pastan: High Shed Antigen Levels within Tumors: An Additional Barrier to Immunoconjugate Therapy. In: Clin Cancer Res 14, 2008, S. 7981–7986, PMID 19088013 (Review).
Literatur
- C. P. Blobel: ADAMs: key components in EGFR signalling and development. In: Nat Rev Mol Cell Biol 6, 2005, S. 32–43. PMID 15688065
- J. Arribas und A. Borroto: Protein ectodomain shedding. In: Chemical Reviews 102, 2002, S. 4627–4638. PMID 12475204 (Review)
- S. Higashiyama und D. Nanba: ADAM-mediated ectodomain shedding of HB-EGF in receptor cross-talk. In: Biochim Biophys Acta 1751, 2005, S. 110–117. PMID 16054021 (Review)
- W. Xia und M. S. Wolfe: Intramembrane proteolysis by presenilin and presenilin-like proteases. In: J Cell Sci 116, 2003, S. 2839–2844. PMID 12808018 (Review)
- K. J. Garton u. a.: Emerging roles for ectodomain shedding in the regulation of inflammatory responses. In: J Leukoc Biol 79, 2006, S. 1105–1116. PMID 16565325 (Review)