pUC19
pUC19 gehört wie auch pUC18 zu einer Reihe im Labor von Joachim Messing gentechnologisch hergestellter Klonierungsvektoren.[1] Diese bakteriellen Plasmide gehören zu den am häufigsten verwendeten Vektoren zur Klonierung im Bakterium Escherichia coli. Damit haben sie in der biologischen Forschung und Gentechnik eine große Bedeutung. Ihr spezieller Vorteil sind die kleine Größe, die Möglichkeit rekombinante Plasmide mittels Blau-Weiß-Screening zu identifizieren und eine besonders hohe Anzahl von Kopien pro Bakterienzelle (sogenannte high copy number-Plasmide), so dass sehr einfach eine große Menge an Plasmid isoliert werden kann.
Eigenschaften
Entwicklungsgeschichtlich handelt es sich um Derivate des pBR322 Plasmids.[2] Die pUC-Plasmide benutzen den modifizierten origin of replication oder kurz ori des pBR322-Plasmids. Der ori wurde verkürzt und ihm fehlt die codierende Region für das ROM/ROP Protein. Durch knockout von ROP kann die Kopienzahl von ColEI Plasmiden generell erhöht werden, solange die Kultivierungstemperatur bei >30 °C liegt.[3] Zusätzlich weist der modifizierte ori eine Punktmutation G → A an Position 112 der RNA II auf. Hieraus resultiert die erhöhte Kopienzahl dieser Vektoren.[4]
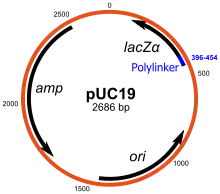
Sie sind mit 2686 bp relativ klein und ihnen können an speziellen Restriktionsenzym-Schnittstellen in der der Multiple Cloning Sites (MCS, Polylinker), zusätzliche DNA-Fragmente eingefügt werden. Zur Selektion von pUC-positiven Bakterien nach Einfügung des Plasmids in einen geeigneten Bakterienstamm (Transformation) dient ein in den pUC-Plasmiden vorhandenes Ampicillin-Resistenzgen (ampR). Die Transkription von eingefügten Genen in E. coli kann durch Induktion des eingebauten Promoterfragmentes des lac-Operons mit IPTG gezielt erreicht werden. Der Vektor codiert für das N-terminale Fragment (Aminosäuren 1-5 und 8-60) der β-Galactosidase (lacZ) von E. coli. Die Codons 6-7 wurden durch MCS ersetzt und erlauben so eine Blau-Weiß-Selektion. Die beiden pUC-Plasmide unterscheiden sich lediglich in der inversen Orientierung der MCS.
Es besitzt eine Masse von . In seinem natürlichen, superspiralisierten Zustand wurde sein gyroskopischer Radius mit 65,6 nm und sein hydrodynamischer Radius zu 43,6 nm bestimmt.[5]
Der Name der Plasmide leitet sich von der University of California (UC) ab, an der diese Plasmide (p) konstruiert wurden.[1]
Quellen
- C. Yanisch-Perron, J. Vieira, J. Messing: Improved M13 phage cloning vectors and host strains: nucleotide sequences of the M13mp18 and pUC19 vectors. In: Gene. Band 33, Nummer 1, 1985, S. 103–119. PMID 2985470.
- J. Vieira, J. Messing: The pUC plasmids, an M13mp7-derived system for insertion mutagenesis and sequencing with synthetic universal primers. In: Gene. Band 19, Nummer 3, Oktober 1982, S. 259–268, ISSN 0378-1119. PMID 6295879.
- Sue Lin-Chao, Wen-Tsuan Chen, Ten-Tsao Wong: High copy number of the pUC plasmid results from a Rom/Rop-suppressible point mutation in RNA II. In: Molecular Microbiology. Band 6, Nr. 22, November 1992, ISSN 0950-382X, S. 3385–3393, doi:10.1111/j.1365-2958.1992.tb02206.x, PMID 1283002.
- C.Helmer-Citterich, M., Anceschi, M. M., Banner, D. W. & Cesareni, G. (1988), Control of ColE1 replication: low affinity specific binding of Rop (Rom) to RNAI and RNAII. The EMBO journal 7(2), 557–66.
- Dominic Störkle, Sabrina Duschner, Nils Heimann, Michael Maskos, Manfred Schmidt: Complex Formation of DNA with Oppositely Charged Polyelectrolytes of Different Chain Topology: Cylindrical Brushes and Dendrimers. In: Macromolecules. Band 40, Nr. 22, 5. Oktober 2007, S. 7998–8006, doi:10.1021/ma0711689 (acs.org [abgerufen am 4. August 2016]).
Siehe auch
- pBR322 (Ausgangsvektor)
Weblinks
- Sequenz des pUC19c
- Informationen und Restriktionskarte von pUC18 und pUC19 (Memento vom 20. Februar 2009 im Internet Archive)