pBR322
Das Plasmid pBR322 wurde 1977 von Francisco Bolivar und Raymond Rodriguez im Labor von Herbert Boyer an der University of San Francisco (UCSF) konstruiert und war eines der ersten künstlich hergestellten Plasmide.[1] Es ist ein Derivat eines R-Plasmids, das einen Replikationsursprung ori (origin of replication), Gene für Ampicillin-Resistenz und Tetracyclin-Resistenz auf sich trägt. Im Unterschied zu gewöhnlichen R-Plasmiden fehlt ihm die Fähigkeit, sich auf andere Zellen selbständig zu übertragen (tra-Gene, oriT (origin of transfer)). Es war ein vielbenutzter Vektor, insbesondere in den 1980er Jahren. pBR322 wird neuerdings durch fortschrittlichere, einfacher zu handhabende Vektoren ersetzt, wie zum Beispiel pUC19 oder pBluescriptII.
Name
Der Name des Plasmids pBR322 setzt sich wie folgt zusammen:
- p steht für Plasmid,
- B und R stehen als Abkürzung für die Namen derjenigen, die das Plasmids konstruiert haben (F. Bolivar & R.L. Rodriguez),
- 322 ist eine fortlaufende Nummer, um ähnliche, aber nicht identische Plasmide unterscheiden zu können.
Konstruktion
Das Plasmid pBR322 wurde aus folgenden DNS-Elementen konstruiert:
- einem Ampicillin-Resistenzgen aus Tn3
- einem Tetracyclin-Resistenzgen aus pSC101
- einem DNA-Replikationsursprung (oriV) aus pMB1
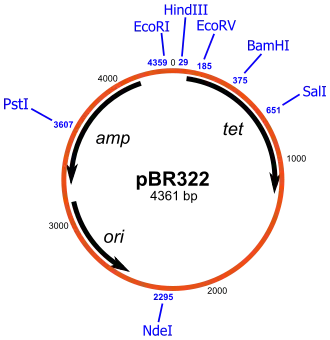
Das konstruierte Plasmid besteht aus 4361 Basenpaaren.[2] Das Ampicillinresistenzgen erstreckt sich von 4153 (Start) bis 3293, das Tetracyclinresistenzgen von Position 86 (Start) bis 1276 und der Replikationsursprung umfasst das Sequenzelement zwischen Position 3133 und 2519.[3]
Verwendung
Wesentlich für das Arbeiten mit dem Plasmid als Vektor sind die zahlreich vorhandenen Restriktionsendonuclease-Schnittstellen. Das Besondere ist auch die Tatsache, dass viele Schnittstellen nur an einer Stelle im Vektor auftreten. Beispielsweise finden sich BamHI-, HindIII oder SalI-Schnittstellen im Tetracyclinresistenzgen, eine PstI-Schnittstelle im Ampicillinresistenzgen sowie eine EcoRI- und eine NdeI-Schnittstelle in der nichtcodierenden Region des Vektors.
pBR322 wird bei Klonierungsarbeiten in E. coli verwendet. Hierbei kann man fremde DNS beispielsweise in den Vektor einbauen, indem man die Restriktionsschnittstellen des Vektors und der Fremd-DNS ausnutzt. Wenn man hierbei die Fremd-DNS in das Tetracyclinresistenzgen einfügt, bringt die nach Transformation dies Vorteile bei der Selektion der E. coli-Bakterien, die den pBR322-Vektor inklusive eingebauter Fremd-DNS tragen. Sie würden auf einem Tetracyclinmedium sensibel reagieren und absterben. Zur Selektion der gewünschten Zellen wird daher die Bakterienkultur zuerst auf einem Nährboden ausgestrichen, der das Antibiotikum Ampicillin enthält, um nur jene Bakterien zu erhalten, die das Plasmid (mit oder ohne eingebaute Fremd-DNS) durch Transformation erhalten haben. Danach wird ein Abdruck auf Nährböden gemacht, die mit Tetracyclin behandelt wurden (Replikaplattierung). An den Stellen, an denen die Kulturen sterben, sind also offensichtlich Kulturen vorhanden, die eine Empfindlichkeit auf Tetracyclin enthalten, was auf das Einfügen von Fremd-DNS zurückzuführen ist.
Die Entwicklung vieler moderner Klonierungsvektoren, wie die im Labor von Joachim Messing konstruierten pUC-Vektoren, z. B. pUC19, basiert auf dem Plasmid pBR322.
Einzelnachweise
- F. Bolivar, R.L. Rodriguez, P.J. Greene, M.C. Betlach, H.L. Heyneker, H.W. Boyer (1977): Construction and characterization of new cloning vehicles. In: Gene. Bd. 2, Nr. 2, S. 95–113. PMID 344137
- N. Watson (1988): A new revision of the sequence of plasmid pBR322. In: Gene. Bd. 70, Nr. 2, S. 399–403, PMID 3063608
- pBR322 Sequence and Map. In: snapgene.com. GSL Biotech LLC, abgerufen am 24. April 2017 (englisch).
Weblinks
- pBR322 in VectorDB (englisch)