Nonsense-mediated mRNA Decay
Als Nonsense-mediated mRNA Decay (NMD) wird ein Kontrollmechanismus in eukaryotischen Zellen bezeichnet, der unerwünschte (vorzeitige) Stopcodons in der mRNA erkennt und deren Expression als verkürzte Proteine verhindert.[1][2][3]
Prinzip
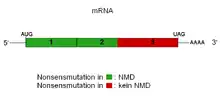
Bei einem vorzeitigen Stopcodon im offenen Leseraster einer mRNA wird der Nonsense-mediated mRNA Decay ausgelöst. Vorzeitige Stopcodons entstehen als Folge von Genmutationen (Nonsense-Mutationen, Trunkierende Mutationen), alternativem bzw. fehlerhaften Spleißen oder Fehlern bei der Transkription. Die entstehenden verkürzten Proteine können zu einem Funktionsverlust führen, jedoch in selteneren Fällen, z. B. bei der Entfernung regulatorischer Proteindomänen, auch zu einem Funktionsgewinn führen.
Bei der Transkription wird die Information der DNA auf die mRNA übertragen, welche jedoch zuerst als prä-mRNA, also als eine Vorstufe vorliegt, die noch prozessiert werden muss. Dabei werden unter anderem im Zuge des Spleißens die Introns entfernt und die Exons zusammengefügt. Dabei bleiben die Exon-Exon-Grenzen innerhalb der mRNA durch Proteinkomplexe markiert (engl. Exon-Junction-Complex). Diese Proteinkomplexe werden im Normalfall während der ersten Translationsrunde entfernt. Erst dann kommt die Translation am regulären Stopcodon zum Stillstand und die Terminationsfaktoren werden aktiviert. Befindet sich jedoch ein vorzeitiges Stopcodon im offenen Leseraster der mRNA, kommt die Translation schon dort zum Stillstand. Auch in diesem Fall werden die Terminationsfaktoren aktiviert. Da es sich jedoch um ein vorzeitiges Stopcodon handelt, an dem die Translation zum Stehen gekommen ist, befindet sich in der Regel (in 5’-3’-Richtung betrachtet) dahinter noch ein Exon-Junction-Komplex, mit dem die Terminationsfaktoren interagieren können.[4] Das hat zur Folge, dass unter anderem Decapping-Enzyme aktiviert werden, welche die 5’-Kappe der mRNA entfernen, was einen schnellen Abbau der mRNA durch 5’-3’-Exonukleasen zur Folge hat. Die Voraussetzung für eine Interaktion der Terminationsfaktoren mit den Exon-Junction-Komplexen ist jedoch, dass sich das vorzeitige Stopcodon mindestens 50-55 Basenpaare (in 5’-3’-Richtung betrachtet) vor der letzten Exon-Exon-Bindung befindet. Daher findet der Mechanismus des NMD nicht statt:
- wenn die prä-mRNA keine Introns hat
- wenn sich das vorzeitige Stopcodon im letzten Exon (bzw. in den letzten 50 Basenpaaren des vorletzten) befindet
Der Nonsense-mediated mRNA Decay besteht aus drei konservierten Teilen, UPF1, UPF2 und UPF3 (bzw. UPF3A und UPF3B in Menschen).[5] Diese drei Faktoren werden up-frameshift proteins genannt. In Säugern sind UPF2 und UPF3 Teil des exon junction complex (EJC) nach dem Spleißvorgang,[6] im Verbund mit den Proteinen eIF4AIII, MLN51 und dem Y14/MAGOH Heterodimer. Der EJC-Proteinkomplex bleibt bei einem vorzeitigen Stopcodon an die mRNA gebunden. Dadurch kommt UPF1 als Teil des Terminationskomplexes mit SMG-1, eRF1 und eRF2 bei der Bindung an die mRNA in die Nähe des UPF2 und des UPF3, wodurch die Phosphorylierung des UPF1 erfolgen kann. Das phosphorylierte UPF3 bindet SMG-5, SMG-6 und SMG-7, die die Dephosphorylierung des UPF3 einleitet. Anschließend erfolgt ein Abbau der defekten mRNA in P-bodies im Zytosol, beginnend mit der Entfernung der Cap-Struktur durch die Exoribonuklease XNR1.
Parallel zur NMD laufen die nonsense-mediated translational repression (NMTR), das nonsense-associated alternative splicing (NAS) und das nonsense-mediated transcriptional gene silencing (NMTGS) ab.[7]
Der NMD ist phylogenetisch hochkonserviert und wurde bisher in jedem untersuchten eukaryotischen Lebewesen gefunden. In unveränderten Zellen spielt NMD wahrscheinlich eine Rolle bei der Qualitätskontrolle von Mechanismen, die die Diversität der Genexpression erhöhen (z. B. Alternatives Spleißen oder V(D)J-Rekombination als Beispiel der somatischen Rekombination). Der NMD hat vor allem bei der Modulation des Schweregrads von verschiedenen Krankheitsbildern große medizinische Bedeutung, wobei der Effekt auf den Organismus positiv aber auch negativ sein kann.
Im Falle von ererbten oder im Rahmen einer Erkrankung erworbenen Genmutationen dient der NMD der Elimination von mRNA-Transkripten, die für verkürzte Proteine kodieren würden. Heterozygote Träger von Nonsensmutationen werden so vor eventuell toxischer Wirkung verkürzter Proteine geschützt. Es werden jedoch auch Nonsens-mRNAs eliminiert, die zur Synthese zumindest teilfunktioneller Proteine geführt hätten, was negative Auswirkungen auf den Organismus haben kann.
β-Thalassämie
Ein Beispiel für den "schadensbegrenzenden" Effekt von NMD stellt die autosomal-rezessiv vererbte β-Thalassämie dar. Für die Ausprägung dieser Krankheit gibt es drei verschiedene Möglichkeiten:
- Homozygote Thalassämie (Thalassaemia major, schwere Erkrankung)
- In beiden Kopien der β-Globin-Gene befinden sich Nonsensmutationen, so dass die entsprechenden mRNAs durch NMD abgebaut werden und als Folge keine β-Globinketten gebildet werden.
- Heterozygote Thalassämie (Thalassaemia minor, milde Erkrankung)
- Nonsensmutationen befinden sich nur in einer Kopie des β-Globin-Gens. Durch NMD werden die mRNAs des fehlerhaften Gens abgebaut, so dass nur das gesunde Gen exprimiert wird.
- Heterozygote Thalassämie (Thalassaemia intermedia, mittelschwere Erkrankung)
- Die Nonsensmutation befindet sich im letzten Exon der mRNA (bzw. in den letzten 50 Basenpaaren des vorletzten). Es erfolgt also keine Aktivierung des NMD (Grund: siehe oben) und es werden sowohl das gesunde β-Globin als auch das verkürzte β-Globin gebildet. Die Folge davon ist, dass die Erythrozyten, in die das fehlerhafte β-Globin eingebaut wird, zu Grunde gehen und somit eine mittelschwere Ausprägung der Erkrankung vorliegt.
Duchenne-Muskeldystrophie
Die Duchenne-Muskeldystrophie (DMD) ist eine X-chromosomal-rezessiv vererbte degenerative Muskelerkrankung. Dementsprechend erkranken fast ausschließlich Männer, da das ergänzende Y-Chromosom keine Dominanzfunktion hat. Eine Mutation am Dystrophin-Gen verursacht einen Frameshift mit einer Nonsensmutation in entsprechender mRNA als Folge. Durch den NMD erfolgt ein Abbau der mRNA, was einen Totalverlust des Dystrophin-Proteins als Folge hat. Somit entstehen auch keine verkürzten aber eventuell doch noch funktionellen oder teilfunktionellen Proteine.
Eine wichtige Rolle spielt der NMD auch bei Krebserkrankungen, bei denen es häufig zu einer Häufung von Mutationen in den kranken Zellen kommt.
Literatur
- B. P. Lewis, R. E. Green, S. E. Brenner: Evidence for the widespread coupling of alternative splicing and nonsense-mediated mRNA decay in humans. In: Proc. Natl. Acad. Sci. USA 100(1), 2003, S. 189–192. PMID 12502788 (PDF)
- E. N. Noensie, H. C. Dietz: A strategy for disease gene identification through nonsense-mediated mRNA decay inhibition. In: Nat. Biotechnol. 19(5), 2001, S. 434–439. PMID 11329012. doi:10.1038/88099
- Y. Ishigaki, X. Li, G. Serin, L. E. Maquat: Evidence for a pioneer round of mRNA translation: mRNAs subject to nonsense-mediated decay in mammalian cells are bound by CBP80 and CBP20. In: Cell. Band 106, Nr. 5, September 2001, S. 607–617, PMID 11551508.
- N. Oh, K. M. Kim, H. Cho, J. Choe, Y. K. Kim: Pioneer round of translation occurs during serum starvation. In: Biochem. Biophys. Res. Commun. Band 362, Nr. 1, Oktober 2007, S. 145–151, doi:10.1016/j.bbrc.2007.07.169, PMID 17693387.
Einzelnachweise
- Yao-Fu Chang, J. Saadi Imam, Miles F. Wilkinson: The Nonsense-Mediated Decay RNA Surveillance Pathway. In: Annual Review of Biochemistry. 2007, S. 51–74.
- P. A. Frischmeyer, H. C. Dietz: Nonsense-mediated mRNA decay in health and disease. In: Hum. Mol. Genet. Band 8, Nr. 10, 1999, S. 1893–1900. PMID 10469842. (PDF)
- P. H. Byers: Killing the messenger: new insights into nonsense-mediated mRNA decay. In: J. Clin. Invest. Band 109, Nr. 1, 2002, S. 3–6. PMID 11781342. (PDF)
- C. Schweingruber, S. C. Rufener, D. Zünd, A. Yamashita, O. Mühlemann: Nonsense-mediated mRNA decay - mechanisms of substrate mRNA recognition and degradation in mammalian cells. In: Biochim Biophys Acta. Band 1829, Nr. 6–7, 2013, S. 612–623. doi:10.1016/j.bbagrm.2013.02.005. PMID 23435113.
- I. Behm-Ansmant: Quality control of gene expression: a stepwise assembly pathway for the surveillance complex that triggers nonsense-mediated mRNA decay. In: Genes & Development. 20, Nr. 4, 2006, ISSN 0890-9369, S. 391–398. doi:10.1101/gad.1407606.
- L. E. Maquat: Nonsense-mediated mRNA decay in mammals. In: J. Cell. Sci. 118(Pt 9), 2005, S. 1773–1776. PMID 15860725.
- J. Hwang, Y. K. Kim: When a ribosome encounters a premature termination codon. In: BMB Rep. Band 46, Nr. 1, 2013, S. 9–16. PMID 23351378. (PDF) (Memento des Originals vom 7. März 2016 im Internet Archive) Info: Der Archivlink wurde automatisch eingesetzt und noch nicht geprüft. Bitte prüfe Original- und Archivlink gemäß Anleitung und entferne dann diesen Hinweis.