Hierarchische Clusteranalyse
Als hierarchische Clusteranalyse bezeichnet man eine bestimmte Familie von distanzbasierten Verfahren zur Clusteranalyse (Strukturentdeckung in Datenbeständen). Cluster bestehen hierbei aus Objekten, die zueinander eine geringere Distanz (oder umgekehrt: höhere Ähnlichkeit) aufweisen als zu den Objekten anderer Cluster. Man kann die Verfahren in dieser Familie nach den verwendeten Distanz- bzw. Proximitätsmaßen (zwischen Objekten, aber auch zwischen ganzen Clustern) und nach ihrer Berechnungsvorschrift unterscheiden.
Untergliedert man nach der Berechnungsvorschrift, so unterscheidet man zwei wichtige Typen von Verfahren:
- die divisiven Clusterverfahren, in denen zunächst alle Objekte als zu einem Cluster gehörig betrachtet und dann schrittweise die bereits gebildeten Cluster in immer kleinere Cluster aufgeteilt werden, bis jeder Cluster nur noch aus einem Objekt besteht. (Auch bezeichnet als „Top-down-Verfahren“)
- die agglomerativen Clusterverfahren, in denen zunächst jedes Objekt einen Cluster bildet und dann schrittweise die bereits gebildeten Cluster zu immer größeren zusammengefasst werden, bis alle Objekte zu einem Cluster gehören. (Auch bezeichnet als „Bottom-up-Verfahren“)
Für beide Verfahren gilt, dass einmal gebildete Cluster nicht mehr verändert werden können. Die Struktur wird entweder stets nur verfeinert („divisiv“) oder nur vergröbert („agglomerativ“), so dass eine strikte Cluster-Hierarchie entsteht. An der entstandenen Hierarchie kann man nicht mehr erkennen, wie sie berechnet wurde.
Vorteile und Nachteile
Die Vorteile der hierarchischen Clusteranalyse sind die Flexibilität durch Verwendung komplexer Distanzmaße, dass das Verfahren außer der Distanzfunktion und der Fusionierungsmethode keine eigenen Parameter hat und dass das Ergebnis eine Cluster-Hierarchie ist, die auch Unterstrukturen erlaubt.
Der Nachteil ist der Analyseaufwand des Ergebnisses. Andere Verfahren, wie z. B. der k-Means-Algorithmus, DBSCAN oder der ebenfalls hierarchische OPTICS-Algorithmus, liefern eine einzelne Partitionierung der Daten in Cluster. Eine hierarchische Clusteranalyse liefert zahlreiche solcher Partitionierungen, und der Anwender muss sich entscheiden, wie er partitioniert. Dies kann aber auch ein Vorteil sein, da dem Anwender so eine Zusammenfassung des qualitativen Verhaltens des Clusterings gegeben wird. Diese Erkenntnis ist grundlegend für die topologische Datenanalyse im Allgemeinen, und persistenter Homologie im Speziellen.
Ein weiterer Nachteil der hierarchischen Clusteranalyse ist die Laufzeit-Komplexität. Eine agglomerative Berechnung kommt in der Praxis und Forschung sehr viel häufiger vor, da es in jedem Schritt Möglichkeiten gibt, Cluster zusammenzufassen, was zu einer naiven Gesamtkomplexität von führt. In speziellen Fällen sind jedoch Verfahren mit einer Gesamtkomplexität von bekannt. Divisiv gibt es aber naiv in jedem Schritt Möglichkeiten, den Datensatz zu teilen.
Als weiterer Nachteil der hierarchischen Clusteranalyse gilt, dass sie keine Clustermodelle liefert. So entstehen beispielsweise je nach verwendeten Maßen Ketteneffekte ("single-link effekt") und es werden aus Ausreißern oft winzige Cluster erzeugt, die nur aus wenigen Elementen bestehen. Die gefundenen Cluster müssen daher meist nachträglich analysiert werden, um Modelle zu erhalten.
Dendrogramm
- Datensatz und Dendrogramm
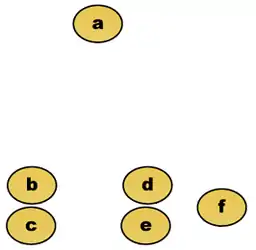 Datensatz. Die Objekte und sowie und liegen sehr dicht zusammen.
Datensatz. Die Objekte und sowie und liegen sehr dicht zusammen.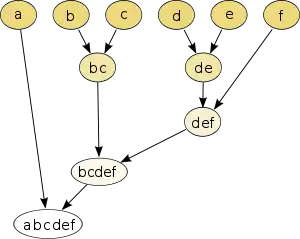 Dendrogramm für single-linkage. und sowie und werden als erstes zusammengefasst.
Dendrogramm für single-linkage. und sowie und werden als erstes zusammengefasst.
Zur Visualisierung der bei einer hierarchischen Clusterung entstehenden Baumstruktur kann das Dendrogramm (griech. δένδρον (dendron) = Baum) genutzt werden. Das Dendrogramm ist ein Baum, der die hierarchische Zerlegung der Datenmenge in immer kleinere Teilmengen darstellt. Die Wurzel repräsentiert ein einziges Cluster, das die gesamte Menge enthält. Die Blätter des Baumes repräsentieren Cluster, in denen sich je ein einzelnes Objekt der Datenmenge befindet. Ein innerer Knoten repräsentiert die Vereinigung aller seiner Kindknoten. Jede Kante zwischen einem Knoten und einem seiner Kindknoten hat als Attribut noch die Distanz zwischen den beiden repräsentierenden Mengen von Objekten.
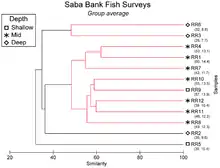
Das Dendrogramm wird informativer, wenn eine Achse zur Darstellung der Distanz oder (Un-)ähnlichkeit verwendet wird. Wenn zwei Cluster zusammengefügt werden, dann haben diese Cluster eine bestimmte Distanz oder (Un-)ähnlichkeit zueinander. Auf dieser Höhe wird der Verbindungsstrich gezeichnet. Im nebenstehenden Beispiel wurden z. B. die Objekte RR1 und RR4 bei einem Wert des Ähnlichkeitsmaßes von ca. 62 zusammengefügt. Die „Höhe“ ist hier die horizontale Achse.
In dieser Darstellung kann man eine gewünschte Zahl von Clustern auswählen, indem man das Dendrogramm auf einer geeigneten Höhe durchschneidet. Typischerweise sucht man eine Stelle, wo es zwischen zwei Fusionierungen einen großen Sprung der Distanz oder (Un-)ähnlichkeit gibt, z. B. im rechten Dendrogramm auf der Höhe 40. Dann ergeben sich vier Cluster, von denen 2 nur einzelne Objekte enthalten (RR2, RR5), ein Cluster enthält zwei Objekte (RR3 und RR6) und das letzte Cluster enthält alle übrigen Objekte. Gibt es hierarchische Cluster mit deutlich unterschiedlichen Größen, so kann es notwendig sein, auf unterschiedlichen Höhen zu zerlegen: während ein Cluster auf einer Höhe noch mit seinen Nachbarn verbunden ist, zerfällt ein anderer ("dünnerer") Cluster auf dieser Höhe schon in einzelne Objekte.
Distanz- und Ähnlichkeitsmaße
Sowohl in der agglomerativen als auch bei den divisiven hierarchischen Clusteranalysen ist es notwendig, Abstände bzw. (Un-)ähnlichkeiten zwischen zwei Objekten, einem Objekt und einem Cluster oder zwei Clustern zu berechnen. Je nach Skalenniveau der zugrunde liegenden Variablen kommen verschiedene Maße zum Einsatz:
- Bei kategorialen (nominalen und ordinalen) Variablen werden Ähnlichkeitsmaße benutzt, d. h. ein Wert von Null bedeutet, dass die Objekte eine maximale Unähnlichkeit haben. Diese können in Distanzmaße umgewandelt werden.
- Bei metrischen Variablen werden Distanzmaße benutzt, d. h. ein Wert von Null bedeutet, dass die Objekte einen Abstand von Null, also maximale Ähnlichkeit haben.
Die folgende Tabelle zeigt einige Ähnlichkeits- bzw. Distanzmaße für binäre und metrische Variablen. Kategorielle Variablen mit mehr als zwei Kategorien können in mehrere binäre Variablen umgewandelt werden. Die Gower Distanz kann auch für nominal skalierte Variablen definiert werden.
| Ähnlichkeitsmaß | Distanzmaß | ||
|---|---|---|---|
| Jaccard | |||
| Tanimoto | Euklidisch | ||
| Simple Matching | Pearson | mit die Standardabweichung der Variable | |
| Russel Rao | City-Block Manhattan | ||
| Dice | Gower | mit die Spannweite der Variable | |
| Kulczynski | Mahalanobis | mit die Kovarianzmatrix der Variablen |
Beispiele
- Ein Internetbuchhändler weiß für zwei Besucher, welche Buch-Webseiten sie sich angesehen haben, für jede der Webseiten wird also eine 0=nicht angesehen oder 1=angesehen gespeichert. Welches Ähnlichkeitsmaß bietet sich an, um zu erfahren, wie ähnlich die beiden Besucher sind? Die Anzahl der Buch-Webseiten, die sich keiner der beiden Besucher angesehen hat (), von denen es viele gibt, sollten in die Berechnung nicht einfließen. Ein möglicher Koeffizient wäre der Jaccard-Koeffizient, d. h. die Anzahl der Buch-Webseiten, die sich beide Besucher angesehen haben (), dividiert durch die Anzahl der Buch-Webseiten, die sich mindestens einer der beiden Besucher angesehen hat ( Anzahl der Buch-Webseiten, die sich nur der erste Besucher angesehen hat, und Anzahl der Buch-Webseiten, die sich nur der zweite Besucher angesehen hat).
- In den ALLBUS Daten wird u. a. nach der Einschätzung der aktuellen Wirtschaftslage mit den Antwortmöglichkeiten Sehr gut, Gut, Teils-Teils, Schlecht und Sehr Schlecht gefragt. Für jede der möglichen Antworten wird nun eine binäre Variable gebildet, so dass die binären Ähnlichkeitsmaße verwendet werden können. Zu beachten ist, dass bei mehreren Variablen mit unterschiedlichen Kategorienzahl noch eine Gewichtung bzgl. der Kategorienzahl stattfinden sollte.
- Im Iris Datensatz werden die vier () Abmessungen von Schwertlilienblütenblättern betrachtet. Um Abstände zwischen zwei Blütenblättern und zu berechnen, kann z. B. der euklidische Abstand benutzt werden.
Welches Ähnlichkeits- bzw. Distanzmaß verwendet wird, hängt letztlich von der gewünschten inhaltlichen Interpretation des Ähnlichkeits- bzw. Distanzmaßes ab.
Agglomerative Berechnung
Die agglomerative Berechnung einer hierarchischen Clusteranalyse ist der einfachste und flexibelste Fall. Zu Beginn wird zunächst jedes Objekt als ein eigener Cluster aufgefasst. Nun werden in jedem Schritt die jeweils einander nächsten Cluster zu einem Cluster zusammengefasst. Besteht ein Cluster aus mehreren Objekten, dann muss angegeben werden, wie die Distanz zwischen Clustern berechnet wird. Hier unterscheiden sich die einzelnen agglomerativen Verfahren. Das Verfahren kann beendet werden, wenn alle Cluster eine bestimmte Distanz/Ähnlichkeit zueinander überschreiten/unterschreiten oder wenn eine genügend kleine Zahl von Clustern ermittelt worden ist. Dies ist bei Clustern mit nur einem Objekt, wie sie zu Anfang vorgegeben sind, trivial.
Für die Durchführung einer agglomerativen Clusteranalyse müssen
- ein Distanz- oder Ähnlichkeitsmaß zur Bestimmung des Abstandes zwischen zwei Objekten und
- ein Fusionierungsalgorithmus zur Bestimmung des Abstandes zwischen zwei Clustern ausgewählt werden.
Dabei ist die Wahl des Fusionierungsalgorithmus oft wichtiger als die des Distanz- oder Ähnlichkeitsmaßes.
Fusionierungsalgorithmen
Die folgende Tabelle zeigt eine Übersicht über gängige Fusionierungsalgorithmen. Der Abstand zwischen Cluster und dem neuen Cluster wird oft über den Abstand oder die Unähnlichkeit von zwei Objekten berechnet. Der neue Cluster B entsteht aus der Fusion des "grünen" und "blauen" Clusters.
| Single-Linkage[1][2][3][4] | 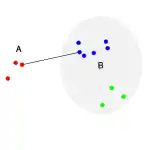 |
|---|---|
| Minimaler Abstand aller Elementpaare aus den beiden Clustern . Dieses Verfahren neigt zur Kettenbildung. | |
| Complete-Linkage[5] | 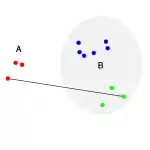 |
| Maximaler Abstand aller Elementpaare aus den beiden Clustern . Dieses Verfahren neigt zur Bildung kleiner Gruppen. | |
| Average-Linkage, Weighted Pair-Group Method using arithmetic Averages (WPGMA)[6] | 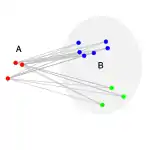 |
| Durchschnittlicher Abstand aller Elementpaare aus den beiden Clustern | |
| Average-Group-Linkage, McQuitty, Unweighted Pair-Group Method using arithmetic Averages (UPGMA)[6] | 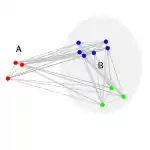 |
| Durchschnittlicher Abstand aller Elementpaare aus der Vereinigung von A und B | |
| Centroid-Method, Unweighted Pair-Group Method using Centroids (UPGMC)[6] | 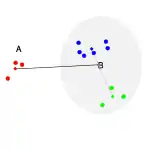 |
| Abstand der Zentren der beiden Cluster wobei das Zentrum des Clusters sei, das des Clusters . | |
| Median-Method, Weighted Pair-Group Method using Centroids (WPGMC)[7] | 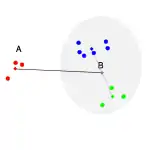 |
| Abstand der Zentren der beiden Cluster wobei das Zentrum des Clusters sei, der Mittelwert aus den Clusterzentren des grünen und blauen Clusters. |
Weitere Methoden sind:
- Ward’s minimum variance[8]
- Zunahme der Varianz beim Vereinigen von A und B
wobei das Zentrum des Clusters sei, das des Clusters . Dieses Verfahren neigt zur Bildung von gleich großen Clustern.
- EML
- Die Distanz zwischen zwei Clustern wird bestimmt durch die Maximierung der likelihood unter den Annahmen, dass die Cluster multivariat normalverteilt mit gleichen Kovarianzmatrizen, aber unterschiedlichen Größen. Das Verfahren ist ähnlich wie Ward’s minimum variance, jedoch neigt zur Bildung unterschiedlich großer Cluster.
Von praktischer Relevanz ist hierbei vor allem single linkage, da es mit dem Algorithmus SLINK eine effiziente Berechnungsmethode erlaubt.
Beispiele zu Fusionierungsalgorithmen
Besonders deutlich wird dies im zweiten Schritt des Algorithmus. Bei der Verwendung eines bestimmten Distanzmaßes wurden im ersten Schritt die beiden einander nächsten Objekte zu einem Cluster fusioniert. Dies kann wie folgt als Distanzmatrix dargestellt werden:
| Distanz zw. | Cluster1 Objekt1 | Cluster2 Objekt2 | Cluster3 Objekt3 | Cluster4 Objekt4 |
|---|---|---|---|---|
| Objekt1 | 0 | |||
| Objekt2 | 4 | 0 | ||
| Objekt3 | 7 | 5 | 0 | |
| Objekt4 | 8 | 10 | 9 | 0 |
Die kleinste Distanz findet sich zwischen dem Objekt1 und Objekt2 (rot in der Distanzmatrix) und man würde daher Objekt1 und Objekt2 zu einem Cluster zusammenfassen (fusionieren). Nun muss die Matrix neu erstellt werden ("o." steht für oder), das heißt die Distanz zwischen dem neuen Cluster und Objekt3 bzw. Objekt4 muss neu berechnet werden (gelb in der Distanzmatrix):
| Distanz zw. | Cluster1 Objekt1&2 | Cluster2 Objekt3 | Cluster3 Objekt4 |
|---|---|---|---|
| Objekt1&2 | 0 | ||
| Objekt3 | 7 o. 5 | 0 | |
| Objekt4 | 8 o. 10 | 9 | 0 |
Welcher der beiden Werte für die Distanzbestimmung relevant ist, bestimmt das Verfahren:
| Verfahren | Distanz zw. | Cluster1 Objekt1&2 | Cluster2 Objekt3 | Cluster3 Objekt4 |
|---|---|---|---|---|
|
Objekt1&2 | 0 | ||
| Objekt3 | 5 | 0 | ||
| Objekt4 | 8 | 9 | 0 | |
|
Objekt1&2 | 0 | ||
| Objekt3 | 7 | 0 | ||
| Objekt4 | 10 | 9 | 0 | |
|
Objekt1&2 | 0 | ||
| Objekt3 | 6 | 0 | ||
| Objekt4 | 9 | 9 | 0 | |
|
Objekt1&2 | 0 | ||
| Objekt3 | 5,3 | 0 | ||
| Objekt4 | 7,3 | 9 | 0 | |
|
Objekt1&2 | 0 | ||
| Objekt3 | 5 | 0 | ||
| Objekt4 | 8 | 9 | 0 |
Density Linkage
Beim Density Linkage wird für jedes Objekt ein Dichtewert geschätzt. Zur Berechnung wird eines der üblichen Distanzmaße, z. B. euklidischer Abstand, Manhattan-Distanz, zwischen den Objekten benutzt. Auf Basis der Dichtewerte zweier Objekte wird dann eine neue Distanz zwischen ihnen berechnet. Diese hängen auch von der Umgebung der Objekte und ab. Für das agglomerative Clustering kann dann eine der vorhergehenden Fusionierungsmethoden verwendet werden.
- Uniform kernel
- Lege den Radius fest
- Schätze die Dichte als den Anteil der Beobachtungen, die eine Entfernung kleiner gleich vom Objekt haben
- Berechne die Distanz zwischen Objekt und als
- nearest neighbour
- Lege die Anzahl der Nachbarn fest
- Berechne die Distanz zum nächsten Nachbarn des Objektes
- Schätze die Dichte als den Anteil der Beobachtungen, die eine Entfernung kleiner gleich vom Objekt haben, dividiert durch das Volumen der Sphäre mit dem Radius
- Berechne die Distanz zwischen den Objekten und als
- Wongs Hybrid
- Führe zunächst ein k-means Clustering durch und betrachte nur die Cluster-Schwerpunkt
- Berechne für jeden Cluster die totale Varianz
- Berechne die Distanz zwischen Cluster-Schwerpunkten und als
- Die Cluster und heißen benachbart, wenn gilt .
Ein Problem der Density linkage Algorithmen ist die Festlegung der Parameter.
Die Algorithmen OPTICS und HDBSCAN* (eine hierarchische Variante von DBSCAN clustering) können ebenfalls als hierarchisches Density Linkage clustering interpretiert werden.
Lance und Williams Formel
Zum Fusionieren der Cluster ist es jedoch nicht notwendig, immer wieder die Distanzen zwischen den Objekten neu zu berechnen. Stattdessen startet man wie in obigem Beispiel mit einer Distanzmatrix. Steht fest, welche Cluster fusioniert werden, so müssen nur die Distanzen zwischen dem fusionierten Cluster und allen anderen Clustern neu berechnet werden. Jedoch kann die neue Distanz zwischen dem fusionierten Cluster und einem anderen Cluster aus den alten Distanzen mit Hilfe der Formel von Lance und Williams berechnet werden:
Lance und Williams haben auch eine eigene Fusionierungsmethode auf Basis ihrer Formel angegeben: Lance-Williams Flexible-Beta.
Für die verschiedenen Fusionierungsmethoden ergeben sich verschiedene Konstanten , , und , die der folgenden Tabelle entnommen werden können. Dabei bedeutet die Anzahl der Objekte im Cluster .
| Methode | ||||
|---|---|---|---|---|
| Single linkage | 1/2 | 1/2 | 0 | -1/2 |
| Complete linkage | 1/2 | 1/2 | 0 | 1/2 |
| Median | 1/2 | 1/2 | -1/4 | 0 |
| Unweighted Group Average linkage (UPGMA) | 0 | 0 | ||
| Weighted Group Average linkage | 1/2 | 1/2 | 0 | 0 |
| Centroid | − | 0 | ||
| Ward | − | 0 | ||
| Lance-Williams Flexible-Beta | Standardwert oft | 0 |
SLINK und CLINK
Während die naive Berechnung einer hierarchischen Clusteranalyse eine schlechte Komplexität hat (bei komplexen Ähnlichkeitmaßen kann eine Laufzeit von oder auftreten), so gibt es für manche Fälle effizientere Lösungen.
So gibt es für single-linkage ein agglomeratives optimal effizientes Verfahren namens SLINK[9] mit der Komplexität , und eine Verallgemeinerung davon auf complete-linkage CLINK[10] ebenfalls mit der Komplexität . Für andere Fusionierungsmethoden wie Average-Linkage sind keine effizienten Algorithmen bekannt.[11]
Beispiel

Der Schweizer Banknoten-Datensatz besteht aus 100 echten und 100 gefälschten Schweizer 1000 Franken-Banknoten. An jeder Banknote wurden sechs Variablen erhoben:
- Die Breite der Banknote (WIDTH),
- die Höhe an der Banknote an der linken Seite (LEFT),
- die Höhe an der Banknote an der rechten Seite (RIGHT),
- der Abstand des farbigen Drucks zur Oberkante der Banknote (UPPER),
- der Abstand des farbigen Drucks zur Unterkante der Banknote (LOWER) und
- die Diagonale (links unten nach rechts oben) des farbigen Drucks auf der Banknote (DIAGONAL).
Als Distanzmaß bietet sich hier die euklidische Distanz an
- .
und für die folgende Grafiken wurden dann verschiedene hierarchische Clustermethoden angewandt. Jede Grafik besteht aus zwei Teilen:
- Im linken Teil werden die ersten zwei Hauptkomponenten der Daten gezeigt. Diese Darstellung wird gewählt, weil bei dieser (zweidimensionalen) Darstellung die Abstände in der Fläche gut den Abständen in sechsdimensionalen Raum entsprechen. Gibt es also zwei klar getrennte Cluster (Abstände zwischen den Clustern sind groß), so hofft man diese auch in dieser Darstellung zu sehen. Die Datenpunkte, die zu demselben Cluster gehören, sind mit der gleichen Farbe markiert; lediglich bei den schwarzen Datenpunkten ist es so, dass jeder Datenpunkt ein Cluster bildet.
- Im rechten Teil sehen wir das zugehörige Dendrogramm. Die „Height“ auf der y-Achse gibt an, bei welcher „Distanz“ Beobachtungen bzw. Cluster zu einem neuen Cluster zusammengefügt werden (entsprechend dem Fusionierungsalgorithmus). Gehören die beiden Teilcluster für eine Fusionierung zum selben Cluster, ist das Dendrogramm in der entsprechenden Farbe des Clusters gezeichnet; gehören sie zu unterschiedlichen Clustern, dann wird die Farbe schwarz benutzt. Die grauen Punkte links im Dendrogramm geben nochmal an, bei welcher „Distanz“ eine Fusionierung stattfand. Um eine gute Clusterzahl zu bestimmen, wird eine möglichst große Lücke bei den grauen Punkten gesucht. Denn eine große Lücke bedeutet, dass bei der nächsten Fusionierung eine große Distanz zwischen den zu fusionierenden Clustern besteht.
 Daten und Dendrogramm für das Average-linkage-Verfahren.
Daten und Dendrogramm für das Average-linkage-Verfahren. Daten und Dendrogramm mit der Ward-Methode.
Daten und Dendrogramm mit der Ward-Methode. Daten und Dendrogramm für das Complete-linkage-Verfahren.
Daten und Dendrogramm für das Complete-linkage-Verfahren. Daten und Dendrogramm für das Single-linkage-Verfahren.
Daten und Dendrogramm für das Single-linkage-Verfahren. Daten und Dendrogramm mit der Median-Methode.
Daten und Dendrogramm mit der Median-Methode. Daten und Dendrogramm mit der Centroid-Methode.
Daten und Dendrogramm mit der Centroid-Methode.
Divisive Berechnung
Wie oben angesprochen gibt es theoretisch Möglichkeiten, einen Datensatz mit Objekten in zwei Teile zu teilen. Divisive Verfahren brauchen daher normalerweise eine Heuristik, um Kandidaten zu generieren, die dann beispielsweise mit denselben Maßen wie in der agglomerativen Berechnung bewertet werden können.
Kaufman und Rousseeuw (1990) beschreiben eine Divisive Clustering Procedure (Diana) wie folgt:[12]
- Starte mit einem Cluster, der alle Beobachtungen enthält.
- Berechne den Durchmesser aller Cluster. Der Durchmesser ist die maximale Distanz oder Unähnlichkeit aller Objekte innerhalb des Clusters.
- Der Cluster mit dem größten Durchmesser wird in zwei Cluster geteilt.
- Dazu wird das Objekt in dem Cluster bestimmt, das die größte durchschnittliche Distanz oder Unähnlichkeit zu allen anderen Objekten hat. Es bildet den Kern der "Splittergruppe".
- Jedes Objekt, das näher an der Splittergruppe liegt als an den restlichen Objekten, wird nun der Splittergruppe zugeordnet.
- Die Schritte 2–5 werden solange wiederholt, bis alle Cluster nur noch ein Objekt enthalten.
Ein weiterer spezieller Algorithmus ist die Spektrale Relaxation.
Literatur
Grundlagen und Verfahren
- M. Ester, J. Sander: Knowledge Discovery in Databases. Techniken und Anwendungen. Springer, Berlin, 2000.
- K. Backhaus, B. Erichson, W. Plinke, R. Weiber: Multivariate Analysemethoden. Springer
- S. Bickel, T. Scheffer, Multi-View Clustering. Proceedings of the IEEE International Conference on Data Mining, 2004.
- J. Shi, J. Malik: Normalized Cuts and Image Segmentation. in Proc. of IEEE Conf. on Comp. Vision and Pattern Recognition, Puerto Rico 1997.
- L. Xu, J. Neufeld, B. Larson, D. Schuurmans: Maximum margin clustering. In: Advances in Neural Information Processing Systems. 17 (NIPS*2004), 2004
Anwendung
- J. Bacher, A. Pöge, K. Wenzig: Clusteranalyse – Anwendungsorientierte Einführung in Klassifikationsverfahren. 3. Auflage. Oldenbourg, München 2010, ISBN 978-3-486-58457-8.
- J. Bortz: Statistik für Sozialwissenschaftler. (Kap. 16, Clusteranalyse). Springer, Berlin 1999.
- W. Härdle, L. Simar: Applied Multivariate Statistical Analysis. Springer, New York 2003.
- C. Homburg, H. Krohmer: Marketingmanagement: Strategie – Instrumente – Umsetzung – Unternehmensführung. 3. Auflage. Kapitel 8.2.2, Gabler, Wiesbaden, 2009.
- H. Moosbrugger, D. Frank: Clusteranalytische Methoden in der Persönlichkeitsforschung. Eine anwendungsorientierte Einführung in taxometrische Klassifikationsverfahren. Huber, Bern 1992, ISBN 3-456-82320-7.
Einzelnachweise
- K. Florek, J. Łukasiewicz, J. Perkal, H. Steinhaus, S. Zubrzycki: Taksonomia wrocławska. In: Przegląd Antropol. 17, 1951, S. 193–211.
- K. Florek, J. Łukaszewicz, J. Perkal, H. Steinhaus, S. Zubrzycki: Sur la liaison et la division des points d’un ensemble fini. In: Colloquium Mathematicae. Vol. 2, No. 3–4, 1951, S. 282–285. Institute of Mathematics Polish Academy of Sciences.
- L. L. McQuitty: Elementary linkage analysis for isolating orthogonal and oblique types and typal relevancies. Educational and Psychological Measurement. 1957.
- P. H. Sneath: The application of computers to taxonomy. In: Journal of general microbiology. 17(1), 1957, S. 201–226.
- P. N. M. Macnaughton-Smith: Some Statistical and Other Numerical Techniques for Classifying Individuals. Research Unit Report 6. London: Her Majesty’s Stationary Office, 1965
- R. R. Sokal, C. D. Michener: A statistical method for evaluating systematic relationships. In: University of Kansas Science Bulletin 38, 1958, S. 1409–1438.
- J. C. Gower: A Comparison of Some Methods of Cluster Analysis. In: Biometrics 23.4, 1967, S. 623.
- J. H. Ward Jr: Hierarchical grouping to optimize an objective function. In: Journal of the American statistical association. 58(301), 1963, S. 236–244.
- R. Sibson: SLINK: an optimally efficient algorithm for the single-link cluster method. In: The Computer Journal. Band 16, Nr. 1. British Computer Society, 1973, S. 30–34 (web.archive.org [PDF; 3,1 MB; abgerufen am 25. Oktober 2021]).
- D. Defays: An efficient algorithm for a complete link method. In: The Computer Journal. Band 20, Nr. 4. British Computer Society, 1977, S. 364–366.
- Johannes Aßfalg, Christian Böhm, Karsten Borgwardt, Martin Ester, Eshref Januzaj, Karin Kailing, Peer Kröger, Jörg Sander, Matthias Schubert, Arthur Zimek: Knowledge Discovery in Databases, Kapitel 5: Clustering. In: Skript KDD I. 2003 (dbs.ifi.lmu.de [PDF]).
- L. Kaufman, P.J. Rousseeuw: Finding Groups in Data: An Introduction to Cluster Analysis. Wiley, New York 1990.