Chloramphenicol-Acetyltransferase
Chloramphenicol-Acetyltransferase (oder CAT) ist ein bakterielles Enzym (EC 2.3.1.28).[1][2] Es acetyliert das Antibiotikum Chloramphenicol unter Verbrauch von Acetyl-CoA an zwei Stellen. Da acetyliertes Chloramphenicol nicht mehr an Ribosomen bindet und damit nicht mehr die Translation hemmen kann, vermittelt das Gen eine Resistenz gegen dieses Antibiotikum. Die antibiotische Wirkung von Chloramphenicol wird genutzt, um bakterielles Wachstum zu inhibieren.
| Chloramphenicol-Acetyltransferase | ||
|---|---|---|
 | ||
| Andere Namen |
CAT | |
| Masse/Länge Primärstruktur | 25663 Da, 219 Aminosäuren | |
| Sekundär- bis Quartärstruktur | Homotrimer | |
| Bezeichner | ||
| Externe IDs |
| |
| Enzymklassifikation | ||
| EC, Kategorie | 2.3.1.28 | |
| Substrat | Chloramphenicol, Acetyl-CoA | |
| Produkte | Chloramphenicol-3-acetat | |
Struktur
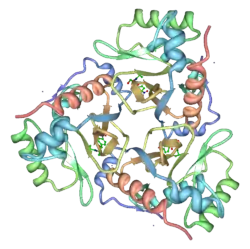
Die Kristallstruktur des Chloramphenicol-gebundenen Typ III-Enzyms aus Escherichia coli wurde schon 1990 aufgeklärt.[3] CAT ist ein Trimer gleicher Untereinheiten (Monomer Mr 25,000) dessen Struktur durch Wasserstoffbrückenbindungen stabilisiert ist. Chloramphenicol bindet eine tiefe Tasche benachbarter Untereinheiten, so dass die meisten Kontakte mit den Resten einer Untereinheit gebildet werden, während das katalytisch essentielle Histidin-195 der benachbarten Untereinheit für die Katalyse ideal positioniert ist.
Anwendung
Das Gen für Chloramphenicol-Acetyltransferase wird in der Mikrobiologie und Molekularbiologie als Resistenzgen eingesetzt, um transformierte Bakterien zu selektieren. Das cat-Gen wird auch als Reportergen verwendet, um z. B. Promotorstärken nach transienter Transfektion zu messen.[4] In einem typischen Versuch, dem sogenannten CAT-Assay, wird in der Regel 24–48 Stunden nach der Transfektion ein Zellextrakt erzeugt, in dem mit radioaktivem Chloramphenicol und Acetyl-CoA die Enzymaktivität bestimmt wird. Das Chloramphenicol wird extrahiert und durch Dünnschichtchromatographie aufgetrennt. Das acetylierte Produkt der Enzymreaktion läuft schneller als die nicht acetylierte Form. Durch Messung der Radioaktivität im Szintillationszähler oder mit einem Phosphoimager lässt sich der Substratumsatz messen. Die weitverbreitete Methode ist mit dem Aufkommen des einfacher und ohne radioaktive Substrate durchzuführenden Luciferase-Assays verdrängt worden.[5]
Einzelnachweise
- William V. Shaw: Chloramphenicol Acetyltransferase: Enzymology and Molecular Biology. In: Critical Reviews in Biochemistry and Molecular Biology. 14, Nr. 1, 1983, ISSN 1040-9238, S. 1–46. doi:10.3109/10409238309102789.
- Theodor Dingermann (Hrsg.), Rudolf Hänsel (Hrsg.) und Ilse Zündorf (Hrsg.): Pharmazeutische Biologie: Molekulare Grundlagen und klinische Anwendungen. Springer Verlag Berlin; 1. Auflage 2002; ISBN 3-540-42844-5; S. 301.
- A.G.W. Leslie: Refined crystal structure of type III chloramphenicol acetyltransferase at 1·75 Å resolution. In: Journal of Molecular Biology. 213, Nr. 1, 1990, ISSN 0022-2836, S. 167–186. doi:10.1016/S0022-2836(05)80129-9.
- C M Gorman, L F Moffat, B H Howard: Recombinant genomes which express chloramphenicol acetyltransferase in mammalian cells.. In: Molecular and Cellular Biology. 2, Nr. 9, 1982, ISSN 0270-7306, S. 1044–1051. doi:10.1128/MCB.2.9.1044.
- Steven R. Kain, Subinay Ganguly: Overview of Genetic Reporter Systems.. In: Current Protocols in Molecular Biology. 9, 2001. doi:10.1002/0471142727.mb0906s36.