Überdispersion
In der Statistik ist Überdispersion[1] (von lateinisch dispersio = „Zerstreuung“; gelegentlich auch Überstreuung oder Hyperdispersion genannt) ein Phänomen, das oft bei der Modellierung von Zähldaten auftritt. Man spricht von Überdispersion, wenn die empirische Varianz in den Daten größer ist, als die vom Modell (z. B. Binomialmodell oder Poisson-Modell) angenommene erwartete „theoretische Varianz“. Die tatsächlich gemessene Variation in den Daten übersteigt also die theoretisch erwartete Variation. Das Diagnostizieren von Überdispersion ist Thematik der Regressionsdiagnostik. Bei Vorliegen eines Poisson-Modells stellt die Anwesenheit von Überdispersion die häufigste in der Praxis auftretende Verletzung der Modellannahmen dieses Modells dar. In der Theorie gilt für eine Poisson-verteilte Zielgröße, dass Varianz und Erwartungswert gleich sind. In der Praxis übersteigt die empirisch beobachtete Varianz jedoch häufig den Erwartungswert. Die Zähldaten streuen also in einem größeren Maße um den Erwartungswert, als durch das Poisson-Modell erwartet wird.
Das Gegenstück der Überdispersion, bei der in der Praxis weniger Variation vorliegt als durch das Modell zu erwarten ist, wird Unterdispersion genannt. Sie tritt allerdings weniger häufig auf als Überdispersion. Die Anwesenheit von Überdispersion hat eine Reihe von negativen Konsequenzen für die Validität eines statistischen Modells und den daraus abgeleiteten Schlussfolgerungen. Beispielsweise ist es möglich, dass das Vorhandensein von Überdispersion die Parameterschätzer verzerrt. Die Hauptgründe für Überdispersion sind unbeobachtete Heterogenität und eine positive Korrelationen zwischen den individuellen Beobachtungen der binären Antwortvariablen.
Überdispersion findet in einer Reihe von biologischen Wissenschaften, wie der Parasitologie und Ökologie und in der Epidemiologie Anwendung.
Ursachen
Überdispersion tritt auf, wenn der Erwartungswert (selbst wenn alle erklärenden Variablen festgesetzt sind) eine gewisse inhärente Variabilität beibehält (sogenannte unbeobachtete Heterogenität). Des Weiteren tritt Überdispersion bei einer positiven Korrelation zwischen den individuellen Beobachtungen der binären Antwortvariablen (eine abhängige Variable, die nur zwei mögliche Werte annehmen kann) auf. Überdispersion tritt auch auf, wenn die Zähldaten in Clustern auftreten oder sich auf irgendeine Weise gemeinsam beeinflussen. Als Konsequenz sind die zugrundeliegenden Ereignisse positiv korreliert und eine Überdispersion der Zähldaten ist die Folge.[2] Auch können Ausreißer oder eine Fehlspezifikation des Regressionsmodells zu Überdispersion führen.[3]
Auswirkungen
Für verallgemeinerte lineare Modelle hat das Auftreten von Überdispersion schwerwiegende Konsequenzen. Es bedeutet, dass die vom verallgemeinerten linearen Modell ausgegebenen Standardfehler unterschätzt werden und Tests bzgl. der erklärenden Variablen im Allgemeinen signifikanter erscheinen als die Daten nahelegen. Die Konsequenz sind übermäßig komplexe Modelle, was wiederum zu weiteren Problemen führt.[4]
Aufdecken von Überdispersion
Überdispersion kann durch einen Anpassungstest festgestellt werden. Wenn die Residuendevianz und die Pearson-Chi-Quadrat-Statistik deutlich größer sind als die residualen Freiheitsgrade, ist entweder das angepasste Modell nicht adäquat (eine relevante Variable wurde ausgelassen) oder es liegt Überdispersion vor. Bleibt auch nach dem Anpassen eines Modells mit der stärksten Erklärungskraft (gesättigtes Modell) und nach dem Eliminieren von Ausreißern ein Anpassungsmangel bestehen, wäre die Anwesenheit von Überdispersion eine alternative Erklärung.[5]
Theoretische Beispiele
Poisson-Modell
Ein Charakteristikum der Poisson-Verteilung ist, dass der Parameter der Verteilung zugleich der Erwartungswert und Varianz darstellt (siehe Poisson-Verteilung#Eigenschaften). Daher gilt für ein Poisson-Modell, bei dem eine Poisson-Verteilte Antwortvariable (Zielgröße) vorliegt:
- .
Aus ähnlichen Gründen wie bei Binomialdaten wird bei Anwendungen der Poisson-Regression häufig eine signifikant höhere empirische Varianz beobachtet.[6] Die einfachste Möglichkeit einer größeren Variabilität als erwartet Rechnung zu tragen, ist die Einführung eines skalaren Faktors , der Überdispersionsparameter (auch Dispersionsfaktor) genannt wird.[7] In anderen wissenschaftlichen Disziplinen gibt es eine abweichende Notation; beispielsweise wird in der Parasitologie und Epidemiologie der Überdispersionsparameter mit [A 1] statt mit bezeichnet. Mithilfe dieses unbekannten Parameters (den es daher zu schätzen gilt) modifiziert man die Varianzformel wie folgt:
- .
Durch die Wahl wird ein Modell erzeugt, bei dem die Varianz größer als der Erwartungswert ist. Man spricht bei dieser Wahl von von einem poissonverteilten verallgemeinerten linearen Modell mit Überdispersion, obwohl genau genommen keine Poisson-Verteilung mehr vorliegt.[8] Für die Poisson-Verteilung in ihrer ursprünglichen Form, bei der die Varianz dem Erwartungswert entspricht, gilt . Man spricht in diesem Fall auch von Äquidispersion. Wählt man hingegen erhält man ein Modell mit Unterdispersion.[9]
Schätzung des Überdispersionsparameters
Mittlere Pearson-Chi-Quadrat-Statistik
Falls der Überdispersionseffekt signifikant ist, ist es sinnvoll den Überdispersionsparameter zu schätzen. Es ist möglich den Überdispersionsparameter mit der mittleren Pearson-Chi-Quadrat-Statistik zu schätzen.[10] Die Pearson-Chi-Quadrat-Statistik, die sich aus der Quadratsumme der Pearson-Residuen ergibt, ist gegeben durch
- .
Hierbei sind die bekannte spezifizierte Gewichte, ist der geschätzte Erwartungswert für Beobachtung und ist die Varianzfunktion. Der Pearson-Schätzer für gegeben ist durch die mittlere Pearson-Chi-Quadrat-Statistik . Diese ist analog zum erwartungstreuen Schätzer der Varianz der Störgrößen definiert, außer dass hier die Pearson-Chi-Quadrat-Statistik die Residuenquadratsumme ersetzt.[11] Sowohl die Residuendevianz als auch die Pearson-Chi-Quadrat-Statistik vergleichen die Anpassung eines Modells relativ zu einem gesättigten Modell und sind asymptotisch -verteilt.[12] Hierbei ist die Chi-Quadrat-Verteilung mit Freiheitsgraden.
Mittlere Residuendevianz
Eine weitere Maßzahl, die zur Schätzung des Überdispersionsparameters herangezogen werden kann, ist die mittlere Residuendevianz. Die Residuendevianz ist eine Maßzahl für die Variabilität der Beobachtungen, nachdem das Modell angepasst wurde. Sie ist definiert durch[13]
- .
Sie folgt ebenso wie die Pearson-Chi-Quadrat-Statistik einer -Verteilung mit Freiheitsgraden. Für lineare Regressionsmodelle entspricht sie gerade der Residuenquadratsumme. Wie gewöhnlich ist der Maximum-Likelihood-Schätzer nicht erwartungstreue für . Daher verwendet man als erwartungstreuen Schätzer für den Überdispersionsparameter die mittlere Residuendevianz .[14]
Unterschiede in der Terminologie zwischen den Disziplinen
Um zu betonen, dass in der Biologie zumeist klumpenartige Anordnungen (sogenannte Cluster) beschrieben werden, wird in der Biologie meist die Bezeichnung „Aggregation“ der Bezeichnung „Überdispersion“ vorgezogen und synonym zu ihr verwendet. Statt davon zu sprechen, dass die „Verteilung Überdispersion aufweist“, spricht man daher in biologischen Anwendungen (wie z. B. der Ökologie und der Parasitologie) oft von einer aggregativen Verteilung (auch geklumpte Verteilung oder gehäufte Verteilung genannt) der untersuchten Zähldaten.[15] Beispielsweise spricht man davon, dass Parasiten eine „aggregative Verteilung“ aufweisen.[16]
Anwendungen in der Biostatistik
Anwendung in der Parasitologie

Überdispersion charakterisiert in der Parasitologie ein Phänomen der Aggregation einer Mehrheit der Parasiten in einer Minderheit der Wirtspopulation. Somit hat die Mehrheit der Wirte keine oder nur wenige Parasiten. Eine sehr kleine Anzahl von Wirten trägt jedoch eine große Anzahl von Parasiten. Eine starke Überdispersion bzw. Aggregation lässt sich in den Daten für Nematodeninfektionen bei Teichfröschen feststellen. In diesem Fall wurden 70 % der Parasiten in nur 4 % der Wirte registriert, während 88 % der Wirte nicht infiziert waren und 8 % leichte Infektionen hatten.[17]
Die Parasitenaggregation in Bezug auf Wirte ist ein charakteristisches Merkmal vielzelliger Parasitenpopulationen.[18] Viele Parasiten, die durch direkten Kontakt übertragen werden (solche, die keinen Krankheitsüberträger verwenden), z. B. Ruderfußkrebse, Walläuse, Läuse, Milben, Hakensaugwürmer, viele Fadenwürmer, Pilze und viele Taxa von Protisten, sind nahezu ausnahmslos durch eine aggregative Verteilung charakterisiert, bei der die meisten einzelnen Wirte nur wenige oder keine Parasiten beherbergen.[19]
In der Theorie gibt es viele Erklärungen dafür, warum Parasitenpopulationen meist aggregativ verteilt sind. Zunächst ist es naheliegend, auch dass die Verteilung von Parasitenpopulationen nicht zufällig, sondern aggregativ ist, wenn schon die Übertragungsstadien nicht zufällig sind. Zudem können Wirte, die bereits befallen sind erfolgreicher parasitiert werden, da sie durch eine vorausgehende Parasitierung bereits geschwächt sind. Oft kommt erschwerend hinzu, dass sich Wirtsindividuen je nach Jahreszeit oder Alter erfolgreicher parasitieren lassen. Auch könnte sich die Befallstärke bei einem bereits parasitierten Wirtsindividuum erhöhen, falls sich die Parasiten ungeschlechtlich oder eingeschlechtlich vermehren.[20]
Anwendung in der Ökologie
Auch in der Ökologie werden häufig „klumpenartige“ Verteilungsmuster modelliert. Wenn ein Verteilungsmuster aus zufällig verstreuten „Klumpen“ (Clustern) besteht, liegt ein „klumpenartiges Verteilungsmuster“ vor. Bei einem solchen sind die Untersuchungseinheiten zu Haufen aggregiert. Eine Auszählung der Objekte würde in diesem Fall Untersuchungsgebiete mit sehr vielen Objekten und andere mit recht wenigen oder überhaupt keinem Objekt ergeben. Die Besetzungszahlen streuen also stark von einem Untersuchungsgebiet zum anderen.[21] Ein Beispiel wäre die Verbreitung der Amerikanerkrähe, die eine „aggregative Verteilung“ aufweist. Sie ist in Nordamerika sehr weit verbreitet, weist jedoch eine hohe Abundanz an einzelnen Aggregationspunkten auf und hat somit ein geklumptes Auftreten.
Auch hier gibt es vielfältige Ursachen für das geklumpte Auftreten von Arten. Beispielsweise können bewohnbare Lebensräume oder gewisse Nährstoffe inselartig über ein größeres Gebiet verbreitet sein. Zudem bilden viele Tierarten soziale Verbände, wie z. B. Herden oder Schwärme.[22]
Anwendung in der Epidemiologie
In der Epidemiologie bezeichnet Überdispersion eine hohe individuen-spezifische Variation in der Verteilung der Anzahl der Sekundärübertragungen, die zu „außergewöhnlichen Übertragungsereignissen“ (englisch superspreading events) führen kann.[23] Üblicherweise werden Zähldaten, wie die Anzahl der Sekundärübertragungen, mithilfe der Poisson-Verteilung modelliert. Allerdings bietet es bei Vorliegen von Überdispersion Vorteile, die Anzahl der Sekundärübertragungen statt mit einer Poisson-Verteilung mit einer negativen Binomialverteilung zu modellieren, da sie einen flexiblen Grad an Übertragungsheterogenität abbilden kann und zu Daten aus einer Reihe von Infektionskrankheiten passt.[24] Zudem scheitert die Poisson-Verteilung daran die relevanten Eigenschaften von „außergewöhnlichen Übertragungsereignissen“ zu erfassen, da sich in diesem Fall Erwartungswert und Varianz entsprechen (siehe #Poisson-Modell).[25] Aus oben genannten Gründen lässt sich die Anzahl der Sekundärübertragungen adäquat durch eine negative Binomialverteilung modellieren. Da sowohl die Basisreproduktionszahl (die „mittlere“ Anzahl an Sekundärübertragungen, die durch ein Individuum in einer anfälligen Population hervorgerufen wurde), als auch der Überdispersionsparameter (die individuelle Übertragungsheterogenität) Rückschlüsse auf die Dynamik eines Krankheitsausbruchs zulassen und beide Größen isoliert betrachtet wenig aussagekräftig sind, parametrisiert man oft die Wahrscheinlichkeitsfunktion der negativen Binomialverteilung durch diese beiden Größen gemeinsam ().[26] Anschließend lassen sich beide Größen, die nun die Verteilung charakterisieren, mittels statistischer Verfahren gemeinsam schätzen.
Allerdings ist man oft primär am geschätzten Überdispersionparameter interessiert, da er die Variabilität in der Anzahl der Sekundärfälle quantifiziert und somit als Maß für die Wirkung von Superspreading interpretiert werden kann. Da die Varianz der angenommenen negativen Binomialverteilung gegeben ist durch liegt ein hoher Grad an individuen-spezifischer Variabilität vor, wenn der Überdispersionsparameter klein, z. B. , ist. Für diesen Spezialfall weist die Verteilung der Sekundärübertragungen exponentielle Verteilungsenden auf, d. h. die Eintrittswahrscheinlichkeit von „außergewöhnlichen Übertragungsereignissen“ nimmt steigender Anzahl an Sekundärfällen exponentiell ab.[27]
Allgemein gilt: Je kleiner der Überdispersionsparameter ist, desto stärker ist die Wirkung von Superspreading.[28] Dagegen würde mit steigendem Überdispersionsparameter der Effekt von Superspreading auf die Epidemie abnehmen.[29] Die Interpretation des Überdispersionsparameters wird weiter vereinfacht, indem sich auf den Anteil der Individuen konzentriert wird, der für 80 % der Sekundärübertragungen verantwortlich ist (ein empirisches Muster, bekannt als 80/20-Regel). Ist der Überdispersionsparameter deutlicher kleiner als Eins () bzw. nahe bei Null, so approximiert er den Anteil infizierter Personen, die 80 % der gesamten Sekundärübertragungen verursachen. Beispielsweise würde ein geschätzter Überdispersionsparameter von 0,1 bedeuten, dass die infektiösesten 10 % der Personen etwa 80 % der gesamten Sekundärübertragungen verursachen.[30]
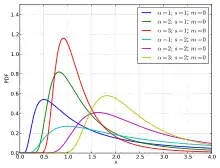
Zusätzlich gibt es für einige Erkrankungen empirische Belege dafür, dass die Verteilung der Anzahl der Sekundärübertragungen „fette Verteilungsenden“ aufweist, d. h. die Verteilung besitzt relativ viel Wahrscheinlichkeitsmasse am auslaufenden Ende der Verteilung. Diese spezielle Eigenschaft bedeutet, dass umso mehr statistische Information in den Extremen und weniger in den Ereignissen steckt, die häufig auftreten. Daher sind „außergewöhnliche Übertragungsereignisse“ zwar extreme, aber dennoch wahrscheinliche Ereignisse, die einen beträchtlichen Beitrag zur Gesamtübertragung leisten. Maßnahmen sollten daher an der endlastigen Natur der Verteilung ansetzen, indem man versucht das Risiko an den Verteilungsenden zu minimieren wie z. B. durch die Immunisierung ausgewählter Personen. Da fette Verteilungsenden jedoch inkonsistent mit der Annahme einer negativen Binomialverteilung sind, werden auch alternative Modellierungsmöglichkeiten zum Negativ-Binomialmodell vorgeschlagen.[32] Die folgende Tabelle gibt die geschätzten Überdispersionsparameter bei ausgewählten Erkrankungen an und ob es Hinweise auf fette Verteilungsenden bei der Verteilung der Sekundärübertragungen gibt:
| Krankheit | Geschätzter Überdispersionsparameter | Hinweise auf fette Verteilungsenden |
|---|---|---|
| COVID-19 | 0,1–0,6[33] | ja[34] |
| SARS | 0,16[35] | ja[36] |
| MERS | 0,25[37] | nicht bekannt |
| Spanische Grippe | 1[37] | nicht bekannt |
| Saisonale Grippe | 1[37] | nicht bekannt |
Bei COVID-19 geht man bislang von einem geschätzten Überdispersionsparameter von etwa 0,1–0,6 aus.[38][39] Laut einem Preprint von Akira Endo und Mitautoren, liegt bei COVID-19 – unter Annahme einer Basisreproduktionszahl von 2,5 – der Überdispersionsparameter mit hoher Glaubwürdigkeit etwa bei 0,1 (95 %-Glaubwürdigkeitsintervall: [0,05–0,2]).[40]
Anmerkungen
- Fälschlicherweise mit statt κ bezeichnet; in der Statistik werden zu schätzende Kenngrößen der Grundgesamtheit konventionell mit griechischen Buchstaben bezeichnet, während Kennwerte für die Stichprobe mit lateinische Buchstaben bezeichnet werden.
Einzelnachweise
- overdispersion. Glossary of statistical terms. In: International Statistical Institute. 1. Juni 2011, abgerufen am 11. September 2020 (englisch).
- Peter K. Dunn, Gordon K. Smyth: Generalized linear models with examples in R. Springer, New York 2018, S. 397.
- Lothar Kreienbrock, Iris Pigeot und Wolfgang Ahrens: Epidemiologische Methoden. 5. Auflage. Springer Spektrum, Berlin/ Heidelberg 2012, ISBN 978-0-19-975455-7, S. 3316.
- Peter K. Dunn, Gordon K. Smyth: Generalized linear models with examples in R. Springer, New York 2018, S. 347.
- Peter K. Dunn, Gordon K. Smyth: Generalized linear models with examples in R. Springer, New York 2018, S. 397.
- Ludwig Fahrmeir, Thomas Kneib, Stefan Lang, Brian Marx: Regression: models, methods and applications. Springer Science & Business Media, 2013, ISBN 978-3-642-34332-2, S. 294.
- L. Fahrmeir, A. Hamerle: Multivariate statistische Verfahren. de Gruyter, Berlin u. a. 1996, ISBN 3-11-008509-7, S. 294.
- Torsten Becker u. a.: Stochastische Risikomodellierung und statistische Methoden. Springer Spektrum, 2016, S. 308.
- Torsten Becker u. a.: Stochastische Risikomodellierung und statistische Methoden. Springer Spektrum, 2016, S. 308.
- Ludwig Fahrmeir, Thomas Kneib, Stefan Lang, Brian Marx: Regression: models, methods and applications. Springer Science & Business Media, 2013, ISBN 978-3-642-34332-2, S. 292.
- Peter K. Dunn, Gordon K. Smyth: Generalized linear models with examples in R. Springer, New York 2018, S. 255.
- Ludwig Fahrmeir, Thomas Kneib, Stefan Lang, Brian Marx: Regression: models, methods and applications. Springer Science & Business Media, 2013, ISBN 978-3-642-34332-2, S. 308.
- Peter K. Dunn, Gordon K. Smyth: Generalized linear models with examples in R. Springer, New York 2018, S. 248.
- Peter K. Dunn, Gordon K. Smyth: Generalized linear models with examples in R. Springer, New York 2018, S. 254.
- Robert Poulin: Evolutionary ecology of parasites. Princeton University Press, 2011, S. 90 ff.
- Theodor Hiepe, Horst Aspöck (Hrsg.): Allgemeine Parasitologie: mit den Grundzügen der Immunbiologie, Diagnostik und Bekämpfung. Georg Thieme Verlag, 2006, S. 278.
- Morgana Camacho u. a.: Recovering parasites from mummies and coprolites: an epidemiological approach. In: Parasites & vectors. Band 11, 2018, Artikel 248.
- Robert Poulin: Explaining variability in parasite aggregation levels among host samples. In: Parasitology. Band 140, Nr. 4, 2013, S. 541–546.
- Robert Poulin, Haseeb S. Randhawa: Evolution of parasitism along convergent lines: from ecology to genomics. In: Parasitology. Band 142, Suppl 1, 2015, S. S6–S15.
- C. Dieter Zander: Parasit-Wirt-Beziehungen: Einführung in die ökologische Parasitologie. Springer-Verlag, 2013, S. 37.
- Werner Timischl: Angewandte Statistik. Eine Einführung für Biologen und Mediziner. 3. Auflage. 2013, S. 78.
- Thomas M. Smith, Robert Leo Smith: Ökologie. Pearson Deutschland, 2009, S. 255.
- Akira Endo, Adam Kucharski, Sebastian Funk u. a.: Estimating the overdispersion in COVID-19 transmission using outbreak sizes outside China, Wellcome Open Research, 2020.
- Seth Blumberg, James O. Lloyd-Smith: Comparing methods for estimating R0 from the size distribution of subcritical transmission chains. In: Epidemics. Band 5, Nr. 3, 2013.
- Benjamin M. Althouse u. a.: Stochasticity and heterogeneity in the transmission dynamics of SARS-CoV-2. arXiv preprint (2020).
- Akira Endo, Adam Kucharski, Sebastian Funk u. a.: Estimating the overdispersion in COVID-19 transmission using outbreak sizes outside China. Wellcome Open Research, 2020.
- Felix Wong, James J. Collins: Evidence that coronavirus superspreading is fat-tailed. Proceedings of the National Academy of Sciences, 2020.
- Julien Riou, Christian L. Althaus: Pattern of early human-to-human transmission of Wuhan 2019 novel coronavirus (2019-nCoV), December 2019 to January 2020. In: Eurosurveillance. Band 25, Nr. 4, 2020, doi:10.2807/1560-7917.ES.2020.25.4.2000058, PMID 32019669, PMC 7001239 (freier Volltext).
- Benjamin M. Althouse u. a.: Stochasticity and heterogeneity in the transmission dynamics of SARS-CoV-2. arXiv preprint (2020).
- Bjarke Frost Nielsen, Kim Sneppen: COVID-19 superspreading suggests mitigation by social network modulation. medRxiv (2020).
- Felix Wong, James J. Collins: Evidence that coronavirus superspreading is fat-tailed. Proceedings of the National Academy of Sciences, 2020.
- Felix Wong, James J. Collins: Evidence that coronavirus superspreading is fat-tailed. Proceedings of the National Academy of Sciences, 2020.
- Felix Wong, James J. Collins: Evidence that coronavirus superspreading is fat-tailed. Proceedings of the National Academy of Sciences, 2020.
- Felix Wong, James J. Collins: Evidence that coronavirus superspreading is fat-tailed. Proceedings of the National Academy of Sciences, 2020.
- J. O. Lloyd-Smith, S. J. Schreiber, P. E. Kopp, W. M. Getz: Superspreading and the effect of individual variation on disease emergence. In: Nature. Band 438, 2005.
- Felix Wong, James J. Collins: Evidence that coronavirus superspreading is fat-tailed. Proceedings of the National Academy of Sciences, 2020.
- Wie Superspreader die Pandemie beeinflussen. Abgerufen am 31. Mai 2020.
- Bjarke Frost Nielsen, Kim Sneppen: COVID-19 superspreading suggests mitigation by social network modulation. medRxiv (2020).
- Felix Wong, James J. Collins: Evidence that coronavirus superspreading is fat-tailed. Proceedings of the National Academy of Sciences, 2020.
- Akira Endo, Adam Kucharski, Sebastian Funk u. a.: Estimating the overdispersion in COVID-19 transmission using outbreak sizes outside China. Wellcome Open Research, 2020.