Einzeldomänenantikörper
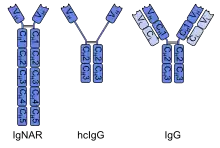
Einzeldomänenantikörper, auch Nanobodies oder Nanoantikörper genannt, sind Antikörperfragmente, die aus einer einzelnen, monomeren variablen Domäne eines Antikörpers aufgebaut sind. Im einfachsten Fall bestehen sie aus den monomeren variablen Domänen von Schwere-Ketten-Antikörpern, die von Vertretern der Familie der Kamele und von Knorpelfischen produziert werden, und werden je nach Herkunft auch als VHH- oder VNAR-Fragmente (Variable New Antigen Receptor) bezeichnet. Alternativ dazu können Einzeldomänenantikörper auch durch Monomerisierung der dimeren variablen Domänen konventioneller Antikörper von Maus oder Mensch mit Hilfe gentechnischer Methoden gewonnen werden. Mit einer Molekülmasse von etwa 12–15 kDa sind sie die kleinsten Antikörperfragmente, die zur Antigenerkennung befähigt sind. Auf Grund ihrer geringen Molekülmasse und wegen ihrer besonderen physikochemischen Eigenschaften besteht die Hoffnung, dass Einzeldomänenantikörper als neuartige Arzneistoffe zur Behandlung von Krankheiten wie dem akuten Koronarsyndrom oder der rheumatoiden Arthritis eingesetzt werden können.[1]
.png.webp)
Eigenschaften
Die aus einer Aminosäurekette bestehenden variablen Domänen von Schwerkettenantikörpern unterscheiden sich von denen konventioneller Antikörper, die aus zwei Aminosäureketten bestehen, durch eine geringere Lipophilie, haben deshalb eine gute Löslichkeit in wässrigen Medien und eine hohe Hitzebeständigkeit. Im Gegensatz zu klassischen Antikörpern, die durch Hitze inaktiviert werden können, behalten Einzeldomänenantikörper auch nach einer Hitzebehandlung bei 90 °C ihre Fähigkeit der Antigenbindung.[2] Einzeldomänenantikörper sind beständiger gegenüber Magensäure und dank einer geringeren Anzahl an Spaltungsstellen resistenter gegenüber proteolytischen Enzymen als klassische Antikörper.[3] Diese Beständigkeit lässt sich durch weitere Optimierung der Struktur der Einzeldomänenantikörper steigern, sodass sie auch den Magen-Darm-Trakt passieren können und sich für eine lokal-perorale Anwendung eignen.[4] Eine für peptidische Arzneistoffe charakteristische niedrige Resorptionsquote schränkt jedoch eine mögliche systemische Anwendung ein. Dank ihrer Beständigkeit in Gegenwart von Detergenzien sind sie auch für einen Einsatz in Shampoos geeignet.[5] Verbunden mit der geringen Molekülgröße von Einzeldomänenantikörpern ist eine gegenüber konventionellen Antikörpern verbesserte Gewebepermeabilität. Da ihre Molekülmasse deutlich unter der Nierenschwelle liegt, besitzen sie eine sehr kurze Plasmahalbwertzeit und können über die Nieren und den Urin ausgeschieden werden.[6] Ebenso zeigen sie durch das Fehlen des FC-Fragments keine auf eine Aktivierung des Komplementsystems zurückzuführende Zytotoxizität.
Einzeldomänenantikörper, die aus Schwerkettenantikörpern von Kamelen oder Knorpelfischen gewonnen wurden, besitzen im Vergleich zu den variablen Domänen klassischer Antikörper ausgeprägte antigenerkennende Loop-Strukturen. Dank dieser sind Einzeldomänenantikörper in der Lage, versteckte Antigenstrukturen zu erkennen, die für klassische Antikörper unerreichbar bleiben.[3] Dazu gehören beispielsweise katalytische Zentren von Enzymen[6] und die Ligandenbindungsdomänen von G-Protein-gekoppelten Rezeptoren[7].
Herstellung
Einzeldomänenantikörper aus Schwerkettenantikörpern
Die Generierung und Selektion von Antigen-spezifischen Einzeldomänenantikörpern abgeleitet von Schwere-Ketten-Antikörpern erfolgt in der Regel durch Immunisierung von Dromedaren, Trampeltieren, Lamas oder Alpakas und im Anschluss daran durch Isolierung der Schwerkettenantikörper-codierenden mRNA. Auch Haie haben sich als Spender für Schwerkettenantikörper und ihrer codierenden mRNA etabliert. Aus der Gesamtheit der so isolierten mRNA wird mit Hilfe molekularbiologischer Methoden, wie reverser Transkription und Polymerasekettenreaktion, eine Immunbibliothek von Einzeldomänenantikörpern erstellt, die mehrere Millionen Klone enthält. Mit Hilfe eines Screenings unter Verwendung von sogenannten Display-Techniken, wie dem Phagen-Display oder dem Ribosomen-Display, werden daraus die Antigen-bindenden Klone identifiziert.[8]
Antigenbindende Klone können auch aus nicht immunen oder naiven Bibliotheken (die aus der mRNA nicht explizit immunisierter Tiere erstellt werden) selektiert werden. Da die Affinität der so gewonnenen Einzeldomänenantikörper zum Antigen meist sehr gering ist, wird insbesondere bei Verwendung von naiven Bibliotheken ein zusätzlicher Affinitätsreifungsschritt in vitro unter Einsatz der zufälligen Mutagenese durchgeführt.[9]
Nach Identifizierung der potentesten Klone wird deren DNA-Sequenz optimiert, um beispielsweise deren enzymatische Stabilität zu erhöhen. Eine Humanisierung, die auf Grund der Homologie zwischen VHH-Fragmenten der Kamele und VH-Fragmenten des Menschen als unproblematisch gilt, soll zudem die Wahrscheinlichkeit von Immunreaktionen gegen Einzeldomänenantikörper bei Verwendung im menschlichen Körper reduzieren.[9] Die Produktion der so optimierten Einzeldomänenantikörper erfolgt schließlich in E. coli, Saccharomyces cerevisiae oder anderen geeigneten Organismen.
Einzeldomänenantikörper aus konventionellen Antikörpern
Alternativ können Einzeldomänenantikörper von konventionellen, aus vier Aminosäureketten bestehenden IgG-Antikörpern der Maus oder des Menschen abgeleitet werden.[10] Für ihre Generierung können ebenfalls Display-Techniken unter Verwendung immuner oder naiver Bibliotheken verwendet werden. Die Generierung von Einzeldomänenantikörpern aus konventionellen Antikörpern ist im Vergleich zu der aus Schwerkettenantikörpern aufwendiger, da die variablen Domänen konventioneller Antikörper als Dimere (VH-VL-Fragmente) vorliegen. Da monomere variable Domänen der Maus oder des Menschen auf Grund ihrer Lipophilie zur Dimerisation oder Aggregation tendieren, werden sie üblicherweise durch Austausch lipophiler gegen hydrophiler Aminosäuren monomerisiert. Häufig ist jedoch auch ein Verlust an Affinität nach Trennung der VH- von den VL-Fragmenten zu beobachten.[11] Sind diese Schwierigkeiten überwunden, können Einzeldomänenantikörper aus konventionellen Antikörpern ebenfalls unter Verwendung der üblichen Produktionsorganismen, wie E. coli oder S. cerevisiae, gewonnen werden.
Von solchen Einzeldomänenantikörpern zu unterscheiden sind scFv-Fragmente (engl.single-chain variable fragments), die zwar auch aus einer einzelnen Aminosäurekette bestehen, die aber zwei bindende Domänen (VH und VL) enthält.
Geschichte
Die Geschichte der Entwicklung von Einzeldomänenantikörpern geht bis in das Jahr 1989 zurück. Damals untersuchten Biologen der Forschergruppe um Raymond Hamers von der Vrije Universiteit Brussel das Immunsystem von Dromedaren. Zu ihrer Überraschung identifizierten sie nicht nur klassische, aus je zwei schweren und zwei leichten Ketten bestehende Antikörper, sondern auch einfacher gebaute Antikörper, die nur aus den schweren Ketten bestanden. Diese Entdeckung wurde 1993 in der Fachzeitschrift Nature veröffentlicht.[12] 1995 folgte dann die Entdeckung von ebenfalls nur aus schweren Ketten bestehenden Antikörpern in Knorpelfischen.[13] Die aus diesen Schwerkettenantikörpern gewonnenen Einzeldomänenantikörper konnten mit Hilfe der Polymerasekettenreaktion in den darauffolgenden Jahren isoliert werden.[8][14]
Einzelnachweise
- Ablynx - Nanobodies in der Arzneimittelentwicklung (Memento vom 28. Februar 2015 im Internet Archive)
- van der Linden RH, Frenken LG, de Geus B, et al.: Comparison of physical chemical properties of llama VHH antibody fragments and mouse monoclonal antibodies. In: Biochim. Biophys. Acta. 1431, Nr. 1, April 1999, S. 37–46. PMID 10209277.
- Harmsen MM, van Solt CB, Hoogendoorn A, van Zijderveld FG, Niewold TA, van der Meulen J: Escherichia coli F4 fimbriae specific llama single-domain antibody fragments effectively inhibit bacterial adhesion in vitro but poorly protect against diarrhoea. In: Vet. Microbiol.. 111, Nr. 1–2, November 2005, S. 89–98. doi:10.1016/j.vetmic.2005.09.005. PMID 16221532.
- Harmsen MM, van Solt CB, van Zijderveld-van Bemmel AM, Niewold TA, van Zijderveld FG: Selection and optimization of proteolytically stable llama single-domain antibody fragments for oral immunotherapy. In: Appl. Microbiol. Biotechnol.. 72, Nr. 3, September 2006, S. 544–51. doi:10.1007/s00253-005-0300-7. PMID 16450109.
- Dolk E, van der Vaart M, Lutje Hulsik D, et al.: Isolation of llama antibody fragments for prevention of dandruff by phage display in shampoo. In: Appl. Environ. Microbiol.. 71, Nr. 1, Januar 2005, S. 442–50. doi:10.1128/AEM.71.1.442-450.2005. PMID 15640220. PMC 544197 (freier Volltext).
- Harmsen MM, De Haard HJ: Properties, production, and applications of camelid single-domain antibody fragments. In: Appl. Microbiol. Biotechnol.. 77, Nr. 1, November 2007, S. 13–22. doi:10.1007/s00253-007-1142-2. PMID 17704915. PMC 2039825 (freier Volltext).
- Jähnichen S, Blanchetot C, Maussang D, et al.: CXCR4 nanobodies (VHH-based single variable domains) potently inhibit chemotaxis and HIV-1 replication and mobilize stem cells. In: Proc. Natl. Acad. Sci. USA. 107, Nr. 47, November 2010, S. 20565–70. doi:10.1073/pnas.1012865107. PMID 21059953. PMC 2996674 (freier Volltext).
- Ghahroudi MA, Desmyter A, Wyns L, Hamers R, Muyldermans S: Selection and identification of single domain antibody fragments from camel heavy-chain antibodies. In: FEBS Lett.. 414, Nr. 3, September 1997, S. 521–6. PMID 9323027.
- Saerens D, Ghassabeh GH, Muyldermans S: Single-domain antibodies as building blocks for novel therapeutics. In: Curr Opin Pharmacol. 8, Nr. 5, Oktober 2008, S. 600–8. doi:10.1016/j.coph.2008.07.006. PMID 18691671.
- Holt LJ, Herring C, Jespers LS, Woolven BP, Tomlinson IM: Domain antibodies: proteins for therapy. In: Trends Biotechnol.. 21, Nr. 11, November 2003, S. 484–90. PMID 14573361.
- Borrebaeck CA, Ohlin M: Antibody evolution beyond Nature. In: Nat. Biotechnol.. 20, Nr. 12, Dezember 2002, S. 1189–90. doi:10.1038/nbt1202-1189. PMID 12454662.
- Hamers-Casterman C, Atarhouch T, Muyldermans S, et al.: Naturally occurring antibodies devoid of light chains. In: Nature. 363, Nr. 6428, Juni 1993, S. 446–8. doi:10.1038/363446a0. PMID 8502296.
- Greenberg AS, Avila D, Hughes M, Hughes A, McKinney EC, Flajnik MF: A new antigen receptor gene family that undergoes rearrangement and extensive somatic diversification in sharks. In: Nature. 374, Nr. 6518, März 1995, S. 168–73. doi:10.1038/374168a0. PMID 7877689.
- Muyldermans S, Atarhouch T, Saldanha J, Barbosa JA, Hamers R: Sequence and structure of VH domain from naturally occurring camel heavy chain immunoglobulins lacking light chains. In: Protein Eng.. 7, Nr. 9, September 1994, S. 1129–35. PMID 7831284.