DamID
DamID (englisch DNA adenine methyltransferase identification) ist eine biochemische Methode zur Bestimmung von Protein-DNA-Interaktionen in Eukaryoten.[1][2]
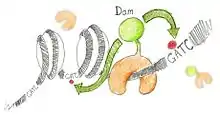
Prinzip
Die DNA-Sequenzen, an die Proteine binden, werden durch ein Fusionsprotein des DNA-bindenden Proteins und einer DNA-Methyltransferase (Dam) nachgewiesen.[3][4] Nach der Bindung des DNA-bindenden Proteins erfolgt eine Methylierung von Adenosin-Resten in benachbarten GATC-Sequenzen durch die angehängte DNA-Methyltransferase zu N6-Methyladeninresten, welche sonst nicht in Eukaryoten vorkommen.[5] Durch die Identifikation der methylierten Adenosine werden die DNA-Sequenzen der Binding bestimmt. Die teilweise methylierte DNA wird mit der Restriktionsendonuklease Dpn I versetzt, welche DNA an methylierten GATC-Sequenzen schneidet. Die Enden der DNA-Fragmente werden mit Oligonukleotiden ligiert. Diese markierte DNA wird in einer Polymerasekettenreaktion mit Primern vervielfältigt, deren Sequenzen jeweils komplementär zu den angefügten Oligonukleotiden sind. Dadurch werden nur Sequenzen vervielfältigt, die an eine DNA-Methylierung angrenzen.
Alternative Methoden sind z. B. EMSA, ChIP, ChIP-on-Chip oder ChIP-Seq. Im Vergleich zu den ChIP-Methoden wird kein Antikörper benötigt, jedoch kann eine Beteiligung posttranslationaler Modifikationen an der DNA-Bindung durch das Protein nicht überprüft werden. Da weiterhin nur DNA-Sequenzen nachgewiesen werden, bei denen die Dam war, und keine Protein-DNA-Interaktion direkt nachgewiesen wird, entstehen fehlerhafte Ergebnisse mit DNA-Klammer-enthaltenden Proteinen wie die RNA-Polymerase, welche an der DNA keine feste Position aufweisen, sondern entlang der DNA gleiten. Es werden aufgrund der PCR nur DNA-Sequenzen zwischen zwei GATC-Sequenzen vervielfältigt. Der mittlere Abstand zwischen zwei aufeinander folgenden GATC-Sequenzen ist etwa 205 Basen in Drosophila (FlyBase release 5), 260 in Mäusen (Mm9) und 264 in Menschen (HG19). Transfizierte Plasmide sollten in Dam-negativen E. coli-Stämmen produziert werden, da bakteriell Dam-methylierte Sequenzen zu falsch positiven Ergebnissen führen.
Weblinks
- Frequently asked questions about DamID (Memento vom 12. September 2018 im Internet Archive)
Einzelnachweise
- van Steensel B, Henikoff S: Identification of in vivo DNA targets of chromatin proteins using tethered dam methyltransferase. In: Nat. Biotechnol.. 18, Nr. 4, April 2000, S. 424–8. doi:10.1038/74487. PMID 10748524.
- Germann S, Juul-Jensen T, Letarnec B, Gaudin V: DamID, a new tool for studying plant chromatin profiling in vivo, and its use to identify putative LHP1 target loci. In: Plant J.. 48, Nr. 1, Oktober 2006, S. 153–63. doi:10.1111/j.1365-313X.2006.02859.x. PMID 16972870.
- Vogel MJ, Peric-Hupkes D, van Steensel B: Detection of in vivo protein-DNA interactions using DamID in mammalian cells. In: Nat Protoc. 2, Nr. 6, 2007, S. 1467–78. doi:10.1038/nprot.2007.148. PMID 17545983.
- Greil F, Moorman C, van Steensel B: DamID: mapping of in vivo protein-genome interactions using tethered DNA adenine methyltransferase. In: Meth. Enzymol.. 410, 2006, S. 342–59. doi:10.1016/S0076-6879(06)10016-6. PMID 16938559.
- Brooks J. E., Roberts R. J.: Modification profiles of bacterial genomes. In: Nucleic Acids Res.. 10, Nr. 3, 1982, S. 913–34. doi:10.1093/nar/10.3.913. PMID 6278441. PMC 326211 (freier Volltext).