Hypervariable Region
Eine hypervariable Region (HVR) ist ein Abschnitt einer DNA oder RNA, der überdurchschnittlich viele Polymorphismen enthält und daher besonders variabel ist. Hypervariable Regionen kommen sowohl in der DNA des Zellkerns als auch in der Kontrollregion der mitochondrialen DNA und in der RNA des Hepatitis-C-Virus vor.
Hypervariable Region der mtDNA
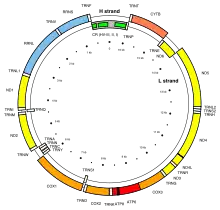
In der mitochondrialen DNA gibt es zwei hypervariable Regionen, HVR1 und HVR2. Während HVR1 zusammenhängend an der Position 16024-16569 liegt, besteht HVR2 aus zwei Abschnitten, die als HVR-II an der Position 57-372 und HVR-III an der Position 438-574 untergliedert werden.[1][2] Aufgrund der hohen Variabilität werden HVR1 und HVR2 zur DNA-Analyse und zur Bestimmung der Haplogruppe verwendet.
In manchen Knochenfischen wie Protacanthopterygii und Gadidae verändert sich die mtDNA-Kontrollregion evolutionär vergleichsweise langsam, sogar langsamer als die mitochondrialen Gene. Der Grund dafür ist noch unbekannt.[3]
Hypervariable Regionen der DNA des Zellkerns
In der DNA des Zellkerns kommen hypervariable Regionen im Abschnitt der Antigen-bindenden Region (complementarity-determining region, CDR) von Genen von Antikörpern und des T-Zell-Rezeptors vor. Sie entstehen in Antikörpergenen sowohl in der CDR der leichten als auch in der CDR der schweren Kette der Antikörper durch VDJ-Rekombination und somatische Hypermutation und dienen der Anpassungsfähigkeit von Antikörpern an neue oder veränderte Antigene im Zuge einer Immunreaktion.[4]
Rab-Proteine besitzen an ihren C-Termini jeweils eine hypervariable Region, welche die jeweilige Lokalisation innerhalb einer Zelle bestimmt.[5] Analoge Funktion hat die hypervariable Region des Ras-Proteins KRAS-4B[6] und bei kleinen GTPasen wie Rac.[7]
Hypervariable Region der RNA des Hepatitis-C-Virus
Das Gen des Hüllproteins 2 des Hepatitis-C-Virus besitzt N-terminal eine hypervariable Region, die als Antigendrift der Immunevasion vor Antikörpern im Rahmen einer Immunantwort gegen das Hepatitis-C-Virus dient.[8][9]
Einzelnachweise
- M. van Oven, M. Kayser: Updated comprehensive phylogenetic tree of global human mitochondrial DNA variation. In: Human Mutation. 30, Nr. 2, Februar 2009, S. E386–94. doi:10.1002/humu.20921. PMID 18853457.
- PhyloTree mt. "Annotated mtDNA reference sequences: revised Cambridge Reference Sequence (rCRS)". Abgerufen am 4. Februar 2016.
- Hirohiko Takeshima, Kei-ichiro Iguchi, Mutsumi Nishida: Unexpected Ceiling of Genetic Differentiation in the Control Region of the Mitochondrial DNA between Different Subspecies of the Ayu, Plecoglossus altivelis. In: Zool. Sci. (2005), Band 22, Heft 4, S. 401–410. doi:10.2108/zsj.22.401.
- Michael Stein, Paul Zei, Gloria Hwang, Radhika Breaden: Cracking The Boards: USMLE Step 1. The Princeton Review, 2000, ISBN 9780375761638 (Abgerufen am 5. September 2011): „Antibodies are remarkably specific, thanks to hypervariable regions in both light and heavy chains.“
- P. Chavrier, J. P. Gorvel, E. Stelzer, K. Simons, J. Gruenberg, M. Zerial: Hypervariable C-terminal domain of rab proteins acts as a targeting signal. In: Nature. Band 353, Nummer 6346, Oktober 1991, S. 769–772, doi:10.1038/353769a0, PMID 1944536.
- A. Banerjee, H. Jang, R. Nussinov, V. Gaponenko: The disordered hypervariable region and the folded catalytic domain of oncogenic K-Ras4B partner in phospholipid binding. In: Current opinion in structural biology. Band 36, Februar 2016, S. 10–17, doi:10.1016/j.sbi.2015.11.010, PMID 26709496, PMC 4785042 (freier Volltext).
- B. D. Lam, P. L. Hordijk: The Rac1 hypervariable region in targeting and signaling: a tail of many stories. In: Small GTPases. Band 4, Nummer 2, 2013 Apr-Jun, S. 78–89, doi:10.4161/sgtp.23310, PMID 23354415, PMC 3747260 (freier Volltext).
- J. Prentoe, J. Bukh: Hypervariable Region 1 in Envelope Protein 2 of Hepatitis C Virus: A Linchpin in Neutralizing Antibody Evasion and Viral Entry. In: Frontiers in immunology. Band 9, 2018, S. 2146, doi:10.3389/fimmu.2018.02146, PMID 30319614, PMC 6170631 (freier Volltext).
- M. U. Mondelli, A. Cerino, A. Meola, A. Nicosia: Variability or conservation of hepatitis C virus hypervariable region 1? Implications for immune responses. In: Journal of biosciences. Band 28, Nummer 3, April 2003, S. 305–310, PMID 12734408.