Boiling Springs Lake RNA-DNA Hybrid Virus
„Boiling Springs Lake RNA-DNA Hybrid Virus“ (BSL-RDHV) ist die Bezeichnung einer vorgeschlagenen Spezies (Art) von Viren, deren Genom-Sequenz (Contig) durch Metagenomanalysen von Proben aus dem Boiling Springs Lake im Geothermalgebieten des Lassen-Volcanic-Nationalparks, Nordkalifornien ermittelt wurde.
| „Boiling Springs Lake RNA-DNA Hybrid Virus“ | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Systematik | ||||||||||||
| ||||||||||||
| Taxonomische Merkmale | ||||||||||||
| ||||||||||||
| Wissenschaftlicher Name | ||||||||||||
| „Boiling Springs Lake RNA-DNA Hybrid Virus“ | ||||||||||||
| Kurzbezeichnung | ||||||||||||
| BSL-RDHV | ||||||||||||
| Links | ||||||||||||
|
Entdeckung
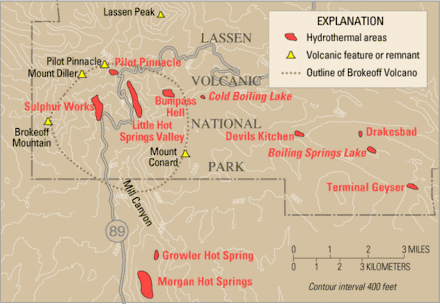
Seit 2001 wurden von Kenneth M. Stedman und Kollegen in den Geothermalgebieten im Lassen-Volcanic-Nationalpark in Nordkalifornien Untersuchungen, insbesondere Metagenomanalysen durchgeführt.[2]
Das wichtigste Ergebnis dieser Metagenomik war die Gensequenz einer Einzelstrang-DNA (ssDNA), in der sich Homologien sowohl zu einem ssDNA-Virus (Rep-Endonuklease, Rep oder REP: hier zu Porcines Circovirus-2, PCV2, Gattung Circovirus, Phylum Cressdnaviricota) als auch zu einem RNA-Virus (Kapsidprotein, Cp, Cap oder CP: zu Familie Tombusviridae) sowie zu den vorgeschlagenen Spezies „Sclerophthora macrospora virus A“, SmV-A[3] und „Plasmopara halstedii virus A“[4] – beide infizieren Eipilze[Anm. 1] – fanden.[2][5]
Diese chimären (oder hybriden) Genomsequenzen (Fachbegriff: Contigs) wurde einem neuen Virus zugeordnet, für dessen Namen nach dem dortigen genauen Fundort Boiling Springs Lake (40,4355° N, 121,3971° W)[6][2] die Bezeichnung „Boiling Springs Lake RNA-DNA Hybrid Virus“ (BSL-RDHV, auch BSL_RDHV) vorgeschlagen wurde (Diemer und Stedman, 2012).[7][2] Das ssDNA-Genom enthält darüber hinaus auch eine konservierte Haarnadelstruktur (en. stem-loop structure),[2] wie sei für Angehörige des Viren-Phylums Cressdnaviricota charakteristisch ist.
Vorgeschlagene RNA-DNA-Hybridisierung
Die folgende Galerie zeigt die der vorgeschlagenen Hybridisierung zugrunde liegenden Ausgangsformen:
- ein ssDNA-Virus der Circoviridae mit zyklischem Genom inkl. Rep und Haarnadelstruktur (stem loop) und kleinem Kapsid, Triangulationszahl T=1
- ein (+)ssRNA-Virus der Tombusviridae mit großem Kapsid, Triangulationszahl T=3
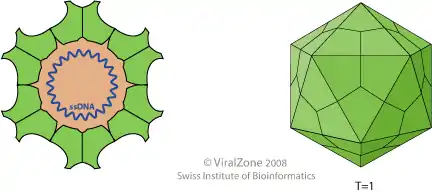 Schema eines Virions der Gattung Circovirus, Querschnitt und Seitenansicht. Zyklisches ssDNA-Genom, Haarnadelstruktur nicht gezeichnet
Schema eines Virions der Gattung Circovirus, Querschnitt und Seitenansicht. Zyklisches ssDNA-Genom, Haarnadelstruktur nicht gezeichnet.png.webp) Genom des „Capybara associated cyclovirus“, Kandidat in der Fam. Circoviridae: Zykl. ssDNA mit stem loop
Genom des „Capybara associated cyclovirus“, Kandidat in der Fam. Circoviridae: Zykl. ssDNA mit stem loop
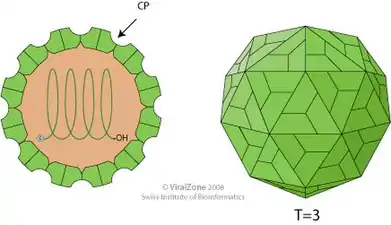 Schemazeichnung eines Virions der Familie Tombusviridae. Querschnitt und Seitenansicht. Lineares (+)ssRNA-Genom.
Schemazeichnung eines Virions der Familie Tombusviridae. Querschnitt und Seitenansicht. Lineares (+)ssRNA-Genom.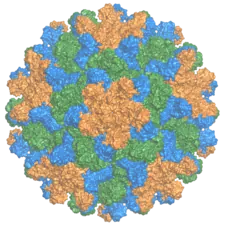 Kapsid des Tomato-bushy-stunt-Virus, Gattung Tombusvirus
Kapsid des Tomato-bushy-stunt-Virus, Gattung Tombusvirus
.png.webp)
Das Ergebnis der vorgeschlagenen Hybridisierung ist ein chimäres Virus mit ssDNA-Genom und Rep mit Haarnadelstruktur, d. es repliziert wie die Circoviridae. Es hat aber das Cap und damit das große Kapsid der Tombusviridae, in dem mehr Platz ist für weitere Genom-Vergrößerungen. Schemazeichnungen dazu finden sich bei de la Higuera und Kollegen (2018)[2] und Richard Harth (2020).[6]
RNA-DNA-Hybridviren (RDHVs)[6][8] dieser Art werden oft auch als „chimäre Viren“ (en. chimeric viruses, CHIVs) bezeichnet.[5] Die Häufigkeit und die Umstände der Rekombination, durch die RNA-DNA-Hybridviren (alias chimäre Viren) entstanden, werden aber noch diskutiert. Zum einen hat es offenbar in der Evolutionsgeschichte der Viren noch weitere RNA-DNA-Hybridisierungsereignisse gegeben.[5][9][10][11] Zum anderen zeigen die Hybridviren mit homologem REP verwirrenderweise eine Reihe unterschiedlicher CAP-Proteine, so dass diskutiert wird, inwieweit diese durch einen weiteren nachträglichen Austausch (innerhalb der Cressdnaviricota) erlangt worden sein könnten.[12]
Für die Klade der RNA-DNA-Hybridviren um BSL-RDHV wurde der Familienname Cruciviridae vorgeschlagen.[2][5] Eine Alternative wäre der ein informelle und nicht-taxonomische Gebrauch der Bezeichnung „Cruciviren“ (englisch Cruciviruses).[13]
Proteom
Vorhergesagte Proteinstrukturen nach Diemer et al. (2012):[7]
.jpg.webp) Vorhergesagte Rep-Struktur von BSL-RDHV
Vorhergesagte Rep-Struktur von BSL-RDHV.png.webp) Anhand der Gensequenz vorhergesagte Struktur des Kapsidproteins Cp von BSL-RDHV im Vergleich zu Tomato-bushy-stunt-Virus (TBSV) und Melon-necrotic-spot-Virus (MNSV), beide Tombusviridae
Anhand der Gensequenz vorhergesagte Struktur des Kapsidproteins Cp von BSL-RDHV im Vergleich zu Tomato-bushy-stunt-Virus (TBSV) und Melon-necrotic-spot-Virus (MNSV), beide Tombusviridae
Weblinks
- Kalia S. I. Bistolas, Ryan M. Besemer, Lars G. Rudstam, Ian Hewson: Distribution and Inferred Evolutionary Characteristics of a Chimeric ssDNA Virus Associated with Intertidal Marine Isopods, in: MDPI Viruses Band 9, Nr 12, 361; Special Issue Viral Recombination: Ecology, Evolution and Pathogenesis; Dezember 2017, Epub 26. November 2017; doi:10.3390/v9120361, PMID 29186875, PMC 5744136 (freier Volltext). Siehe insbes. Fig. 2B: Maximum likelihood phylogeny of Cp
- Caitlin Devor: Scientists discover hybrid virus – Findings point to a virus with genes from RNA and DNA predecessors, in: Journal of Young Investigators (JYI), Allegheny College, 12. Juli 2012
Anmerkungen
- Eipilze sind keine Pilze, sondern Verwandte der Braun- und Goldalgen.
- Das Material wurde von dieser Quelle kopiert, die unter einer Creative Commons Attribution 4.0 International License verfügbar ist.
Einzelnachweise
- ICTV: ICTV Master Species List 2020.v1, New MSL including all taxa updates since the 2019 release, March 2021 (MSL #36)
- Ignacio (Nacho) de la Higuera, Ellis Torrance, Amber Maluenda, George Kasun, Max Larson; Rita Clare: Cruciviruses / RNA-DNA Hybrid Viruses, Extreme Virus Lab (Stedman Lab), 2018. Insbesondere Fig. Cruciviridae
- NCBI: Sclerophthora macrospora virus A (species), (+)ssRNA-Virus
- NCBI: Plasmopara halstedii virus A (species)
- Simon Roux, François Enault, Gisèle Bronner, Daniel Vaulot, Patrick Forterre, Mart Krupovic: Chimeric viruses blur the borders between the major groups of eukaryotic single-stranded DNA viruses, in: Nat Commun 4:2700, 6. November 2013, doi:10.1038/ncomms3700. Siehe insbesondere Supplement 2 (xls).
-
Richard Harth: Criss-crossing viruses give rise to peculiar hybrid variants, Arizona State University, Biodesign Institute, 29. Oktober 2020; sowie
Cruciviruses: Criss-Crossing Viruses Give Rise to Peculiar Hybrid Variants, auf: SciTechDaily vom 2. November 2020. Siehe insbes.
Cruciviruses, alternativ hier: Cruciviruses - Geoffrey S. Diemer, Kenneth M. Stedman: A novel virus genome discovered in an extreme environment suggests recombination between unrelated groups of RNA and DNA viruses, auf: PDXScholar, University Library, Portland State University, 6. Dezember 2012. Ebenso in: Biol Direct. Band 7, Nr. 13, 11. Juni 2012, doi:10.1186/1745-6150-7-13, PMID 22515485, PMC 3372434 (freier Volltext)
- Mart Krupovic, Eugene V. Koonin: Evolution of eukaryotic single-stranded DNA viruses of the Bidnaviridae family from genes of four other groups of widely different viruses, in: Nature Sci Rep Band 4, Nr. 5347, 18. Juni 2014, doi:10.1038/srep05347. Siehe insbes. Fig. 1
- Eugene V. Koonin, Valerian V. Dolja: Expanding networks of RNA virus evolution, In: BMC Biology Band 10, Nr. 54, 20. Juni 2012, doi:10.1186/1741-7007-10-54
- Xiao Yu, Bo Li, Yanping Fu, Daohong Jiang, Said A Ghabrial, Guoqing Li, Youliang Peng, Jiatao Xie, Jiasen Cheng, Junbin Huang, Xianhong Yi: A geminivirus-related DNA mycovirus that confers hypovirulence to a plant pathogenic fungus, in: PNAS USA 107(18). 4. Mai 2010, S. 8387–8392, doi:10.1073/pnas.0913535107, Epub 19. April 2010
- Darius Kazlauskas, Anisha Dayaram, Simona Kraberger, Sharyn Goldstein, Arvind Varsani, Mart Krupovic: Evolutionary history of ssDNA bacilladnaviruses features horizontal acquisition of the capsid gene from ssRNA nodaviruses. In: Virology. 504, 2017, S. 114–121. doi:10.1016/j.virol.2017.02.001. PMID 28189969.
- Ignacio (Nacho) de la Higuera, George W. Kasun, Ellis L. Torrance, Alyssa A. Pratt, Amberlee Maluenda, Jonathan Colombet, Arvind Varsani, Kenneth M. Stedman et al.; Stephen J. Giovannoni (Hrsg.): Unveiling Crucivirus Diversity by Mining Metagenomic Data, in: ASM mBio Band 11, Nr. 5, e01410-20; 1. September 2020; doi:10.1128/mBio.01410-20, PMID 32873755, PMC 7468197 (freier Volltext).
- Marli Vlok: Distribution and Diversity of Aquatic RNA Virus Assemblages in an Environmental Context, Thesis/Dissertation, Faculty of Graduate and Postdoctoral Studies (Botany) an der University of British Columbia (Vancouver), März 2018, 277 Seiten