Langsames Bienenlähmungsvirus
Das Langsame Bienenlähmungsvirus (auch kurz Langsames Lähmungsvirus genannt, wissenschaftlich Slow bee paralysis virus, SBPV oder SPV, auch Honey bee slow paralysis virus) ist eine Spezies (Art) von Viren in der Gattung Iflavirus (der mit Stand Januar 2021 einzigen Gattung in der Familie Iflaviridae).[2][3] Es ist nicht zu verwechseln mit dem Akuten Bienenlähmungsvirus oder dem „Chronischen Bienenlähmungsvirus“. SBPV wurde 1974 in England entdeckt.[4] Es befällt Honigbienen (Apis mellifera), Hummeln (Bombus spp.)[5] und Seidenraupen (Bombyx mori). Übertragen werden die Viren durch die Varroamilbe (Varroa destructor) als Vektor.
| Langsames Bienenlähmungsvirus | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Systematik | ||||||||||||||||||
| ||||||||||||||||||
| Taxonomische Merkmale | ||||||||||||||||||
| ||||||||||||||||||
| Wissenschaftlicher Name | ||||||||||||||||||
| Slow bee paralysis virus | ||||||||||||||||||
| Kurzbezeichnung | ||||||||||||||||||
| SBPV, SBV | ||||||||||||||||||
| Links | ||||||||||||||||||
|
Das Virus verursacht Lähmungen in den vorderen beiden Beinpaaren erwachsener Bienen, was letztlich zu ihrem Tod führt. Infektionen mit Viren aus der Familie Iflaviridae gehören zu den häufigsten Todesursachen von Honigbienenvölkern.[6] Da Bienen und Seidenraupen von großer wirtschaftlicher und biologischer Bedeutung sind, ist das Virus Gegenstand laufender Forschung.
Aufbau
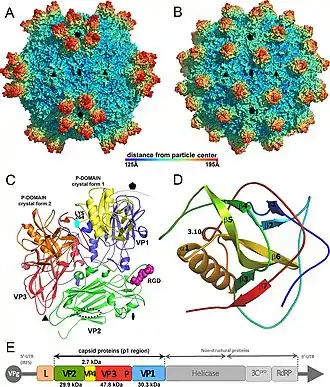
Die Virionen (Virusteilchen) von SBPV haben ein ikosaedrisches Kapsid, das aus 60 Kopien von drei Proteinen besteht, die allen Picornavirales gemeinsam sind.[7] Diese Proteine haben eine Größe von 46, 27 und 29 kDa.[8] Eines dieser drei Proteine (VP3) hat eine C-terminale kugelige Domäne, die sich zu einer einzelnen kugeligen Protein-P-Domäne faltet.[6]
Dieses Protein hat im Kapsid eine hervorstehende Position, so dass es leicht mit den Wirtszellen interagieren kann, wenn das Virusteilchen auf diese trifft. Diese Rezeptoraktivität ist bei vielen Pflanzenviren bekannt, wurde aber bei der Ordnung Picornavirales zuvor noch nicht beobachtet.[7]
Genom
SBPV ist ein unbehülltes Virus. Es hat ein einzelsträngiges RNA-Genom positiver Polarität mit einer Länge von 9470 nt (Nukleotiden).[9][10] Am 5'-Ende hat die nicht-translatierte Region (Leader-Sequenz, 5'-UTR, englisch five prime untranslated region) eine Länge von ca. 300 nt, am 3'-Ende hat die nicht-translatierte Region (3'-UTR) eine Länge von ca. 270 nt, terminiert durch einen Poly(A)-Schwanz.[8] Die kodierende Region der RNA kodiert (u. a.) eine RNA-abhängige RNA-Polymerase. Obwohl sie noch nicht identifiziert wurde, wird erwartet, dass ein virales Genom-gebundenes Protein, das an Stabilität, Replikation und Translation beteiligt ist, an das 5'-Ende gebunden ist.[8]
Obwohl es nicht identifiziert wurde, wird erwartet, dass ein virales Protein (VPg, en. viral genome-linked protein) an das 5'-Ende gebunden ist und so nicht nur für Stabilität sorgt, sondern auch an Replikation und Translation beteiligt ist.[8]
Replikationszyklus
Es gibt derzeit (Stand 10. Dezember 2020) noch wenig gesicherte Erkenntnisse über den Replikationsmechanismus von SBPV. Verschiedene Vermutungen sind diskutiert bei:
Übertragung



SBPV wird, wie etliche andere Bienenviren durch einen weit verbreiteten Parasiten, die Varroamilbe (Varroa destructor), als Vektor übertragen.[12] Dies geschieht, wenn sich die Milben von der Hämolymphe der (adulten) Bienen und Puppen ernähren und diese dabei infizieren.[5] Das Virus reichert sich hauptsächlich im Kopf, in den Speicheldrüsen und im Fettgewebe der Biene an. Weniger betroffen sind die Hinterbeine, der Mitteldarm und das Rektum. Aus diesem Grund kann das Virus auch durch orale Übertragung zwischen Bienen (z. B. von Adulten auf Puppen) verbreitet werden.[13]
Systematik
Es gibt zur Spezies SBPV zwei Virusstämme, Rothamsted und Harpenden, die auf Nukleotidebene zu 83 % und auf Aminosäureebene zu 94 % identisch sind.[8]
Neben SBPV gibt es in der Gattung Iflavirus weitere Bienen parasitierende Virusspezies, das Flügeldeformationsvirus (DWV) inklusive Kakugo-Virus (KV, KaV),[14] das Sackbrut-Virus (SBV), das Varroa destructor virus 1 (VDV1),[15] und — mit Stand Januar 2021 vom ICTV noch unbestätigt – das „Bienen-Iflavirus 1“ (BeeIV-1).[16][17][18]
Symptome
Wie der Name schon andeutet, verursacht das Langsame Bienenlähmungsvirus eine Lähmung, von der zehn bis zwölf Tage nach der Infektion die Vorderbeine betroffen sind.
Bekämpfung
Das Virus hat in weiten Teilen Europas eine geringe natürliche Prävalenz, kann aber in Bienenvölkern mit verschieden starkem Varroa-Befall verbreitet werden.[8]
SBPV kann effektiv durch das Bekämpfung der Varroa-Milbe erreicht werden. Die Bekämpfungs- und Managementstrategien umfassen:[19]
- Mechanische Kontrolle wie z. B. Einstäuben der Bienen (bee dusting)
- Verwendung milbentoleranter Bienenstämme
- Chemische Kontrolle mit biologischen und synthetischen Pestiziden
Einzelnachweise
- ICTV: ICTV Master Species List 2019.v1, New MSL including all taxa updates since the 2018b release, March 2020 (MSL #35)
- Benjamin Dainat, Anton Imdorf, Jean-Daniel Charrière, Peter Neumann: Bienenviren (Teil 2), in: Schweizerische Bienen-Zeitung 5/2008: Forschung, Zentrum für Bienenforschung, Agroscope Liebefeld-Posieux ALP, 3003 Bern (PDF)
- NCBI: Slow bee paralysis virus (species)
- Slow Bee Paralysis virus, Polyprotein gene, Techne qPCR test, Quantification of Slow Bee Paralysis virus genomes, Advanced kit handbook HB10.01.08 (PDF)
- Robyn Manley: Condition-dependent virulence of slow bee paralysis virus in Bombus terrestris: are the impacts of honeybee viruses in wild pollinators underestimated?. In: Oecologia. 184, Nr. 2, 2017, S. 305–315. bibcode:2017Oecol.184..305M. doi:10.1007/s00442-017-3851-2. PMID 28361244. PMC 5487845 (freier Volltext).
- Sergei Kalynych at al.: Virion structure of iflavirus slow bee paralysis virus at 2.6 Å resolution. In: Journal of Virology. 90, Nr. 16, 27. Juli 2016, S. 7444–7455. doi:10.1128/JVI.00680-16. PMID 27279610. PMC 4984619 (freier Volltext).
- Kalynych: Slow Bee Paralysis Virus. In: Protein Data Bank in Europe.
- Joachim R. de Miranda et al.: Genetic characterization of slow bee paralysis virus of the honeybee. In: Journal of General Virology. 91, Nr. Pt 10, 1. Oktober 2010, S. 2524–2530. doi:10.1099/vir.0.022434-0. PMID 20519455.
- V. A. Govan: Analysis of the Complete Genome Sequence of Acute Bee Paralysis Virus Shows That It Belongs to the Novel Group of Insect-Infecting RNA Viruses. In: Virology. 277, Nr. 2, 25. November 2000, S. 457–463. doi:10.1006/viro.2000.0616. PMID 11080493.
- Sergei Kalynych et al.: Cryo-EM study of slow bee paralysis virus at low pH reveals iflavirus genome release mechanism. In: PNAS. 114, Nr. 3, 17. Januar 2017, S. 598–603. doi:10.1073/pnas.1616562114. PMID 28053231. PMC 5255585 (freier Volltext).
- Philip A. Moore, et al.: Honey Bee Viruses, the Deadly Varroa Mite Associates. In: Department of Entomology and Plant Pathology, University of Tennessee, Knoxville TN, USA. 21. August 2014.
- C. H. Denholm: Inducible honeybee viruses associated with varroa jacobsoni. In: PHD Thesis, Keele University, UK. 1999.
- NCBI: Kakugo virus (no rank)
- Eugene V. Ryabov, A. K. Childers, Y. Chen et al.: Recent spread of Varroa destructor virus-1, a honey bee pathogen, in the United States, in: Nature Sci Rep Band 7, Nr. 17447, 12. Dezember 2017, doi:10.1038/s41598-017-17802-3
- NCBI: Bee iflavirus 1 (species)
- Karel Schoonvaere et al.: Study of the Metatranscriptome of Eight Social and Solitary Wild Bee Species Reveals Novel Viruses and Bee Parasites, in: Front. Microbiol., 14. Februar 2018, doi:10.3389/fmicb.2018.00177
- Uta Müller: Sustainable agriculture through protection of wild bee health: Investigation of transmission risk of the honey bee pathogen Nosema ceranae, Dissertation, Department of Biology, Chemistry and Pharmacy, FU Berlin, 2. Mai 2019 (PDF)
- Managing Varroa Mites in Honey Bee Colonies. In: NC State Extension. 23. Februar 2016.