Exosom (Proteinkomplex)
Das Exosom (auch PM/Scl-Komplex) ist ein Proteinkomplex, der beim Abbau von Ribonukleinsäuren (RNA) eine Rolle spielt. Im Kern des Exosoms befinden sich unter anderem sechs charakteristisch ringförmig angeordnete Proteine.[1] Das Exosom ist in der belebten Natur ein sehr weit verbreiteter und ein entwicklungsgeschichtlich alter Proteinkomplex. Er kommt sowohl bei Archäen als auch in den Zellen der Eukaryoten vor. In eukaryotischen Zellen kann der Proteinkomplex sowohl im Zytoplasma als auch im Zellkern gefunden werden und ist essenziell für das Wachstum dieser Zellen. In Bakterien wird seine Funktion durch das Degradosom übernommen.[2]
| Übergeordnet |
| Zelle |
| Untergeordnet |
| Degradosom |
| Gene Ontology |
|---|
| QuickGO |
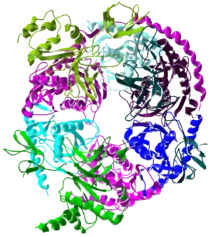
Aufbau
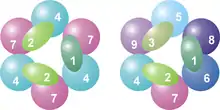
Der Kern des Exosom der Eukaryoten und Archäen besteht, wie auch die strukturell verwandten Polynukleotid-Phosphorylasen (PNPasen) des Degradosoms der Bakterien[2], aus sechs ringförmig angeordneten Kernproteinen, drei Cap-Proteinen und einer zentralen Pore.[1] Häufig treten diese Kern bildenden Proteine in Komplex mit zusätzlichen Proteinen auf.
Alle sechs Kernproteine sind Ribonukleasen (RNasen) der Ribonuklease-PH-Familie. Während der Kern des Exosoms der Archäen aus nur zwei verschiedenen Ribonukleasen (je drei Moleküle Rrp41 und Rrp42) besteht, wird der Kern des Exosoms der Eukaryoten durch sechs verschiedene Ribonukleasen (Rrp41, Rrp45, Rrp42, Rrp43, Mtr3 und Rrp46) aufgebaut. Dabei finden die eukaryotischen Proteine Rrp41, Mtr3 und Rrp46 im Rrp41 der Archäen und die eukaryotischen Proteine Rrp45, Rrp42 und Rrp43 im Rrp42 der Archäen ihre jeweilige Entsprechung.
Die direkt auf den sechs Kern-Ribonukleasen aufliegenden Cap-Proteine besitzen eine S1-RNA-Bindungsdomäne. Bei Archäen werden die Cap-Proteine durch zwei Moleküle Rrp4 und ein Molekül Csl4 repräsentiert. Die Cap-Proteine der Eukaryoten bestehen aus drei verschiedenen Molekülen (Rrp4, Rrp40 und Csl4). Dabei entsprechen sowohl Rrp4 als auch Rrp40 der Eukaryoten dem Rrp4 der Archäen.
Die häufigsten zwei eukaryotischen Proteine, die in fester Gesellschaft mit den Kern bildenden Kern- und Cap-Proteinen auftreten, sind Rrp44 und Rrp6. Während Rrp44 eine Ribonuklease der RNase-R-Familie ist, gehört Rrp6 zur Ribonukleasenfamilie D. Zusätzlich können die Bestandteile des Exosom-Proteinkomplexes mit zahlreichen regulatorischen Proteinen wechselwirken.
Funktion
Das Exosom ist ein an der Spaltung von Ribonukleinsäuren beteiligter Enzymkomplex. Je nach Zellkompartiment und je nach beteiligten regulatorischen Proteinen werden messenger RNA (mRNA), ribosomale RNA (rRNA) oder kleine RNAs als Substrat umgesetzt. Im Zytosol ist das Exosom insbesondere am Abbau der messenger RNA beteiligt. Der Enzymkomplex tritt dabei unter anderem mit Proteinen, welche spezifische Muster (sogenannte AU-reiche Elemente) im 3'-untranslatierten Bereich der messenger RNA erkennen, in Verbindung.[3] Im Zellkern wird der Exosom-Proteinkomplex für die korrekte Prozessierung kleiner RNAs benötigt.[4] Im Nukleolus, dem Zellkernbereich mit der höchsten Exosomendichte, spielt darüber hinaus der Enzymkomplex bei der Prozessierung der ribosomalen RNA eine zentrale Rolle.[5]
Die Funktionen des Exosom-Enzymkomplex beruhen auf seiner Ribonukleaseaktivität. Das Exosom besitzt eine Exoribonukleasefunktion, wodurch das Substrat vom 3'-Ende eines Ribonukleinsäurestrangs abgebaut werden kann. Im Gegensatz zum Exosom der Archäen besitzt das Exosom der Eukaryoten eine zusätzliche Endoribonukleasefunktion, welche eine Spaltung innerhalb eines Ribonukleinsäurestrangs erlaubt. Während die Exoribonukleasefunktion des Exosoms der Archäen von den ringförmig angeordneten Kernproteinen Rrp41 und Rrp42 übernommen wird, sind für die RNA-spaltende Funktion des Exosoms der Eukaryoten die Exosom-assoziierten Proteine Rrp6 und Rrp44 verantwortlich.
Obwohl Eukaryoten über weitere RNA-abbauende Enzyme verfügen, ist das Exosom essenziell für das Überleben der Zellen. Eine experimentelle Hemmung der Exosom-Funktion, beispielsweise mit Hilfe der RNA-Interferenz, führt zu einem Stopp des Zellwachstums oder zum Zelltod.
Literatur
- Schilders G, van Dijk E, Raijmakers R, Pruijn GJ: Cell and molecular biology of the exosome: how to make or break an RNA. In: Int. Rev. Cytol.. 251, 2006, S. 159–208. doi:10.1016/S0074-7696(06)51005-8. PMID 16939780.
- Torben Heick Jensen (Hrsg.): RNA Exosome. Springer, 2010, ISBN 1441978402.
Einzelnachweise
- Lorentzen E, Walter P, Fribourg S, Evguenieva-Hackenberg E, Klug G, Conti E: The archaeal exosome core is a hexameric ring structure with three catalytic subunits. In: Nat. Struct. Mol. Biol.. 12, Nr. 7, Juli 2005, S. 575–81. doi:10.1038/nsmb952. PMID 15951817.
- Lin-Chao S, Chiou NT, Schuster G: The PNPase, exosome and RNA helicases as the building components of evolutionarily-conserved RNA degradation machines. In: Journal of Biomedical Science. 14, Nr. 4, 2007, S. 523–32. doi:10.1007/s11373-007-9178-y. PMID 17514363.
- Chen CY, Gherzi R, Ong SE, et al.: AU binding proteins recruit the exosome to degrade ARE-containing mRNAs. In: Cell. 107, Nr. 4, November 2001, S. 451–64. PMID 11719186.
- Allmang C, Kufel J, Chanfreau G, Mitchell P, Petfalski E, Tollervey D: Functions of the exosome in rRNA, snoRNA and snRNA synthesis. In: EMBO J.. 18, Nr. 19, Oktober 1999, S. 5399–410. doi:10.1093/emboj/18.19.5399. PMID 10508172. PMC 1171609 (freier Volltext).
- Schilders G, Raijmakers R, Raats JM, Pruijn GJ: MPP6 is an exosome-associated RNA-binding protein involved in 5.8S rRNA maturation. In: Nucleic Acids Res.. 33, Nr. 21, 2005, S. 6795–804. doi:10.1093/nar/gki982. PMID 16396833. PMC 1310903 (freier Volltext).