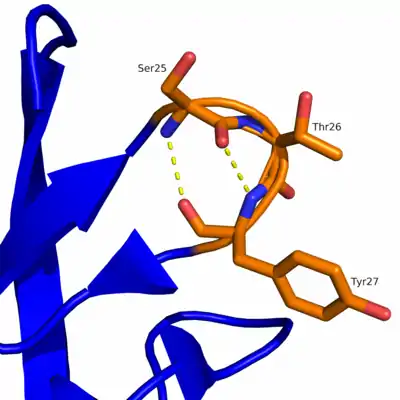
γ-Schleife
Eine γ-Schleife ist ein Sekundärstrukturelement in Proteinen, das den vollständigen Richtungswechsel einer Polypeptidkette erlaubt. Unter den Schleifenstrukturen in Proteinen bezeichnet die γ-Schleife eine Wechselwirkung zwischen einer Aminosäure mit einer zwei Positionen entfernten Aminosäure, d. h. in γ-Position.
Die γ-Schleife ist in der Lage, zwei antiparallele β-Faltblattstrukturen mithilfe von drei Aminosäureresten zu verbinden. Des Weiteren enthält die γ-Schleife zwei Wasserstoffbrückenbindungen, die sich jeweils zwischen dem ersten und dritten Aminosäurerest befinden. Eine inverse γ-Schleife verhält sich spiegelbildlich zur klassischen γ-Schleife.
Struktur der klassischen γ-Schleife
Die γ-Schleife enthält zwei Wasserstoffbrückenbindungen. Dabei wird eine Wasserstoffbrückenbindung zwischen der Carbonylgruppe (–C=O) des ersten Aminosäurerestes (n) und dem Amidproton (–NH) des dritten Aminosäurerestes (n+2) gebildet, was zur Bildung eines siebengliedrigen Ringes führt. Diese Ringstruktur mit Torsionswinkeln von und wurde in mehreren Studien als stabile Konformation vorgeschlagen.[1][2][3] Obwohl diese Wasserstoffbrückenbindung sehr stark gebeugt ist, weist sie eine normale H…O-Länge auf. Dennoch hat sie einen beträchtlichen Einfluss auf die Stabilität des gefalteten, siebengliedrigen Ringes (siehe Tabelle 2). Die Faltung des zweiten Aminosäurerestes erlaubt außerdem die Bildung einer zweiten Wasserstoffbrückenbindung zwischen dem Amidproton des ersten Aminosäurerestes und dem Carbonylsauerstoff des dritten Aminosäurerestes. Diese Bindung ist fast gerade und weist eine optimale Bindungsstärke auf (siehe Tabelle 2), die mit den Wasserstoffbrückenbindungen der antiparallelen β-Faltblattstruktur übereinstimmt.[4]
| Sequenz | Aminosäure- rest | φ in Grad | Ψ in Grad | ω in Grad |
|---|---|---|---|---|
| –L-Ala3– | n | 172 | 128 | −170 |
| n+1 | 68 | −61 | 172 | |
| n+2 | −131 | 162 | — | |
| –Gly3– | n | 171 | 125 | −170 |
| n+1 | 70 | −61 | 172 | |
| n+2 | −129 | 165 | — |
| Eigenschaften | N1H1…O3C3 | N3H3…O1C1 |
|---|---|---|
| H…O-Länge in Å | 1,82 | 1,78 |
| Gesamtenergie in kcal/mol | −5,5 | −4,5 |
| NHO-Winkel in Grad | 166 | 138 |
| HOC-Winkel in Grad | 166 | 102 |
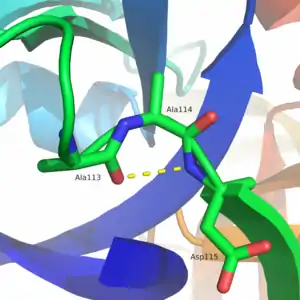
Inverse γ-Schleife
Die inverse γ-Schleife weist verschiedene Torsionswinkel auf und hängt mit der klassischen γ-Schleife dahingehend zusammen, dass durch Vorzeichenumkehr der - und -Winkel die jeweiligen Backbone-Konformationen sich spiegelbildlich zueinander verhalten. Die Mittelwerte für die Torsionswinkel der inversen γ-Schleife liegen bei und .[9] Ein Beispiel für eine inverse γ-Schleife ist im Enzym Proteinase A von S. griseus an den Positionen 113–115 (Ala-Ala-Asp) zu erkennen.[8]
Einzelnachweise
- V. F. Bystrov, S. L. Portnova, V. I. Tsetlin, V. T. Ivanov, Y. A. Ovchinnikov: Conformational studies of peptide systems. The rotational states of the NH–CH fragment of alanine dipeptides by nuclear magnetic resonance. In: Tetrahedron. Band 25, Nummer 3, Februar 1969, S. 493–515, doi:10.1016/s0040-4020(01)83261-0, PMID 5352158.
- G. M. Crippen, H. A. Scheraga: MINIMIZATION OF POLYPEPTIDE ENERGY, VIII. APPLICATION OF THE DEFLATION TECHNIQUE TO A DIPEPTIDE. In: Proceedings of the National Academy of Sciences. Band 64, Nummer 1, 1. September 1969, S. 42, doi:10.1073/pnas.64.1.42.
- Bernard Maigret, Bernard Pullman, David Perahia: Molecular orbital calculations on the conformation of polypeptides and proteins. In: Journal of Theoretical Biology. Band 31, Nummer 2, Mai 1971, S. 269, doi:10.1016/0022-5193(71)90187-1.
- George Némethy, Morton P. Printz: The γ Turn, a Possible Folded Conformation of the Polypeptide Chain. Comparison with the β Turn. In: Macromolecules. Band 5, Nummer 6, 1. November 1972, S. 755, doi:10.1021/ma60030a017.
- M. A. Holmes, B. W. Matthews: Structure of thermolysin refined at 1.6 A resolution. In: Journal of molecular biology. Band 160, Nummer 4, Oktober 1982, S. 623–639, doi:10.1016/0022-2836(82)90319-9, PMID 7175940.
- C. Mueller-Dieckmann, S. Panjikar, P. A. Tucker, M. S. Weiss: On the routine use of soft X-rays in macromolecular crystallography. Part III. The optimal data-collection wavelength. In: Acta crystallographica. Section D, Biological crystallography. Band 61, Nummer 9, September 2005, S. 1263–1272, doi:10.1107/S0907444905021475, PMID 16131760.
- IUPAC-IUB Commission on Biochemical Nomenclature. Abbreviations and symbols for the description of the conformation of polypeptide chains. In: Journal of molecular biology. Band 52, Nummer 1, August 1970, S. 1–17, PMID 5485910.
- M. N. G. James, A. R. Sielecki, G. D. Brayer, L. T. J. Delbaere, C. A. Bauer: Structures of product and inhibitor complexes of Streptomyces griseus protease A at 1.8 Å resolution. In: Journal of Molecular Biology. Band 144, Nummer 1, 25. November 1980, S. 43, doi:10.1016/0022-2836(80)90214-4.
- E. Milner-White, B. M. Ross, R. Ismail, K. Belhadj-Mostefa, R. Poet: One type of gamma-turn, rather than the other gives rise to chain-reversal in proteins. In: Journal of molecular biology. Band 204, Nummer 3, Dezember 1988, S. 777–782, doi:10.1016/0022-2836(88)90368-3, PMID 3225851.