Support Vector Machine
Eine Support Vector Machine [səˈpɔːt ˈvektə məˈʃiːn] (SVM, die Übersetzung aus dem Englischen, „Stützvektormaschine“ oder Stützvektormethode, ist nicht gebräuchlich) dient als Klassifikator (vgl. Klassifizierung) und Regressor (vgl. Regressionsanalyse). Eine Support Vector Machine unterteilt eine Menge von Objekten so in Klassen, dass um die Klassengrenzen herum ein möglichst breiter Bereich frei von Objekten bleibt; sie ist ein sogenannter Large Margin Classifier (dt. „Breiter-Rand-Klassifikator“).
Support Vector Machines sind keine Maschinen im herkömmlichen Sinne, bestehen also nicht aus greifbaren Bauteilen. Es handelt sich um ein rein mathematisches Verfahren der Mustererkennung, das in Computerprogrammen umgesetzt wird. Der Namensteil machine weist dementsprechend nicht auf eine Maschine hin, sondern auf das Herkunftsgebiet der Support Vector Machines, das maschinelle Lernen.
Grundlegende Funktionsweise

Ausgangsbasis für den Bau einer Support Vector Machine ist eine Menge von Trainingsobjekten, für die jeweils bekannt ist, welcher Klasse sie zugehören. Jedes Objekt wird durch einen Vektor in einem Vektorraum repräsentiert. Aufgabe der Support Vector Machine ist es, in diesen Raum eine Hyperebene einzupassen, die als Trennfläche fungiert und die Trainingsobjekte in zwei Klassen teilt. Der Abstand derjenigen Vektoren, die der Hyperebene am nächsten liegen, wird dabei maximiert. Dieser breite, leere Rand soll später dafür sorgen, dass auch Objekte, die nicht genau den Trainingsobjekten entsprechen, möglichst zuverlässig klassifiziert werden.
Beim Einsetzen der Hyperebene ist es nicht notwendig, alle Trainingsvektoren zu beachten. Vektoren, die weiter von der Hyperebene entfernt liegen und gewissermaßen hinter einer Front anderer Vektoren „versteckt“ sind, beeinflussen Lage und Position der Trennebene nicht. Die Hyperebene ist nur von den ihr am nächsten liegenden Vektoren abhängig – und auch nur diese werden benötigt, um die Ebene mathematisch exakt zu beschreiben. Diese nächstliegenden Vektoren werden nach ihrer Funktion Stützvektoren (engl. support vectors) genannt und verhalfen den Support Vector Machines zu ihrem Namen.
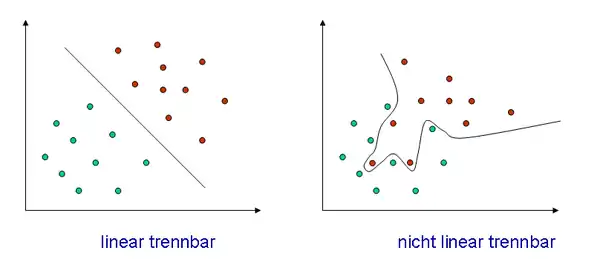
Eine Hyperebene kann nicht „verbogen“ werden, sodass eine saubere Trennung mit einer Hyperebene nur dann möglich ist, wenn die Objekte linear trennbar sind. Diese Bedingung ist für reale Trainingsobjektmengen im Allgemeinen nicht erfüllt. Support Vector Machines verwenden im Fall nichtlinear trennbarer Daten den Kernel-Trick, um eine nichtlineare Klassengrenze einzuziehen.
Die Idee hinter dem Kernel-Trick ist, den Vektorraum und damit auch die darin befindlichen Trainingsvektoren in einen höherdimensionalen Raum zu überführen. In einem Raum mit genügend hoher Dimensionsanzahl – im Zweifelsfall unendlich – wird auch die verschachteltste Vektormenge linear trennbar. In diesem höherdimensionalen Raum wird nun die trennende Hyperebene bestimmt. Bei der Rücktransformation in den niedrigerdimensionalen Raum wird die lineare Hyperebene zu einer nichtlinearen, unter Umständen sogar nicht zusammenhängenden Hyperfläche, welche die Trainingsvektoren sauber in zwei Klassen trennt.
Bei diesem Vorgang stellen sich zwei Probleme: Die Hochtransformation ist enorm rechenlastig und die Darstellung der Trennfläche im niedrigdimensionalen Raum im Allgemeinen unwahrscheinlich komplex und damit praktisch unbrauchbar. An dieser Stelle setzt der Kernel-Trick an. Verwendet man zur Beschreibung der Trennfläche geeignete Kernelfunktionen, die im Hochdimensionalen die Hyperebene beschreiben und trotzdem im Niedrigdimensionalen „gutartig“ bleiben, so ist es möglich, die Hin- und Rücktransformation umzusetzen, ohne sie tatsächlich rechnerisch ausführen zu müssen. Auch hier genügt ein Teil der Vektoren, nämlich wiederum die Stützvektoren, um die Klassengrenze vollständig zu beschreiben.
Sowohl lineare als auch nichtlineare Support Vector Machines lassen sich durch zusätzliche Schlupfvariablen flexibler gestalten. Die Schlupfvariablen erlauben es dem Klassifikator, einzelne Objekte falsch zu klassifizieren, „bestrafen“ aber gleichzeitig jede derartige Fehleinordnung. Auf diese Weise wird zum einen Überanpassung vermieden, zum anderen wird die benötigte Anzahl an Stützvektoren gesenkt.
Mathematische Umsetzung
Die SVM bestimmt anhand einer Menge von Trainingsbeispielen
eine Hyperebene, die beide Klassen möglichst eindeutig voneinander trennt.
Anschaulich bedeutet das Folgendes: Ein Normalenvektor beschreibt eine Gerade durch den Koordinatenursprung. Senkrecht zu dieser Geraden verlaufen Hyperebenen. Jede schneidet die Gerade in einer bestimmten Entfernung vom Ursprung (gemessen in der Gegenrichtung zu ). Diese Entfernung nennt man Bias. Gemeinsam bestimmen der Normalenvektor und der Bias eindeutig eine Hyperebene, und für die zu ihr gehörenden Punkte gilt:
- .
Für Punkte, die nicht auf der Hyperebene liegen, ist der Wert nicht Null, sondern positiv (auf der Seite, zu der zeigt) bzw. negativ (auf der anderen Seite). Durch das Vorzeichen kann man die Seite benennen, auf der der Punkt liegt.
Wenn die Hyperebene die beiden Klassen voneinander trennt, dann ist das Vorzeichen für alle Punkte der einen Klasse positiv und für alle Punkte der anderen Klasse negativ. Ziel ist nun, eine solche Hyperebene zu finden.
Drückt man in den Trainingsbeispielen die Klassenzugehörigkeit durch aus, dann ergibt das eine formelmäßige Bedingung
Wenn überhaupt eine solche Hyperebene existiert, dann gibt es gleich unendlich viele, die sich nur minimal unterscheiden und teilweise sehr dicht an der einen oder anderen Klasse liegen. Dann besteht aber die Gefahr, dass Datenpunkte, denen man zukünftig begegnet, auf der „falschen“ Seite der Hyperebene liegen und somit falsch interpretiert werden.
Die Hyperebene soll daher so liegen, dass der kleinste Abstand der Trainingspunkte zur Hyperebene, das sogenannte Margin (von englisch margin, Marge), maximiert wird, um eine möglichst gute Generalisierbarkeit des Klassifikators zu garantieren.
Das sogenannte Training hat das Ziel, die Parameter und dieser „besten“ Hyperebene zu berechnen.
Die Hyperebene wird dann als Entscheidungsfunktion benutzt. Das heißt: man geht davon aus, dass auch für zukünftige Datenpunkte das berechnete Vorzeichen die Klassenzugehörigkeit richtig wiedergeben wird. Insbesondere kann ein Computer die Klassifikation leicht und automatisch ausführen, indem er einfach das Vorzeichen berechnet.
Linear separierbare Daten
Viele Lernalgorithmen arbeiten mit einer Hyperebene, die im zweidimensionalen durch eine lineare Funktion dargestellt werden kann. Sind zwei Klassen von Beispielen durch eine Hyperebene voneinander trennbar, d. h. linear separierbar, gibt es jedoch in der Regel unendlich viele Hyperebenen, die die beiden Klassen voneinander trennen. Die SVM unterscheidet sich von anderen Lernalgorithmen dadurch, dass sie von allen möglichen trennenden Hyperebenen diejenige mit minimaler quadratischer Norm auswählt, so dass gleichzeitig für jedes Trainingsbeispiel gilt. Dies ist mit der Maximierung des kleinsten Abstands zur Hyperebene (dem Margin) äquivalent. Nach der statistischen Lerntheorie ist die Komplexität der Klasse aller Hyperebenen mit einem bestimmten Margin geringer als die der Klasse aller Hyperebenen mit einem kleineren Margin. Daraus lassen sich obere Schranken für den erwarteten Generalisierungsfehler der SVM ableiten.
Das Optimierungsproblem kann dann geschrieben werden als:
- Minimiere den Term durch Anpassung der Variablen ,
- so dass die Nebenbedingung für alle gilt.
Nicht-linear separierbare Daten
In der Regel sind die Trainingsbeispiele nicht streng linear separierbar. Dies kann u. a. an Messfehlern in den Daten liegen, oder daran, dass die Verteilungen der beiden Klassen natürlicherweise überlappen. Für diesen Fall wird das Optimierungsproblem derart verändert, dass Verletzungen der Nebenbedingungen möglich sind, die Verletzungen aber so klein wie möglich gehalten werden sollen. Zu diesem Zweck wird eine positive Schlupfvariable für jede Nebenbedingung eingeführt, deren Wert gerade die Verletzung der Nebenbedingungen ist. bedeutet also, dass die Nebenbedingung verletzt ist. Da in der Summe die Verletzungen möglichst klein gehalten werden sollen, wird die Summe der Fehler der Zielfunktion hinzugefügt und somit ebenso minimiert. Zusätzlich wird diese Summe mit einer positiven Konstante multipliziert, die den Ausgleich zwischen der Minimierung von und der korrekten Klassifizierung der Trainingsbeispiele regelt. Das Optimierungsproblem besitzt dann folgende Form:
- Minimiere den Term durch Anpassung der Variablen ,
- so dass die Nebenbedingung für alle gilt.
Klassische Lösung: Überführung in ein Duales Problem
Beide Optimierungskriterien sind konvex und können mit modernen Verfahren effizient gelöst werden. Diese einfache Optimierung und die Eigenschaft, dass Support Vector Machines eine Überanpassung an die zum Entwurf des Klassifikators verwendeten Testdaten großteils vermeiden, haben der Methode zu großer Beliebtheit und einem breiten Anwendungsgebiet verholfen.
Das oben beschriebene Optimierungsproblem wird normalerweise in seiner dualen Form gelöst. Diese Formulierung ist äquivalent zu dem primalen Problem, in dem Sinne, dass alle Lösungen des dualen auch Lösungen des primalen Problems sind. Die Umrechnung ergibt sich dadurch, dass der Normalenvektor als Linearkombination aus Trainingsbeispielen geschrieben werden kann:
Die duale Form wird mit Hilfe der Lagrange-Multiplikatoren und den Karush-Kuhn-Tucker-Bedingungen hergeleitet. Sie lautet:
- maximiere für : ,
- so dass die Nebenbedingungen und gelten.
Damit ergibt sich als Klassifikationsregel:
Ihren Namen hat die SVM von einer speziellen Untermenge der Trainingspunkte, deren Lagrangevariablen sind. Diese heißen Support-Vektoren und liegen entweder auf dem Margin (falls ) oder innerhalb des Margin ().
Lösung mittels stochastischem Gradientenabstieg
SVMs können mit stochastischem Gradientenabstieg trainiert werden[1].
Nichtlineare Erweiterung mit Kernelfunktionen
Der oben beschriebene Algorithmus klassifiziert die Daten mit Hilfe einer linearen Funktion. Diese ist jedoch nur optimal, wenn auch das zu Grunde liegende Klassifikationsproblem linear ist. In vielen Anwendungen ist dies aber nicht der Fall. Ein möglicher Ausweg ist, die Daten in einen Raum höherer Dimension abzubilden.
Dabei gilt . Durch diese Abbildung wird die Anzahl möglicher linearer Trennungen erhöht (Theorem von Cover[2]). SVMs zeichnen sich dadurch aus, dass sich diese Erweiterung sehr elegant einbauen lässt. In das dem Algorithmus zu Grunde liegende Optimierungsproblem in der zuletzt dargestellten Formulierung gehen die Datenpunkte nur in Skalarprodukten ein. Daher ist es möglich, das Skalarprodukt im Eingaberaum durch ein Skalarprodukt im zu ersetzen und stattdessen direkt zu berechnen. Die Kosten dieser Berechnung lassen sich sehr stark reduzieren, wenn eine positiv definite Kernelfunktion stattdessen benutzt wird:
Durch dieses Verfahren kann eine Hyperebene (d. h. eine lineare Funktion) in einem hochdimensionalen Raum implizit berechnet werden. Der resultierende Klassifikator hat die Form
mit . Durch die Benutzung von Kernelfunktionen können SVMs auch auf allgemeinen Strukturen wie Graphen oder Strings operieren und sind daher sehr vielseitig einsetzbar. Obwohl durch die Abbildung implizit ein möglicherweise unendlich-dimensionaler Raum benutzt wird, generalisieren SVM immer noch sehr gut. Es lässt sich zeigen, dass für Maximum-Margin-Klassifizierer der erwartete Testfehler beschränkt ist und nicht von der Dimensionalität des Raumes abhängt.
Geschichte
Die Idee der Trennung durch eine Hyperebene hatte bereits 1936 Ronald A. Fisher.[3] Wieder aufgegriffen wurde sie 1958 von Frank Rosenblatt in seinem Beitrag[4] zur Theorie künstlicher neuronaler Netze. Die Idee der Support Vector Machines geht auf die Arbeit von Wladimir Wapnik und Alexei Jakowlewitsch Tscherwonenkis[5] zurück. Auf theoretischer Ebene ist der Algorithmus vom Prinzip der strukturellen Risikominimierung motiviert, welches besagt, dass nicht nur der Trainingsfehler, sondern auch die Komplexität des verwendeten Modells die Generalisierungsfähigkeit eines Klassifizierers bestimmen. In der Mitte der 1990er Jahre gelang den SVMs der Durchbruch, und zahlreiche Weiterentwicklungen und Modifikationen wurden in den letzten Jahren veröffentlicht.
Software
Bibliotheken für SVMs
- libsvm, implementiert in C++ und portiert nach Java
- liblinear, implementiert in C++ und portiert nach Java
- SVMlight, basiert auf C
Software für Maschinelles Lernen und Data-Mining, die SVMs enthalten
- GNU Octave verwendet SVMlight
- KNIME
- Matlab verwendet libsvm, SVMlight und Spider
- RapidMiner verwendet libsvm und eine in Java implementierte SVM
- Scikit-learn verwendet libsvm und liblinear.
- Shogun verwendet libsvm und SVMlight
- WEKA verwendet libsvm und Spider
SVM-Module für Programmiersprachen (Auswahl)
Literatur
- Bernhard Schölkopf, Alex Smola: Learning with Kernels: Support Vector Machines, Regularization, Optimization, and Beyond (Adaptive Computation and Machine Learning), MIT Press, Cambridge, MA, 2002, ISBN 0-262-19475-9.
- Ingo Steinwart, Andreas Christmann: Support Vector Machines, Springer, New York, 2008. ISBN 978-0-387-77241-7. 602 pp.
- Nello Cristianini, John Shawe-Taylor: Kernel Methods for Pattern Analysis, Cambridge University Press, Cambridge, 2004, ISBN 0-521-81397-2
- Christopher J. C. Burges: A Tutorial on Support Vector Machines for Pattern Recognition, Data Mining and Knowledge Discovery, 2(2):121-167, 1998.
- Vladimir Vapnik: Statistical Learning Theory, Wiley, Chichester, GB, 1998.
- Vladimir Vapnik: The Nature of Statistical Learning Theory, Springer Verlag, New York, NY, USA, 1995.
Einzelnachweise
- David Forsyth: Probability and Statistics for Computer Science. Springer, 2017, ISBN 978-3-319-64410-3 (google.com [abgerufen am 21. Oktober 2021]).
- Schölkopf, Smola: Learning with Kernels, MIT Press, 2001
- Fisher, R.A. (1936), "The use of multiple measurements in taxonomic problems", in Annals of Eugenics 7: 179-188, doi:10.1111/j.1469-1809.1936.tb02137.x
- Rosenblatt, F. (1958), "The Perceptron, a Probabilistic Model for Information Storage and Organisation in the Brain", in Psychological Review, 62/386, S. 386-408, doi:10.1037/h0042519.
- Vapnik und Chervonenkis, Theory of Pattern Recognition,1974 (dt. Übersetzung: Wapnik und Tschervonenkis, Theorie der Mustererkennung, 1979)
- David Meyer et al.: R-Paket e1071. Misc Functions of the Department of Statistics, Probability Theory Group (Formerly: E1071), TU Wien. In: CRAN. The R Foundation, abgerufen am 14. Mai 2016 (englisch, aktuelle Version: 1.6-7).
- Uwe Ligges et al.: R-Paket klaR. Classification and visualization. In: CRAN. The R Foundation, abgerufen am 14. Mai 2016 (englisch, aktuelle Version: 0.6-12).
- Alexandros Karatzoglou, Alex Smola, Kurt Hornik: R-Paket kernlab. Kernel-Based Machine Learning Lab. In: CRAN. The R Foundation, abgerufen am 14. Mai 2016 (englisch, aktuelle Version: 0.9-24).