SILAC
SILAC (Abkürzung von stable isotope labeling by/with amino acids in cell culture) ist eine massenspektrometrische Methode zur Mengenbestimmung durch Isotopenmarkierung.[1][2][3][4] SILAC wird in der Proteomik verwendet.
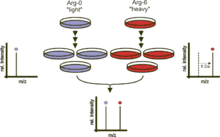
Prinzip
Zwei ursprünglich gleiche Zellkulturen werden mit unterschiedlichen Nährmedien kultiviert. Eine der Zellkulturen erhält im Medium nur Aminosäuren, die ein schweres Isotop tragen. Beispielsweise kann das Medium Arginin mit sechs 13C-Atomen enthalten, anstatt des üblichen 12C. Bei der Proteinbiosynthese werden die Proteine markiert. Dadurch sind in Folge alle Arginin-enthaltenen Peptide sechs Dalton schwerer als die nicht-markierten Peptide der anderen Zellkultur. Alternativ können die beiden Zellkulturen auch mit 13C bzw. 15N markiert werden. Zur Analyse werden die Proteine beider Zellkulturen vereint und gemeinsam vermessen. Anhand der unterschiedlichen Massen können die markierten Peptide identifiziert werden. Die Verhältnisse der Signalintensitäten entsprechen den Mengenverhältnissen der Peptide.[5][6]
Alternativen zur SILAC sind z. B. ICAT, die Isobarenmarkierung, die Tandem Mass Tags (TMT), iTRAQ und die markierungsfreie Quantifizierung (englisch Label-free quantification).
Anwendungen
Ein SILAC-basierter Ansatz wurde zur Untersuchung der Signaltransduktion verwendet, z. B. zur Bestimmung der Substrate von Rezeptortyrosinkinasen,[7] Posttranslationale Modifikationen wie Phosphorylierungen,[7][8] Protein-Protein-Interaktion und zur Untersuchung der Regulation der Genexpression.[9][10]
Pulsed SILAC
Pulsed SILAC (pSILAC) ist eine Variante von SILAC, bei der die markierten Aminosäuren nur eine begrenzte Zeit zu den Zellkulturen gegeben werden, wodurch neu entstandene Proteine gemessen werden und ihre Syntheserate abgeleitet wird.[11]
Literatur
- Ong SE, Kratchmarova I, Mann M: Properties of 13C-substituted arginine in stable isotope labeling by amino acids in cell culture (SILAC). In: Journal of Proteome Research. 2, Nr. 2, 2003, S. 173–81. doi:10.1021/pr0255708. PMID 12716131.
- Ong SE, Mann M: A practical recipe for stable isotope labeling by amino acids in cell culture (SILAC). In: Nature Protocols. 1, Nr. 6, 2006, S. 2650–60. doi:10.1038/nprot.2006.427. PMID 17406521.
- Ong SE, Mann M: Stable isotope labeling by amino acids in cell culture for quantitative proteomics. In: Methods in Molecular Biology. 359, 2007, S. 37–52. doi:10.1007/978-1-59745-255-7_3. PMID 17484109.
Weblinks
Einzelnachweise
- Oda Y, Huang K, Cross FR, Cowburn D, Chait BT: Accurate quantitation of protein expression and site-specific phosphorylation.. In: Proc Natl Acad Sci U S A. 96, Nr. 12, 1999, S. 6591–6. PMID 10359756. PMC 21959 (freier Volltext).
- Jiang H, English, AM: Quantitative Analysis of the Yeast Proteome by Incorporation of Isotopically Labeled Leucine. In: Journal of Proteome Research. 1, Nr. 4, 2002, S. 345–50. doi:10.1021/pr025523f. PMID 12645890.
- Ong SE, Blagoev B, Kratchmarova I, Kristensen DB, Steen H, Pandey A, Mann M: Stable isotope labeling by amino acids in cell culture, SILAC, as a simple and accurate approach to expression proteomics. In: Molecular & Cellular Proteomics. 1, Nr. 5, 2002, S. 376–86. doi:10.1074/mcp.M200025-MCP200. PMID 12118079.
- Zhu H, Pan S, Gu S, Bradbury EM, Chen X: Amino acid residue specific stable isotope labeling for quantitative proteomics. In: Rapid Communications in Mass Spectrometry. 16, Nr. 22, 2002, S. 2115–23. doi:10.1002/rcm.831. PMID 12415544.
- Amanchy R, Kalume DE, Pandey A: Stable isotope labeling with amino acids in cell culture (SILAC) for studying dynamics of protein abundance and posttranslational modifications. In: Science's STKE. 2005, Nr. 16, 2005, S. pl2. doi:10.1126/stke.2672005pl2. PMID 15657263.
- Harsha HC, Molina H, Pandey A: Quantitative proteomics using stable isotope labeling with amino acids in cell culture. In: Nature Protocols. 3, Nr. 3, 2008, S. 505–16. doi:10.1038/nprot.2008.2. PMID 18323819.
- Ibarrola N, Molina H, Iwahori A, Pandey A: A novel proteomic approach for specific identification of tyrosine kinase substrates using 13C tyrosine. In: The Journal of Biological Chemistry. 279, Nr. 16, 2004, S. 15805–13. doi:10.1074/jbc.M311714200. PMID 14739304.
- Ibarrola N, Kalume DE, Gronborg M, Iwahori A, Pandey A: A Proteomic Approach for Quantitation of Phosphorylation Using Stable Isotope Labeling in Cell Culture. In: Analytical Chemistry. 75, Nr. 22, 2003, S. 6043–49. doi:10.1021/ac034931f. PMID 14615979.
- Yetrib Hathout: Approaches to the study of the cell secretome. In: Expert Review of Proteomics. 4, Nr. 2, 2007, S. 239–48. doi:10.1586/14789450.4.2.239. PMID 17425459.
- Martin Polacek, Jack-Ansgar Bruun, Oddmund Johansen, Inigo Martinez: Differences in the secretome of cartilage explants and cultured chondrocytes unveiled by SILAC technology. In: Journal of Orthopaedic Research. 28, Nr. 8, 2010, S. 1040–9. doi:10.1002/jor.21067. PMID 20108312.
- Björn Schwanhäusser, Manfred Gossen, Gunnar Dittmar, Matthias Selbach: Global analysis of cellular protein translation by pulsed SILAC. In: Proteomics. 9, Nr. 1, Dezember, S. 205–9. doi:10.1002/pmic.200800275. PMID 19053139.