Protein-fragment complementation assay
Ein Protein-fragment Complementation Assay, kurz PCA, ist eine biochemische Methode zur Detektion von in vivo Protein-Protein-Interaktionen.
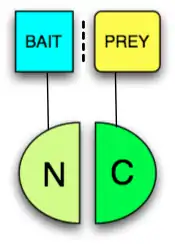
Prinzip
Ein PCA ist eine Methode zum Nachweis einer Bindung durch Komplementation von Fragmenten eines Reporterproteins, das zuvor in zwei inaktive Teile zerlegt wurde. Je ein Teil wird an die beiden zu untersuchenden Proteine als Fusionsprotein angehängt, wodurch bei einer Bindung der beiden Proteine die angehängten Teile rekonstituiert und das Reporterprotein aktiv wird. Ein Fragment wird als Fusionsprotein an ein Köderprotein (engl. bait protein) gekoppelt, das zweite Fragment an ein Beuteprotein (engl. prey protein).
- Bimolekulare Fluoreszenzkomplementation
- Dihydrofolatreduktase (DHFR)[1]
- Beta-Lactamase[2][3]
- Yeast Gal4 (wie im Yeast Two-Hybrid System)
- Split-TEV (Tobacco etch virus-Protease)[4]
- Luciferase[5]
- Ubiquitin[6]
- Grün fluoreszierendes Protein (split-GFP), z. B. EGFP[7][8][9]
- LacZ (beta-Galaktosidase)[10]
Anwendung
Zur in vivo Selektion von Antikörpern[11] wird das Enzym Dihydrofolatreduktase verwendet, welches in zwei Proteinfragmente geteilt wird, die einzeln nicht funktionell sind. Der Genabschnitt, der für einen Teil des Enzyms codiert, wird die genetische Information eines Antigens fusioniert. Dadurch erhält dieser Enzymteil das Antigen als Protein-Tag. Der andere Teil des Enzyms bekommt nun auf ähnliche Weise ein Antikörperfragment, in Form einer Bibliothek angehängt. Bindet nun einer der zufälligen Antikörper an das Antigen, so werden die beiden Hälften des Enzyms wieder nahe genug zusammengebracht, dass sie ein aktives Enzym bilden können. Da es sich bei der Dihydrofolatreduktase um ein Enzym handelt, das die Bakterienzellen zum Überleben benötigen, werden nur diejenigen Zellen überleben, die einen passenden Antikörper zum Antigen besitzen. Diese Zellen werden dann in der Regel vermehrt und der Genabschnitt, der für den Antikörper codiert, sequenziert um den Antikörper näher zu untersuchen.
Einzelnachweise
- K. Tarassov, V. Messier, C. R. Landry, S. Radinovic, M. M. Serna Molina, I. Shames, Y. Malitskaya, J. Vogel, H. Bussey, S. W. Michnick: An in vivo map of the yeast protein interactome. In: Science. Band 320, Nummer 5882, Juni 2008, S. 1465–1470, doi:10.1126/science.1153878. PMID 18467557.
- J. H. Park, J. H. Back, S. H. Hahm, H. Y. Shim, M. J. Park, S. I. Ko, Y. S. Han: Bacterial beta-lactamase fragmentation complementation strategy can be used as a method for identifying interacting protein pairs. In: Journal of microbiology and biotechnology. Band 17, Nummer 10, Oktober 2007, S. 1607–1615, PMID 18156775.
- I. Remy, G. Ghaddar, S. W. Michnick: Using the beta-lactamase protein-fragment complementation assay to probe dynamic protein-protein interactions. In: Nature protocols. Band 2, Nummer 9, 2007, S. 2302–2306, doi:10.1038/nprot.2007.356. PMID 17853887.
- M. C. Wehr, R. Laage, U. Bolz, T. M. Fischer, S. Grünewald, S. Scheek, A. Bach, K. A. Nave, M. J. Rossner: Monitoring regulated protein-protein interactions using split TEV. In: Nature methods. Band 3, Nummer 12, Dezember 2006, S. 985–993, doi:10.1038/nmeth967. PMID 17072307.
- P. Cassonnet, C. Rolloy, G. Neveu, P. O. Vidalain, T. Chantier, J. Pellet, L. Jones, M. Muller, C. Demeret, G. Gaud, F. Vuillier, V. Lotteau, F. Tangy, M. Favre, Y. Jacob: Benchmarking a luciferase complementation assay for detecting protein complexes. In: Nature methods. Band 8, Nummer 12, Dezember 2011, S. 990–992, doi:10.1038/nmeth.1773. PMID 22127214.
- A. Dünkler, J. Müller, N. Johnsson: Detecting protein-protein interactions with the Split-Ubiquitin sensor. In: Methods in molecular biology (Clifton, N.J.). Band 786, 2012, S. 115–130, doi:10.1007/978-1-61779-292-2_7. PMID 21938623.
- E. Barnard, D. J. Timson: Split-EGFP screens for the detection and localisation of protein-protein interactions in living yeast cells. In: Methods in molecular biology (Clifton, N.J.). Band 638, 2010, S. 303–317, doi:10.1007/978-1-60761-611-5_23. PMID 20238279.
- B. D. Blakeley, A. M. Chapman, B. R. McNaughton: Split-superpositive GFP reassembly is a fast, efficient, and robust method for detecting protein-protein interactions in vivo. In: Molecular bioSystems. Band 8, Nummer 8, August 2012, S. 2036–2040, doi:10.1039/c2mb25130b. PMID 22692102.
- S. Cabantous, H. B. Nguyen, J. D. Pedelacq, F. Koraïchi, A. Chaudhary, K. Ganguly, M. A. Lockard, G. Favre, T. C. Terwilliger, G. S. Waldo: A new protein-protein interaction sensor based on tripartite split-GFP association. In: Scientific reports. Band 3, 2013, S. 2854, doi:10.1038/srep02854. PMID 24092409. PMC 3790201 (freier Volltext).
- F. Rossi, C. A. Charlton, H. M. Blau: Monitoring protein-protein interactions in intact eukaryotic cells by beta-galactosidase complementation. In: Proceedings of the National Academy of Sciences. Band 94, Nummer 16, August 1997, S. 8405–8410, PMID 9237989. PMC 22934 (freier Volltext).
- H. Koch, N. Gräfe, R. Schiess & A. Plückthun (2006): Direct Selection of Antibodies from Complex Libraries with the Protein Fragment Complementation Assay. In: J. Mol. Biol. Bd. 357, S. 427–441. PMID 16442560 doi:10.1016/j.jmb.2005.12.043