Complementarity Determining Region
Complementarity-determining regions (CDRs, komplementaritätsbestimmende Regionen) sind Teil der variablen Domänen von Immunglobulinen (Antikörper; hergestellt von B-Zellen) und T-Zell-Rezeptoren (hergestellt von T-Zellen). Jede B- oder T-Zelle produziert eine Sorte Proteinmolekül, das ein spezifisches Antigen, genauer ein spezifisches Epitop, z. B. auf in den Körper eindringenden Viren, Bakterien usw. erkennen kann. Die CDRs sind der variabelste Teil dieser Antikörper und T-Zell-Rezeptoren und somit die eigentliche Quelle der immensen Flexibilität der Immunabwehr.
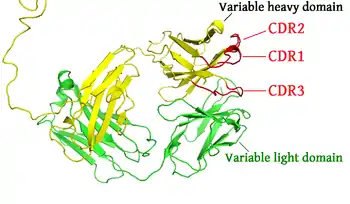
Ein Satz (von sechs) CDRs bildet ein sogenanntes Paratop. Als variabelste Teile der Immunmoleküle sind CDRs unverzichtbar für das Hervorbringen der großen Vielfalt der von Lymphozyten erzeugten Antigen-bindenden Moleküle. Sie werden manchmal auch als hypervariable Regionen bezeichnet.
Lage und Struktur
Die Aminosäuresequenz einer Variabeldomäne eines Antikörpers (oder sonstigen Antigenrezeptors) enthält drei komplementaritätsbestimmende Regionen (CDR1, CDR2 und CDR3). Diese liegen in der Proteinsequenz (der linear vom N- zum C-Terminus, betrachteten Primärstruktur, also Aminosäuresequenz) nicht direkt nebeneinander. Antigenrezeptoren umfassen normalerweise zwei Variabeldomänen (auf zwei verschiedenen Proteinketten; der schweren und der leichten Kette).
Als Ergebnis hat jeder Antigenrezeptor sechs CDRs, die gemeinsam das Antigen binden können. Ein einzelner Antikörper hat zwei Antigenbindungsstellen, die zusammen zwölf CDRs tragen. Räumlich gesehen, gibt es drei CDR-Schleifen pro Variabeldomäne in Antikörpern. (Das pentamere IgM-Molekül hat daher sogar sechzig CDRs.)
Die CDRs werden auch als hypervariable Regionen des Immunglobulins und des T-Zell-Rezeptors bezeichnet, da sie die meiste Sequenzvariation aufweisen.[1] Diese Variation wird durch die Neuordnung (Rearrangement) der beteiligten Gene während der Lymphozytenentwicklung durch V(D)J-Rekombination verursacht.
Auf DNA-Ebene betrachtet, befinden sich CDR1 und CDR2 im Variabel-Segment (V) des Gens für die schwere bzw. leichte Kette. CDR3 umfasst einen Teil des V-Segments, das ganze Diversity-Segment (D, nur für Schwerkette) und das Joining Segment (J).[2] Als Ergebnis ist die Schwerketten-CDR3 ("HCDR3") die variabelste aller komplementaritätsbestimmenden Regionen.
Die (räumliche) Tertiärstruktur ist wichtig für die Analyse und zum Designen neuer Antikörper. Die dreidimensionale Struktur der (Nicht-H3) CDRs in Antikörpern wurde von der Forschergruppe des Cyrus Chothia († 2019) geclustert und klassifiziert,[3] was die gängigen Definitionen der CDR-Grenzen auf Aminosäuresequenz-Ebene nach Elvin A. Kabat verfeinerte. Neuere Bioinformatik-Ansätze zur Homologie-Modellierung ermöglichten das Herleiten der dreidimensionalen Struktur eines Antikörperproteins aus der Aminosäuresequenz. Dies geschieht durch Berechnungen basierend auf den sogenannten H3-Regeln, empirischen Regeln zur Konstruktion von CDR3-Modellen.[4]
Einzelnachweise
- Abbas AK and Lichtman AH: Cellular and Molecular Immunology, 5th. Auflage, Saunders, Philadelphia, 2003, ISBN 0-7216-0008-5.
- William E. Paul: Fundamental Immunology, 6th. Auflage, Lippincott Williams & Wilkins, 2008, ISBN 978-0-7817-6519-0.
- B. Al-Lazikani, A. M. Lesk, C. Chothia: Standard conformations for the canonical structures of immunoglobulins, in: Journal of Molecular Biology 273 1997, S. 927–948, doi:10.1006/jmbi.1997.1354.
- H Shirai, A Kidera, H Nakamura: H3-rules: identification of CDR-H3 structures in antibodies, in: FEBS Letters 455 1999, S. 188–97.