GFP-cDNA
Im Rahmen des GFP-cDNA-Projektes wird die Lokalisation von Proteinen in eukaryotischen Zellen mit Hilfe von Fluoreszenzmikroskopie dokumentiert. Experimentelle Ergebnisse werden durch bioinformatische Analysen ergänzt und im Internet frei zugänglich in einer Datenbank veröffentlicht. Mittels einer Suchfunktion kann in dieser Datenbank nach Proteinnamen und Proteinen mit speziellen Eigenschaften oder Motiven gesucht werden. Das Projekt entsteht in Zusammenarbeit der Forschungsgruppen von Rainer Pepperkok am European Molecular Biology Laboratory (EMBL) und Stefan Wiemann am Deutschen Krebsforschungszentrum (DKFZ).
Welche Experimente werden durchgeführt?
Die cDNA-Produkte neu entdeckter offener Leserahmen (englisch open reading frame) werden mit GFP (Grün Fluoreszierendes Protein) markiert, in eukaryotischen Zellen exprimiert und die Lokalisation mittels Fluoreszenzmikroskopie beobachtet.
Dazu sind folgende Schritte nötig:
Klonierung im Hochdurchsatz
Die Klonierung sehr vieler ORFs wird durch eine Klonierungstechnik ermöglicht, die auf Rekombinationsmechanismen von Bakteriophagen beruht (Gateway von Invitrogen oder Creator von BD Biosciences). Dadurch sind keine Restriktionsenzyme nötig und mögliche Restriktionsstellen müssen nicht berücksichtigt werden. Das ORF wird zunächst in einen Empfängervektor, den sogenannten entry clone, eingebracht. Von diesem Vektor aus können die ORFs mittels Rekombination in andere Plasmide transferiert werden. Für die Analyse der Proteinlokalisation werden die ORFs in GFP-Fusionsvektoren geklont. Dabei wird jedes ORF jeweils einmal C-terminal und N-terminal mit GFP fusioniert. Dies ist notwendig, da die GFP-Markierung Signalsequenzen an den Enden des Proteins maskieren kann.
Transfektion in eukaryotische Zellen, Expression
Die GFP-Fusionsvektoren werden in Vero-Zellen (Nierenfibroblasten der grünen Meerkatze) transfiziert und exprimiert. Besonders interessante ORFs werden auch in PC12-Zellen und Neuronen des Hippocampus eingebracht.
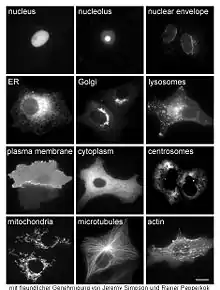
Proteinlokalisierung
Die subzelluläre Lokalisierung des Fusionsproteins wird unter dem Fluoreszenz-Mikroskop zu verschiedenen Zeitpunkten beobachtet. Nach der Lokalisierung in den lebenden Zellen können die Zellen fixiert und zusätzliche Kolokalisationsexperimente mittels Immunfluoreszenzfärbung durchgeführt werden.
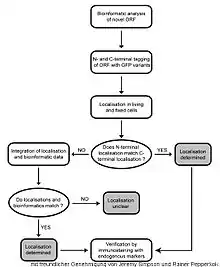
Bioinformatische Analyse
Die Sequenz der eingesetzten cDNA ist bekannt und kann für bioinformatische Analysen verwendet werden. Es werden Vorhersagen zur Lokalisation und Funktion des Proteins gemacht und diese mit den experimentellen Ergebnissen verglichen. Die bioinformatische Analyse wird durch die Bioinformatik-Suchmaschine Harvester erleichtert.
Zuordnung einer subzellulären Lokalisation
Die experimentellen Ergebnisse werden mit der bioinformatischen Analyse verglichen und dem Protein eine subzelluläre Lokalisation zugeordnet (ca. 20 Kategorien). Haben die N-terminale und die C-terminale GFP-Fusion zur selben Lokalisation des Proteins geführt, so hatte die Fusion keinen Einfluss auf die Lokalisation und das Ergebnis wird als bestätigt erachtet. Stimmen die Ergebnisse der beiden Fusionen nicht überein, werden weitere Kriterien wie die bioinformatische Analyse angewandt, um eine Entscheidung treffen zu können. Nicht immer ist eine eindeutige Zuordnung möglich.
Welche Daten werden veröffentlicht?
Jedes Datenblatt enthält die Fluoreszenzbilder der N-terminalen und der C-terminalen Fusion, die Angabe der zugewiesenen Lokalisation, darüber hinaus beobachtete Lokalisationen, Kommentare und die Swissprot ID. Zu jedem Proteineintrag wird ein Link zur entsprechenden Harvester-Seite bereitgestellt.
Wie benutze ich die GFP-cDNA-Datenbank?
Die Bilder aller lokalisierten Proteine und die entsprechende bioinformatische Analyse können über den „Results Table“ oder den „Results Images“ Link von der Startseite aus aufgerufen werden. Mit dem Suchfenster auf der Startseite kann gezielt nach Proteinen gesucht werden. Spezielle Eigenschaften oder Motive sind ebenfalls als Suchbegriffe möglich.