Ashbya gossypii
Ashbya gossypii (auch Eremothecium gossypii) ist ein Schimmelpilz, der mit Bäckerhefe nahe verwandt ist. Er wurde 1926 erstmals von Ashby und Nowell als Pathogen von Baumwollpflanzen beschrieben, bei denen er die so genannte Stigmatomykose verursacht. Die Krankheit betrifft die Entwicklung von Haarzellen in den Samenkapseln von Baumwolle und kann auf Zitrusfrüchte übertragen werden, die daraufhin austrocknen und zusammenfallen (Trockenfäule). A. gossypii ist ein natürlicher Überproduzent von Riboflavin (Vitamin B2), weshalb er auch industriell interessant wurde.[1][2]
| Ashbya gossypii | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|
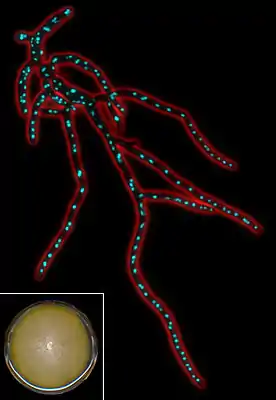
Fluoreszenzmikroskopaufnahme des Mycels von Ashbya gossypii (Zellkerne markiert) | ||||||||||||
| Systematik | ||||||||||||
| ||||||||||||
| Wissenschaftlicher Name der Gattung | ||||||||||||
| Eremothecium | ||||||||||||
| Borzì | ||||||||||||
| Wissenschaftlicher Name der Art | ||||||||||||
| Ashbya gossypii | ||||||||||||
| (S.F. Ashby & W. Nowell) Guillierm |
Beschreibung
Wachstum, Entwicklung und Morphologie
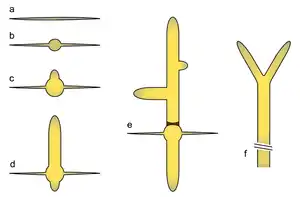
a) Nichtkeimende Spore
b) Isotropische Wachstumsphase in der Samenblase
c) Unipolarer Keimling
d) Zweite Keimröhre erscheint
e) Seitenzweige und Erzeugung des Septum
f) Apikale Verzweigung in reifen Hyphen
Der Lebenszyklus von A. gossypii fängt mit dem nach allen Seiten gleichmäßigen (Isotropie) Wachstum der haploiden Spore, ihrer Keimung an. Darauf folgt apikales Wachstum, das zwei Keimröhren nacheinander auf gegenüberliegenden Seiten der Keimblase hervorbringt. Weitere Achsen werden beim lateralen Abzweigen in jungem Myzel gebildet. Die Reifung ist gekennzeichnet durch apikale Abzweigungen (Spaltung von Spitzen) und einem dramatischen Ansteigen der Wachstumsgeschwindigkeit (bis zu 200 μm/h bei 30 °C), was es A. gossypii ermöglicht, eine Petrischale mit 8 cm Durchmesser in etwa 7 Tagen auszufüllen. Die Sporulation wird wahrscheinlich durch Nahrungsentzug hervorgerufen und führt zu einer Kontraktion des Septum, Zytokinese und nachfolgender Absonderung der Sporangien, die bis zu acht haploide Sporen enthalten können. Die Hyphen selbst sind im Allgemeinen durch Septa unterteilt, die zunächst als Ring erscheinen, der den Transfer von Kernmaterial zulässt; später ändert sich das Aussehen der Septa zu einer geschlossenen Scheibe. Jedes Hyphenkompartiment enthält etwa acht Zellkerne.[3]
Karyotyp und Genom
Die genetische Information von A. gossypii liegt im Zellkern in 7 Autosomen sowie im Kern der Mitochondrien vor. Das Genom des Stamms ATCC 10895 wurde im Jahr 2004 erstmals vollständig analysiert; es besteht aus 9 Millionen Basenpaaren und geschätzten 4.750 Genen. Es ist damit das kleinste bekannte Genom eines frei lebenden Eukaryoten. Neunzig Prozent der Gene in A. gossypii sind sowohl homolog als auch in derselben Reihenfolge (Syntänie) wie in Saccharomyces cerevisiae. Der Vorfahr beider Organismen lebte vor 100 Millionen Jahren. Während A. gossypii sich nur wenig veränderte, ereignete sich bei der folgenden Evolution der Bäckerhefe eine Genduplikation.[4][5][6]
Pathologie
A. gossypii und zwei weitere Pilze, die Stigmatomykose verursachen (Eremothecium coryli, Aureobasidium pullulans), machten es in der ersten Hälfte des 20. Jahrhunderts nahezu unmöglich, Baumwolle in manchen subtropischen Regionen zu kultivieren. Erst die Kontrolle der sporenübertragenden Insektenarten Dysdercus suturellus und Antestia angulosa konnte daraufhin die Infektion der Baumwolle verhindern.[7]
A. gossypii als Modellorganismus
Die erste gezielte Einbringung von DNA (Transformation) als Voraussetzung für weitere molekulargenetische Arbeiten zeigte, dass im Gegensatz zu anderen filamentösen Pilzen ringförmige Plasmide in A.gossypii frei replizieren[8]. Mit Plasmiden, die A.gossypii-DNA enthalten und in dieser Region linearisiert werden, wird homologe Integration erreicht[9]. Aufgrund seines kleinen und außerdem haploiden Genoms und bestehender effizienter Methoden zum Gene-Targeting ist A. gossypii anerkannt als Modellorganismus, insbesondere zur Untersuchung des Wachstums langer und mehrkerniger Pilzzellen. Es wird allgemein angenommen, dass ein besseres Verständnis des Hyphenwachstums die Entwicklung neuartiger Fungizide ermöglichen wird. Besonders vielversprechend ist Ashbya gossypii als Modellorganismus auch aufgrund der hohen Konservierung der Genreihenfolge (Syntänie) zwischen den Genomen von A. gossypii und Saccharomyces cerevisiae.[6]
Vitamin B2 (Riboflavin)
A. gosyypii produziert große Mengen an Riboflavin. A.gossypii diente zur Aufklärung des Riboflavinbiosyntheseweges[10]. Der Organismus wird zur industriellen Produktion von Riboflavin genutzt und wurde dazu weiter optimiert[11][12].
Taxonomische Position
In neueren Werken sind die Gattungen Ashbya, Holleya, und Nematospora synonym zu Eremothecium. Die vier Gattungen zeichnen sich durch nadelförmige Ascosporen aus, die gerade oder gebogen sein können. Die Taxa sind pathogen an verschiedenen Pflanzenarten. Phylogenetische Analysen ribosomaler RNA und DNA in den 1990er Jahren sind sich in der Auffassung einig, dass die vier Arten Kongenere sind.[13][14][15]
Literatur
- P. Philippsen, A. Kaufmann, H. P. Schmitz: Homologues of yeast polarity genes control the development of multinucleated hyphae in Ashbya gossypii. In: Curr Opin Microbiol. Band 8, Nr. 4, 2005, S. 370–377, doi:10.1016/j.mib.2005.06.021, PMID 16023404.
- A. S. Gladfelter: Nuclear anarchy: asynchronous mitosis in multinucleated fungal hyphae. In: Curr Opin Microbiol. Band 9, Nr. 6, 2006, S. 547–552, doi:10.1016/j.mib.2006.09.002, PMID 17045513.
Weblinks
- Cotton Stainer (englisch)
- Antestia
- The Ashbya Genome Database. Abgerufen am 14. Februar 2011.
- Peter Philippsen’s lab at the Biozentrum in Basel, Switzerland. Abgerufen am 14. Februar 2011.
- Hans-Peter Schmitz’ lab at Universität Osnabrück, Germany. (Nicht mehr online verfügbar.) Archiviert vom Original am 22. Februar 2012; abgerufen am 14. Februar 2011.
- Amy Galdfelter’s lab at Dartmouth College, USA. Abgerufen am 14. Februar 2011.
- Jürgen Wendland’s lab at the CRC in Kopenhagen, Denmark. Abgerufen am 14. Februar 2011.
- Fred Dietrich’s lab at Duke University, USA. Abgerufen am 14. Februar 2011.
Einzelnachweise
- S. F. Ashby, W. Nowell: The Fungi of Stigmatomycosis. In: Ann Bot. os-40, Nr. 1, 1926, S. 69–84 (oxfordjournals.org).
- K. P. Stahmann, J. L. Revuelta, H. Seulberger: Three biotechnical processes using Ashbya gossypii, Candida famata, or Bacillus subtilis compete with chemical riboflavin production. In: Appl. Microbiol. Biotechnol. Band 53, Nr. 5, Mai 2000, S. 509–516, PMID 10855708.
- A. Kaufmann: Polarized growth and septation in the filamentous ascomycete "Ashbya gossypii" analyzed by live cell imaging. In: edoc.unibas.ch. Univ. Basel, 13. Februar 2009, abgerufen am 13. Februar 2011 (PhD Thesis, University of Basel, Faculty of Science).
- NCBI-Eintrag Genomprojekt
- Integr8 entry
- F. S. Dietrich, S. Voegeli, S. Brachat, A. Lerch, K. Gates, S. Steiner, C. Mohr, R. Pohlmann, P. Luedi, S. Choi, R. A. Wing, A. Flavier, T. D. Gaffney, P. Philippsen: The Ashbya gossypii genome as a tool for mapping the ancient Saccharomyces cerevisiae genome. In: Science. Band 304, Nr. 5668, 2004, S. 304–307, doi:10.1126/science.1095781, PMID 15001715.
- L. R. Batra: Nematosporaceae (Hemiascomycetidae): taxonomy, pathogenicity, distribution, and vector relations. In: United States. Dept. of Agriculture (Hrsg.): Technical bulletin. Band 1469, 1973.
- M. C. Wright, P. Philippsen: Replicative transformation of the filamentous fungus Ashbya gossypii with plasmids containing Saccharomyces cerevisiae ARS elements. In: Gene. Band 109, Nr. 1, 20. Dezember 1991, ISSN 0378-1119, S. 99–105, doi:10.1016/0378-1119(91)90593-z, PMID 1756987 (nih.gov [abgerufen am 23. Januar 2022]).
- S. Steiner, J. Wendland, M. C. Wright, P. Philippsen: Homologous recombination as the main mechanism for DNA integration and cause of rearrangements in the filamentous ascomycete Ashbya gossypii. In: Genetics. Band 140, Nr. 3, Juli 1995, ISSN 0016-6731, S. 973–987, doi:10.1093/genetics/140.3.973, PMID 7672596, PMC 1206681 (freier Volltext) – (nih.gov [abgerufen am 23. Januar 2022]).
- A. Bacher, S. Eberhardt, M. Fischer, K. Kis, G. Richter: Biosynthesis of Vitamin B 2 (Riboflavin). In: Annual Review of Nutrition. Band 20, Nr. 1, Juli 2000, ISSN 0199-9885, S. 153–167, doi:10.1146/annurev.nutr.20.1.153 (annualreviews.org [abgerufen am 23. Januar 2022]).
- Susanne Katharina Schwechheimer, Enoch Y. Park, José Luis Revuelta, Judith Becker, Christoph Wittmann: Biotechnology of riboflavin. In: Applied Microbiology and Biotechnology. Band 100, Nr. 5, März 2016, ISSN 1432-0614, S. 2107–2119, doi:10.1007/s00253-015-7256-z, PMID 26758294 (nih.gov [abgerufen am 23. Januar 2022]).
- José Luis Revuelta, Rodrigo Ledesma-Amaro, Patricia Lozano-Martinez, David Díaz-Fernández, Rubén M. Buey: Bioproduction of riboflavin: a bright yellow history. In: Journal of Industrial Microbiology & Biotechnology. Band 44, Nr. 4-5, Mai 2017, ISSN 1476-5535, S. 659–665, doi:10.1007/s10295-016-1842-7, PMID 27696023 (nih.gov [abgerufen am 23. Januar 2022]).
- C. P. Kurtzman, J. W. Fell: The yeasts: a taxonomic study. Elsevier, Amsterdam/ New York 2000, ISBN 0-444-81312-8, S. 204–208.
- C. P. Kurtzman: Relationships among the generaAshbya, Eremothecium, Holleya andNematospora determined from rDNA sequence divergence. In: Journal of Industrial Microbiology. Band 14, Nr. 6, 1. Juni 1995, S. 523–530, doi:10.1007/BF01573968, PMID 7662294.
- R. Messner, H. Prillinger, M. Ibl, G. Himmler: Sequences of ribosomal genes and internal transcribed spacers move three plant parasitic fungi, Eremothecium ashbyi, Ashbya gossypii, and Nematospora coryli, towards Saccharomyces cerevisiae. In: J Gen. Appl. Microbiol. Band 41, Nr. 1, 1995, S. 31–42.