Allosterischer Modulator
Als allosterischer Modulator wird in der Biochemie und Pharmakologie eine Substanz bezeichnet, welche die Effekte eines orthosterischen Liganden, z. B. eines Agonisten oder eines inversen Agonisten, an einem Zielprotein, in der Regel einem Rezeptor, verändert (moduliert). Allosterische Modulatoren binden an eine andere (allosterische) Bindungsstelle als die orthosterische Agonistenbindungsstelle. Sie führen zu einer Konformationsänderung des Rezeptor-Proteins, wodurch sich die Rezeptor-Affinität oder die Efficacy des orthosterischen Liganden ändert. Der positive allosterische Modulator (PAM) führt zu einer Verstärkung, der negative allosterische Modulator (NAM) zu einer Abschwächung der Effekte eines Agonisten oder inversen Agonisten, ohne selbst, d. h. in Abwesenheit eines Agonisten oder inversen Agonisten, eine Wirkung zu zeigen. Stoffe, die die allosterische Bindungsstelle besetzen, sich aber funktionell neutral verhalten, werden stille Modulatoren genannt (SAM). Benzodiazepine sind seit langem bekannte Arzneistoffe, welche als allosterische Modulatoren an GABAA-Rezeptoren wirken, indem sie die Affinität des Transmitters GABA erhöhen. Seit dem Jahr 2016 sind Kristallstrukturen von G-Protein-gekoppelten Rezeptoren bekannt, welche mit allosterischen Liganden komplexiert wurden.[1][2][3][4]
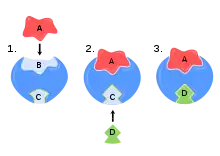
A) positiver allosterischer Modulator
B) allosterisches Zentrum
C) orthosterische Bindungsstelle
D) orthosterischer Ligand
Auch die natürliche Umgebung von Proteinen kann sich auf deren Funktion auswirken. In diesem Sinne können Lipide allosterisch modulierend wirken. Von Bedeutung ist dies hinsichtlich der Beschaffenheit von rezeptorhaltigen Lipidmembranen.[5]
Von den Modulatoren zu unterscheiden sind allosterische Agonisten, welche fähig sind, in Abwesenheit eines orthosterischen Liganden einen Rezeptor über eine Bindung an einer allosterischen Bindungsstelle unmittelbar zu aktivieren. Ferner gibt es ago-allosterische Modulatoren, die sowohl als allosterische Agonisten (Aktivatoren) als auch als allosterische Modulatoren wirken.
In der englischsprachigen Literatur wird mit den Begriffen on-target und off-target allosterism der Ort der Bindung klassifiziert und zeigt an, ob ein Modulator am selben Protein bindet wie der orthosterische Ligand oder ob er an einem Partner-Protein bindet, wie dies bei GPCR-Oligomeren vorkommt.
Literatur
- Christopoulos A, Kenakin T: G protein-coupled receptor allosterism and complexing. In: Pharmacol. Rev.. 54, Nr. 2, Juni 2002, S. 323–74. PMID 12037145.
- Schwartz TW, Holst B: Allosteric enhancers, allosteric agonists and ago-allosteric modulators: where do they bind and how do they act?. In: Trends Pharmacol. Sci.. 28, Nr. 8, 2007, S. 366–373. doi:10.1016/j.tips.2007.06.008. PMID 17629958.
- Wenthur CJ, Gentry PR, Mathews TP, Lindsley CW: Drugs for Allosteric Sites on Receptors. In: Annu. Rev. Pharmacol. Toxicol.. 2013. doi:10.1146/annurev-pharmtox-010611-134525. PMID 24111540.
Einzelnachweise
- Zheng Y, Qin L, Zacarías NV, de Vries H, Han GW, Gustavsson M, Dabros M, Zhao C, Cherney RJ, Carter P, Stamos D, Abagyan R, Cherezov V, Stevens RC, IJzerman AP, Heitman LH, Tebben A, Kufareva I, Handel TM: Structure of CC chemokine receptor 2 with orthosteric and allosteric antagonists. In: Nature. 540, Nr. 7633, 2016, S. 458–461. doi:10.1038/nature20605. PMID 27926736. PMC 5159191 (freier Volltext).
- Oswald C, Rappas M, Kean J, Doré AS, Errey JC, Bennett K, Deflorian F, Christopher JA, Jazayeri A, Mason JS, Congreve M, Cooke RM, Marshall FH: Intracellular allosteric antagonism of the CCR9 receptor. In: Nature. 540, Nr. 7633, 2016, S. 462–465. doi:10.1038/nature20606. PMID 27926729.
- Jazayeri A, Doré AS, Lamb D, Krishnamurthy H, Southall SM, Baig AH, Bortolato A, Koglin M, Robertson NJ, Errey JC, Andrews SP, Teobald I, Brown AJ, Cooke RM, Weir M, Marshall FH: Extra-helical binding site of a glucagon receptor antagonist. In: Nature. 533, Nr. 7602, 2016, S. 274–7. doi:10.1038/nature17414. PMID 27111510.
- Liu X, Ahn S, Kahsai AW, Meng KC, Latorraca NR, Pani B, Venkatakrishnan AJ, Masoudi A, Weis WI, Dror RO, Chen X, Lefkowitz RJ, Kobilka BK: Mechanism of intracellular allosteric β2AR antagonist revealed by X-ray crystal structure. In: Nature. 548, Nr. 7668, 2017, S. 480–484. doi:10.1038/nature23652. PMID 28813418.
- Dawaliby R, Trubbia C, Delporte C, Masureel M, Van Antwerpen P, Kobilka BK, Govaerts C: Allosteric regulation of G protein-coupled receptor activity by phospholipids. In: Nat Chem Biol. 12, Nr. 1, 2016, S. 35–39. doi:10.1038/nchembio.1960.