Lasso-Peptide
Lasso-Peptide sind 16 bis 21 Aminosäure, lange ribosomal synthetisierte und posttranslational modifizierte Peptide mit sehr unterschiedlichen biologischen Funktionen, die jedoch eine strukturelle Gemeinsamkeit haben.
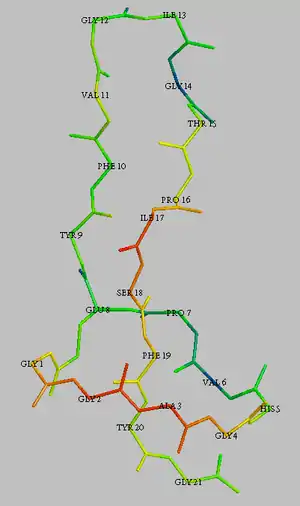
3D-Struktur
Die freie Aminogruppe der ersten Aminosäure (Glycin oder Cystein) formt eine Peptidbindung zu einer Aspartat- oder Glutamatseitenkette in Position 8 oder 9 und erzeugt dadurch einen Lactamring. Der 7 bis 13 Aminosäuren umfassende Schwanz ist durch diesen Ring geführt. Meist ist dieser Schwanz durch große Reste auf beiden Seiten des Rings sterisch fixiert. Diese ungewöhnliche Struktur, die sowohl thermisch als auch gegen Proteolyse sehr stabil ist, gab dieser erst wenige Vertreter umfassenden Peptidgruppe ihren Namen. Die Lasso-Peptide deren 3D-Struktur noch nicht bekannt ist, werden aufgrund der Ähnlichkeiten in der Aminosäuresequenz und ihrer Stabilität zu dieser Gruppe gezählt, auch wenn diese Zuordnung nicht endgültig bestätigt ist.
Struktur der Gencluster
Von einigen Lasso-Peptiden sind die zugehörigen Gencluster sequenziert. Prinzipiell besteht ein solcher Gencluster aus 4 offenen Leserastern. Für den bisher am besten untersuchten Gencluster von Microcin J25 wurden die Gene McjA, McjB, McjC und McjD genannt. Das Gen A codiert für das Vorläuferpeptid, das von den Enzymen B und C zum Lassopeptid prozessiert wird und vom Transporter D exportiert wird. Die Vorläuferpeptide sind 40 bis 60 Aminosäuren lang und tragen die im nativen Lasso-Peptid vorkommende Sequenz am C-Terminus. BLAST-Suchen haben Ähnlichkeiten des Enzyms B mit Transglutaminasen bzw. Proteasen ergeben, daher ist es sehr wahrscheinlich an der Spaltung des Vorläuferpeptids beteiligt. Das Enzym C weist Ähnlichkeiten mit Asparaginsynthetasen der Klasse B und β-Lactam-Synthetasen auf und ist daher wahrscheinlich für die Bildung des Rings verantwortlich. Das Protein D besitzt Homologien mit ABC (ATP-Binding-Casette) Transportern und ist am Export des Lasso-Peptids aus der Zelle beteiligt.
In der folgenden Tabelle sind die bisher bekannten Lasso-Peptide aufgelistet. Die Aminosäuren, die den Lactamring schließen, sind fett, Cysteinreste, die Disulfidbrücken bilden, sind kursiv gedruckt.
| Name | Sequenz | Peptidlänge | 3D-Struktur bekannt | Produzierender Organismus |
|---|---|---|---|---|
| Microcin J25[1] | GGAGHVPEYFVGIGTPISFYG | 21 (1-8) | ja | Escherichia coli |
| Anantin[2] | GFIGWGNDIFGHYSGDF | 17 (1-8) | nein | Streptomyces sp. |
| Capistruin[3] | GTPGFQTPDARVISRFGFN | 19 (1-9) | ja | Burkholderia sp. |
| Lariatin A[4] | GSQLVYREWVGHSNVIKP | 18 (1-8) | ja | Rhodococcus sp. |
| Lariatin B | GSQLVYREWVGHSNVIKGPP | 20 (1-8) | ja | Rhodococcus sp. |
| Propeptin I[5] | GYPWWDYRDLFGGHTFISP | 19 (1-9) | nein | Microbispora sp. |
| Propeptin II | GYPWWDYRDLFGGHTFI | 17 (1-9) | nein | Microbispora sp. |
| RES 701-1[6] | GNWHGTAPDWFFNYYW | 16 (1-9) | nein | Streptomyces sp. |
| RES 701-3[7] | GNWHGTSPDWFFNYYW | 16 (1-9) | nein | Streptomyces sp. |
| Aborycin[8] | CLGIGSCNDFAGCGYAVVCFW | 21 (1-9) | ja | Streptomyces sp. |
| Syamicin I[9] | CLGVGSCNDFAGCGYAIVCFW | 21 (1-9) | nein | Streptomyces sp. |
Quellen
- Craik et al. JACS, 2003, 125 (41): 12464-12474
- Wyss et al. J. antibiotics, 1991, 4 (2): 172-180
- Knappe et al. JACS, 2008, 130 (34): 11446-11454
- Iwatsuki et al. JACS, 2006, 128 (23): 7486-7491
- Kimura et al. J. antibiotics, 1996, 50 (5): 373-378
- Morishita et al. J. antibiotics, 1993, 47 (3): 269-275
- Ogawa et al. J. antibiotics, 1995, 48 (11): 1213-1220
- Helynck et al. J. antibiotics, 1993, 46 (11): 1756-1757
- Yamasaki et al. B.&M. Chem., 1996, 4 (1): 115-120