Copy number variation
Copy number variation (kurz CNV, deutsch Kopienzahlvariation) bezeichnet eine Form struktureller Variation des Erbguts (kurz SV, englisch structural variation), die Abweichungen der Anzahl der Kopien eines bestimmten DNA-Abschnittes innerhalb eines Genoms erzeugt.[1][2] Wurde zunächst angenommen, dass Gene im Genom in der Regel in zwei Kopien vorliegen (je eine Kopie pro Chromosomensatz), so zeigten einige Gene eine Variation der Genkopienzahl zwischen verschiedenen Individuen. So kann ein Gen in nur einer Kopienzahl vorliegen (Gendeletion) oder in mehr als drei oder vier Kopien (Genduplikation). Gene können auch vollständig fehlen (homozygote Gendeletion). Ähnlich wie mit Einzelnukleotid-Polymorphismen (oder SNPs) lassen sich auch aufgrund des Vorkommens bzw. Fehlens von CNVs Individuen voneinander eindeutig unterscheiden. CNVs können Auswirkungen auf die Prädisposition für bestimmte Erkrankungen haben. Verglichen mit SNPs erzeugen CNVs eine größere Anzahl von Erbgut-Unterschieden zwischen Personen, wenn man die Anzahl der durch CNVs betroffenen DNA-Bausteine (Nukleotide) zu Grunde legt.[3]
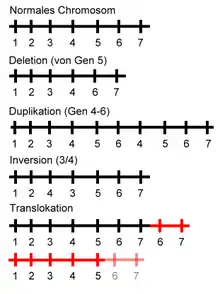
Analysemethoden
Zur CNV-Identifizierung im Genom werden sowohl auf Mikroarrays basierende Techniken[4] als auch immer häufiger die DNA-Sequenzierung, insbesondere Methoden des "Second Generation Sequencing" verwendet. Eine Analyse von Daten einer Pilotstudie des 1000-Genome-Projektes ermöglichte die Identifizierung von 28.000 CNVs in den Genomen von 185 Individuen mit vier verschiedenen DNA-Sequenz basierten Analysemethoden.[2] Diese vier Methoden umfassen die Endpaar-Sequenzierung und deren bioinformatische Analyse (paired-end mapping),[5][6] die direkte Alinierung von DNA-Sequenzen über die Endstellen (oder "Bruchpunkte") von CNVs (split-read analysis),[7][8] die Sequenztiefenanalyse (read-depth analysis)[9][10] und die Verwendung von bioinformatischen Methoden zur Sequenz-Assemblierung (sequence assembly). Im Gegensatz zu Mikroarray-basierten Techniken ermöglicht die DNA-Sequenzierung die Auffindung einer größeren Zahl an Klassen struktureller Variationen, inklusive Inversionen und Translokationen.
Medizinische Bedeutung
Duplikationen und Deletionen können je nach Größe (Länge) des betroffenen DNA-Abschnitts und je nach Gen-Dichte der betroffenen Chromosomenregion sehr unterschiedliche Wirkungen haben. Zunächst können gen-freie Zonen, ein Teil eines Gens, ein komplettes Gen, oder mehrere Gene betroffen sein. Dann hängen die Folgen von der Funktion der betroffenen Gene ab. Hier reicht das Spektrum von tödlich (Fehlgeburt) bis harmlos. Zudem kann – auch innerhalb einer Familie – die Durchschlagskraft (Penetranz und Expressivität) einer solchen genetischen Abweichung von Person zu Person höchst unterschiedlich sein (0–100 %).
Da bei Deletionen ein Teil der Erbinformation nur einfach statt doppelt vorhanden ist oder gar völlig fehlt, ist die Möglichkeit von hierdurch verursachten Störungen einleuchtend. Störungen durch Duplikationen sind schwerer zu erklären. Hier gibt es Beobachtungen, dass bei einer zusätzlichen Kopie eines Gens eine übermäßige Gen-Aktivität (Genexpression) entstehen kann. Zum Beispiel können in einem bestimmten Zeitfenster übermäßig viele Bauelemente für einen Rezeptor hergestellt werden, was zu Stress und Fehlentwicklung von Zellen führen kann.[11][12]
Viele Entwicklungsstörungen, wie etwa Störungen im Autismusspektrum, sind häufig durch Kopienzahlvariationen bedingt – oft durch mehrere gleichzeitig an verschiedenen Orten.[13][14]
Literatur
- M. Saitou, O. Gokcumen: An Evolutionary Perspective on the Impact of Genomic Copy Number Variation on Human Health. In: Journal of molecular evolution. Band 88, Nummer 1, Januar 2020, S. 104–119, doi:10.1007/s00239-019-09911-6, PMID 31522275 (Review).
- V. Salpietro, A. Manole, S. Efthymiou, H. Houlden: A Review of Copy Number Variants in Inherited Neuropathies. In: Current genomics. Band 19, Nummer 6, September 2018, S. 412–419, doi:10.2174/1389202919666180330153316, PMID 30258273, PMC 6128387 (freier Volltext) (Review).
- A. B. Wilfert, A. Sulovari, T. N. Turner, B. P. Coe, E. E. Eichler: Recurrent de novo mutations in neurodevelopmental disorders: properties and clinical implications. In: Genome medicine. Band 9, Nummer 1, 11 2017, S. 101, doi:10.1186/s13073-017-0498-x, PMID 29179772, PMC 5704398 (freier Volltext) (Review).
- S2k-Leitlinie Humangenetische Diagnostik und Genetische Beratung der Deutschen Gesellschaft für Humangenetik. In: AWMF online (Stand 31. Dezember 2018).
Weblinks
- Universitätsklinikum Halle (Saale) - Institut für Humangenetik: Array-CGH-Diagnostik (Methode und Anwendung der Bestimmung von Kopienzahlvariationen, CNV).
- Database of Genomic Variants
- Database of Genomic Structural Variation
- A Deep Catalog of Human Genetic Variation by the 1000 Genomes Project
- Deutsche Gesellschaft für Humangenetik e.V.: Verzeichnis genetischer Beratungsstellen
Einzelnachweise
- Conrad, D.F. et al. (2010). Origins and functional impact of copy number variation in the human genome. In: Nature. Bd. 464, S. 704–712. PMID 19812545
- Mills, R.E. et al. (2011). Mapping copy number variation by population-scale genome sequencing. In: Nature. Bd. 470, S. 59–65. PMID 21293372
- Sudmant, P. et al. (2015). An integrated map of structural variation in 2,504 human genomes In: Nature. Bd. 526, S. 75–81. PMID 26432246
- McCarroll, D.F. et al. (2008). Integrated detection and population-genetic analysis of SNPs and copy number variation. In: Nat Genet. Bd. 40, S. 66–74. PMID 18776908
- Korbel, J.O. et al. (2007). Paired-end mapping reveals extensive structural variation in the human genome. In: Science. Bd. 318, S. 420–426. PMID 17901297
- Kidd, J.M. et al. (2008). Mapping and sequencing of structural variation from eight human genomes. In: Nature. Bd. 453, S. 56–64. PMID 18451855
- Ye, K. et al. (2009): Pindel: a pattern growth approach to detect break points of large deletions and medium sized insertions from paired-end short reads. In: Bioinformatics. Bd. 25, S. 2865–2871. PMID 19561018
- Lam, H.Y. et al. (2010): Nucleotide-resolution analysis of structural variants using BreakSeq and a breakpoint library. In: Nat Biotechnol. Bd. 28, S. 47–55. PMID 20037582.
- Yoon, S. (2009): Sensitive and accurate detection of copy number variants using read depth of coverage. In: Genome Res. Bd. 19, S. 1586–1592. PMID 19657104
- Alkan, C. (2009): Personalized copy number and segmental duplication maps using next-generation sequencing. In: Nat Genet. Bd. 41, S. 1061–1067. PMID 19718026
- M. A. Gillentine, J. Yin, A. Bajic, P. Zhang, S. Cummock, J. J. Kim, C. P. Schaaf: Functional Consequences of CHRNA7 Copy-Number Alterations in Induced Pluripotent Stem Cells and Neural Progenitor Cells. In: American Journal of Human Genetics. Band 101, Nummer 6, Dezember 2017, S. 874–887, doi:10.1016/j.ajhg.2017.09.024, PMID 29129316, PMC 5812918 (freier Volltext).
- M. A. Gillentine, C. P. Schaaf: The human clinical phenotypes of altered CHRNA7 copy number. In: Biochemical pharmacology. Band 97, Nummer 4, Oktober 2015, S. 352–362, doi:10.1016/j.bcp.2015.06.012, PMID 26095975, PMC 4600432 (freier Volltext) (Review).
- G. Ramaswami, D. H. Geschwind: Genetics of autism spectrum disorder. In: Handbook of clinical neurology. Band 147, 2018, S. 321–329, doi:10.1016/B978-0-444-63233-3.00021-X, PMID 29325621 (Review), PDF.
- M. Woodbury-Smith, S. W. Scherer: Progress in the genetics of autism spectrum disorder. In: Developmental medicine and child neurology. Band 60, Nummer 5, 05 2018, S. 445–451, doi:10.1111/dmcn.13717, PMID 29574884 (Review) (freier Volltext).