Alaninscan
Der Alaninscan ist ein Verfahren der Molekularbiologie und Proteinbiochemie zur Identifizierung der für die Funktion, Form und Stabilität von Proteinen oder Peptiden essenziellen molekularen Bestandteile. Es basiert auf dem systematischen sequenziellen Austausch jeder einzelnen Aminosäure oder alternativ dazu mehrerer Aminosäuren eines Peptids, Proteins oder Proteinteils gegen die Aminosäure Alanin. Ein Austausch einer funktionell oder strukturell essenziellen Aminosäure eines Proteins gegen Alanin hat in der Regel einen Verlust der Proteinfunktion zur Folge.
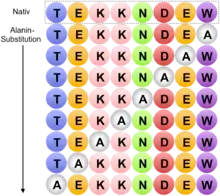
Prinzip
Der Alaninscan basiert auf dem systematischen, sequenziellen Austausch jeder einzelnen Aminosäure gegen Alanin. Diese Substitution hat zur Folge, dass alle betroffenen Seitenkettenatome bis auf eine funktionell weitgehend neutrale Methylgruppe entfernt werden. Theoretisch wäre zwar auch eine vollständige Entfernung der Seitenketten durch einen Austausch mit der Aminosäure Glycin denkbar, doch hat eine solche Substitution dank der konformationellen Flexibilität der Peptidbindung des Glycins einen starken Einfluss auf die Sekundärstruktur des Proteins. Wird eine für die Funktion essenzielle Aminosäure gegen Alanin ausgetauscht, kann in der Regel ein Verlust der Proteinfunktion beobachtet werden. Ebenso kann ein Verlust der Proteinfunktion bei Austausch einer für die innere Struktur oder die Form essenziellen Aminosäure eintreten. Die Methode funktioniert trotzdem, da Alanin die Sekundärstruktur-Präferenzen der Mehrzahl der proteinogenen Aminosäuren nachahmt.[1] Diese Tatsache ist ausführlich durch die Alanin-Welt-Hypothese erklärt.
Methodik
Klassische Methoden des Alaninscans eines Proteins basieren auf dem sequenziellen Austausch jeder einzelnen Aminosäure mit Hilfe der ortsspezifischen Mutagenese. Diese Methode ist sehr aufwendig, da jedes einzelne modifizierte Protein separat konstruiert, exprimiert und analysiert werden muss. Durch Kombination der zufälligen Mutagenese mit einem Displayverfahren, wie dem Phagendisplay, kann der Arbeitsaufwand reduziert werden.[2] Auch kombinatorische Ansätze des Alaninscans sind möglich und erleichtern die Untersuchung der Funktion von Proteinen nach gleichzeitigem Austausch mehrerer Aminosäuren.[3] Ein Alaninscan eines kleinen Peptids ist mit Hilfe der chemischen Synthese möglich.
Literatur
- Morrison KL, Weiss GA: Combinatorial alanine-scanning. In: Curr Opin Chem Biol. 5, Nr. 3, Juni 2001, S. 302–7. PMID 11479122.
Einzelnachweise
- Vladimir Kubyshkin, Nediljko Budisa: The Alanine World Model for the Development of the Amino Acid Repertoire in Protein Biosynthesis. In: Int. J. Mol. Sci.. 20, Nr. 21, 24. September 2019, S. 5507. doi:10.3390/ijms20215507.
- Cain SA, Ratcliffe CF, Williams DM, Harris V, Monk PN: Analysis of receptor/ligand interactions using whole-molecule randomly-mutated ligand libraries. In: J. Immunol. Methods. 245, Nr. 1–2, November 2000, S. 139–45. PMID 11042291.
- Morrison KL, Weiss GA: Combinatorial alanine-scanning. In: Curr Opin Chem Biol. 5, Nr. 3, Juni 2001, S. 302–7. PMID 11479122.