Rekombination (evolutionärer Algorithmus)
Als Rekombination oder Crossover wird bei evolutionären Algorithmen die Erzeugung eines neuen Genoms (auch als Filialgenom bezeichnet) aus (in der Regel) zwei Elterngenomen (Parentalgenomen) bezeichnet. Eine Funktion, die eine zulässige Menge von Parentalgenomen auf eine Menge von Filialgenomen abbildet, heißt Rekombinationsfunktion. Eine Rekombinationsfunktion ist ein genetischer Operator.
In der Literatur ist neben der Rekombination auch häufig von Crossover die Rede und beide Begriffe werden meist synonym verwendet.
Ziel der Rekombination ist es, gute Eigenschaften zweier verschiedener Eltern auf ein Kind zu übertragen. Im Vergleich zu Algorithmen, die nur die Mutation zur Veränderung der Genome benutzen, können so möglicherweise schneller Individuen gefunden werden, die zwei gute Eigenschaften A und B in sich tragen, wenn es vorher nur Individuen gab, die entweder nur über A oder B verfügten. Generell gilt, dass die Erzeugung von Elternklonen aus Effizienzgründen zu vermeiden ist.
Gute Rekombinationsfunktionen zeichnen sich dadurch aus, dass sie zumindest die guten Eigenschaften der Eltern erhalten und nicht so rekombinieren, dass diese Eigenschaften zerstört werden.
Für verschiedene Genom- und Problemtypen eignen sich verschiedene Rekombinationstypen unterschiedlich gut:
Rekombination von binären Zahlen (Bitstrings)
Bei der Rekombination binärer Zahlen werden die Parentalgenome an einer oder mehreren Stellen unterteilt und das Filialgenom aus diesen Teilen zusammengesetzt.
Zu den schon frühzeitig verwendeten Rekombinationsoperatoren gehören das 1-Punkt- und das n-Punkt-Crossover. Bei beiden Operatoren werden Crossoverpunkte zufällig innerhalb des Genoms eines Elters bestimmt, die dann für beide Parentalgenome gelten. Das n-Punkt-Crossover beginnt mit der zufälligen Bestimmung der Anzahl der Crossoverpunkte, deren Anzahl kleiner sein muss als die der Gene des Genoms. Beim 1-Punkt-Crossover gilt . Das Kindgenom wird dadurch gebildet, das abwechselnd die Gene des ersten und des zweiten Parentalgenoms bis zum jeweils nächsten Crossoverpunkt auf das Kindgenom kopiert werden.
Als Beispiel soll ein 2-Punkt-Crossover dienen:
| Verfahren | Beispiel |
|
und |
|
, , |
|
Ein ebenfalls häufig genutzter Operator ist das Uniform Crossover, bei dem für jedes Gen (hier jedes Bit) zufällig entschieden wird, von welchem Parentalgenom es stammen soll.[1]
Je nach Ausgestaltung eines Rekombinationsoperators können auch die bei den vorgestellten drei Operatoren verbleibenden Genomstücke zu einem zweiten Kindgenom zusammengefügt werden. Dann erzeugt der so modifizierte Rekombinationsoperator zwei an Stelle von einem Nachkommen pro Ausführung.
Rekombination von ganzzahligen oder reellwertigen Genomen
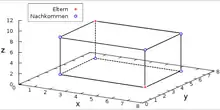
Für die oben vorgestellten und für die meisten anderen Rekombinationsoperatoren für Bitstrings gilt, dass sie auch auf ganzzahlige oder reellwertige Genome, deren Gene aus je einer ganzen oder reellwertigen Zahl bestehen, entsprechend angewandt werden können. Anstelle einzelner Bits werden dann einfach ganze oder reelle Zahlen in das Kindgenom kopiert. Die Nachkommen liegen auf den verbleibenden Ecken des durch die beiden Eltern aufgespannten Hyperkörpers. Nebenstehendes Bild zeigt dies beispielhaft für den dreidimensionalen Fall, bei dem die Nachkommen auf den Ecken des durch die beiden Eltern und aufgespannten Quaders liegen. Wenn bei der Erzeugung des Nachkommen die Regeln des Uniform Crossover für Bitstrings angewandt werden, spricht man auch von diskreter Rekombination.
Intermediäre Rekombination
Bei diesem Rekombinationsoperator werden die Allelwerte des Filialgenoms durch Mischung aus den Allelen der beiden Parentalgenome und erzeugt:
- mit jeweils zufällig gleichverteilt pro Gen
Die Wahl des Intervalls bewirkt die Einbeziehung des Inneren des durch die Allelwerte der Elterngene aufgespannten Hyperkörpers und einer gewissen Umgebung. Für wird ein Wert von empfohlen, um der bei einem Wert von sonst vorhandenen Tendenz zur Verkleinerung der Allelwerte entgegenzuwirken.
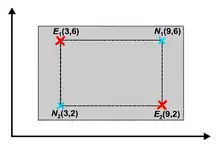
Nebenstehendes Bild zeigt beispielhaft für den zweidimensionalen Fall den grau dargestellten Wertebereich der möglichen neuen Allele der beiden Parentalgenome und bei intermediärer Rekombination. Die möglichen Nachkommen der diskreten Rekombination und sind ebenfalls eingezeichnet. Die intermediäre Rekombination erfüllt die nach der Theorie der virtuellen Alphabete geforderte arithmetische Berechnung der Allelwerte des Filialgenoms. Diskrete und intermediäre Rekombination finden bei der Evolutionsstrategie standardmäßig Verwendung.
Rekombination von Permutationen
Für kombinatorische Aufgabenstellungen werden in der Regel Permutationen verwendet, die speziell für Genome ausgelegt sind, die selbst Permutationen einer Menge sind. Die zu Grunde liegende Menge ist in der Regel eine Teilmenge von oder . Wenn man für solche Genome 1- oder n-Punkt- oder Uniform Crossover für ganzzahlige Genome verwendet, kann es vorkommen, dass das Filialgenom einige Werte doppelt enthält und andere fehlen. Dies kann durch Reparaturmaßnehmen (genetic repair) behoben werden, etwa indem man die überzähligen Gene positionstreu gegen fehlende aus dem anderen Filialgenom austauscht.
Es wurden spezielle Rekombinationsoperatoren für Permutationen entwickelt, die diesen Nachteil nicht haben und stärker an den Erfordernissen der Kombinatorik orientiert sind. Beispielhaft seien drei Operatoren vorgestellt.
Position-based Crossover
Das Position-based Crossover[2] und das nachfolgend vorgestellte Order-based Crossover geben die relative Reihenfolge der Elterngenome an das oder die Kinder weiter. Der Rekombinationsoperator wird anhand eines Beispiels erläutert:
| Verfahren | Beispiel |
|
und |
|
|
|
|
|
|
|
Order-based Crossover
Neben dem feingranularen Position-based Crossover gibt es noch das Order-based Crossover, das in größerem Maße mit zusammenhängenden Teilstücken der Genome arbeitet. Dazu werden Anzahl und Länge der Teilstücke ausgewürfelt und danach mit den entstandenen Gensequenzen ähnlich verfahren, wie zuvor beschrieben, siehe auch:[3]
| Verfahren | Beispiel |
|
und |
|
|
|
|
|
|
|
Das Order-based Crossover ist unter anderem gut für das Scheduling von Workflows geeignet, wenn es in Verbindung mit 1- und n-Punkt-Crossover eingesetzt wird.[4] Workflows bedeuten eine zusätzliche Einschränkung für das Scheduling, da zumindest ein Teil der Einzeloperationen eines zu verplanenden Auftrags in einer vergebenen Reihenfolge auszuführen ist. In diesem Zusammenhang sei angemerkt, dass beide Operatoren nicht garantieren, dass eine Reihenfolgekorrektheit der Eltern weitervererbt wird. Dies ist jedoch kein Nachteil gegenüber anderen Operatoren, welche die Weitervererbung gewährleisten.[4]
Man kann mit den vorgestellten Operatoren auch ein zweites (in gewisser Weise inverses) Kind erzeugen, indem man die Eltern vertauscht und das Verfahren erneut anwendet.
Edge-Rekombination
Eine weitere Variante der Rekombination von Permutationen ist die Edge-Rekombination, bei der die Nachbarschaftsbeziehungen zwischen den Elementen der Elterngenome so gut wie möglich erhalten werden. Bei der Edge-2-Rekombination werden dabei Verbindungen bevorzugt, die in beiden Elterngenomen vorkommen. Die Edge-3- und Edge-4-Rekombination versuchen zusätzlich, durch Inversion der Genome noch zusätzliche Nachbarschaften auszunutzen, die bei der Edge-2-Rekombination verloren gingen. Dieses Verfahren ist besonders gut geeignet für kombinatorische Optimierungsprobleme wie das Traveling Salesman Problem.[5]
Rekombination von Bäumen
Die Rekombination von Bäumen ist speziell für Genome ausgelegt, die selbst Bäume sind.
Ein Beispiel für eine Rekombination von Bäumen ist folgendes Verfahren:
- Gegeben seien zwei Eltern-Bäume (Eltern-Genome).
- Wähle in jedem Eltern-Baum einen Teilbaum aus.
- Vertausche diese zwei Teilbäume.
Die zwei so neu entstandenen Bäume sind nun die zwei Kind-Genome.
Literatur
- Hartmut Pohlheim: Evolutionäre Algorithmen. Verfahren, Operatoren und Hinweise für die Praxis. Springer, Berlin 1999, ISBN 3540664130.
- Karsten Weicker: Evolutionäre Algorithmen. Teubner, Stuttgart 2002, ISBN 3519003627.
- Keshav P. Dahal, Kay Chen Tan, Peter I. Cowling (Hrsg.): Evolutionary Scheduling. Studies in Computational Intelligence, Bd. 49, Springer, Berlin, Heidelberg, 2007. doi: 10.1007/978-3-540-48584-1, ISBN 978-3-642-08017-3
- Amir H. Gandomi, Ali Emrouznejad, Mo M. Jamshidi, Kalyanmoy Deb, Iman Rahimi (Hrsg.): Evolutionary Computation in Scheduling. John Wiley & Sons, 2020. ISBN 978-1-119-57387-6
Einzelnachweise
- Gilbert Syswerda: Uniform Crossover in Genetic Algorithms. In: David Schaffer (Hrsg.): Proc. of Int. Conf. on Genetic Algorithms (3rd ICGA). Morgan Kaufman Publishers Inc., 1989, ISBN 1-55860-066-3, S. 2–9.
- Gilbert Syswerda: Schedule Optimization Using Genetic Algorithms. In: Lawrence Davis (Hrsg.): Handbook of Genetic Algorithms. Van Nostrand Reinhold, New York 1991, ISBN 978-0-442-00173-5, S. 332–349.
- Lawrence Davis: Handbook of Genetic Algorithms. Van Nostrand Reinhold, New York 1991, ISBN 0-442-00173-8.
- Wilfried Jakob, Alexander Quinte, Karl-Uwe Stucky, Wolfgang Süß: Fast Multi-objective Scheduling of Jobs to Constrained Resources Using a Hybrid Evolutionary Algorithm. In: Günter Rudolph (Hrsg.): Parallel Problem Solving from Nature – PPSN X. LNCS, Nr. 5199. Springer, Berlin, Heidelberg 2008, ISBN 978-3-540-87699-1, S. 1031–1040, doi:10.1007/978-3-540-87700-4_102 (springer.com [abgerufen am 13. Februar 2022]).
- Darrell Whitley, Timothy Starkweather, Daniel Shaner: The Traveling Salesman and Sequence Scheduling: Quality Solutions Using Genetic Edge Recombination. In: Lawrence Davis (Hrsg.): Handbook of Genetic Algorithms. Van Nostrand Reinhold, New York 1991 (psu.edu [PDF]).