Ogataea angusta
Ogataea angusta (meist noch unter den Synonymen Pichia angusta oder auch Hansenula polymorpha bekannt) ist eine methylotrophe Hefeart mit ungewöhnlichen Eigenschaften. Sie wird als Proteinfabrik für pharmazeutische Produkte genutzt und gehört zu einer begrenzten Anzahl methylotropher Hefen (Hefen, die auf Methanol, früher auch Methylalkohol genannt, wachsen können).
| Ogataea angusta | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Systematik | ||||||||||||
| ||||||||||||
| Wissenschaftlicher Name | ||||||||||||
| Ogataea angusta | ||||||||||||
| (Teun., H.H. Hall & Wick.) Suh & Zhou |
Grundlagen
Andere Hefearten mit dieser Fähigkeit sind Candida boidinii, Pichia methanolica und Komagataella phaffii. P. angusta gehört taxonomisch zur Familie der Saccharomycetaceae. In neueren Taxonomiebüchern wurden die Genera Pichia and Hansenula miteinander vereint, und H. polymorpha wurde in Pichia angusta umbenannt. Nach dem Wunsch vieler Wissenschaftler soll der populäre Name H. polymorpha jedoch beibehalten werden. Nachdem aber festgestellt wurde, dass Pichia polyphyletisch ist, wurde die Art in die Gattung Ogataea gestellt und heißt daher gültig Ogataea angusta.[1] Es existieren drei unterschiedliche Stämme dieser Art von ungeklärter Verwandtschaft und unabhängiger Herkunft. Sie wurden seit dem Beginn der fünfziger Jahre des letzten Jahrhunderts in Bodenproben, im Darm von Insekten und in verdorbenem Orangensaft entdeckt. Sie weisen unterschiedliche Eigenschaften auf und sind entweder beliebte Objekte der wissenschaftlichen Grundlagenforschung oder werden nach gentechnischer Veränderung für die Produktion von Proteinen (Eiweißen) genutzt:
- Stamm CBS4732 (CCY38-22-2; ATCC34438, NRRL-Y-5445)
- Stamm DL-1 (NRRL-Y-7560; ATCC26012)
- Stamm NCYC495 (CBS1976; ATAA14754, NRLL-Y-1798).
Die Stämme CBS4732 und NCYY495 können miteinander gekreuzt werden, Stamm DL-1 ist mit den anderen beiden nicht kreuzbar. Die Stämme CBS4732 und DL-1 werden für die gentechnische Produktion von Proteinen eingesetzt, Stamm NCYC495 wird hauptsächlich für Untersuchungen der Nitratassimilation genutzt. Das Genom von CBS4732 wurde vollständig sequenziert.
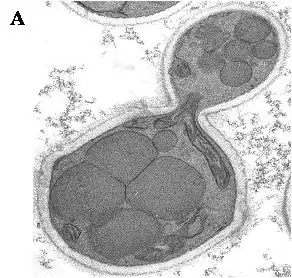
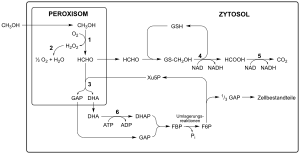
„H. polymorpha“ ist ein thermo-toleranter Mikroorganismus – einige Stämme können noch bei Temperaturen über 50 °C wachsen. Außerdem kann die Hefe Nitrat assimilieren (dies ist sehr ungewöhnlich für eine Hefe) und auf unterschiedlichen Zuckern, Glycerin oder auch Methanol wachsen (die besser bekannte Bäckerhefe wächst nur auf Traubenzucker und macht daraus Alkohol). Zellen, die bei erhöhter Temperatur wachsen, reichern Trehalose an, einen Zucker, der bei Insekten zu finden ist, und nutzen ihn zum Hitzeschutz. Trehalose ist zwar nicht für das Wachstum, aber für den Erwerb der Thermo-Toleranz notwendig. Die für die Synthese von Trehalose notwendigen Schritte wurden für diese Hefeart aufgeklärt und TPS1, das Gen für das Schlüsselenzym des Syntheseweges, isoliert und charakterisiert. Alle methylotrophen Hefearten weisen einen identischen Stoffwechselweg für die Nutzung von Methanol auf. Das Wachstum auf Methanol wird von einer massiven Zunahme von Peroxisomen, bestimmten Zellorganellen, begleitet (Abb. 1).
In ihnen finden die ersten Schritte des Methanolstoffwechsels statt. „H. polymorpha“ ist ein Modellorganismus, um die Funktionen von Peroxisomen und die den Funktionen zugrundeliegende Molekularbiologie zu studieren. Während des Wachstums auf Methanol werden bestimmte Enzyme des Stoffwechselweges in großen Mengen produziert, darunter MOX (Methanol Oxidase), FMDH (Formiat Dehydrogenase) und DHAS (Dihydroxyaceton Synthase). Ihre Anwesenheit wird durch Steuerung der Transkription (Bildung von mRNA) der entsprechenden Gene reguliert. In den bereits erwähnten verwandten Hefearten C. boidinii, P. methanolica, und K. phaffii (Pichia pastoris) ist diese Genexpression strikt von der Anwesenheit von Methanol abhängig, während in „H. polymorpha“ dies auch durch geeignete Mengen Glycerin oder unter Zuckerhungerbedingungen (Glucose Starvation) hervorgerufen wird.
Pichia angusta produziert Glycoproteine (Proteine, an die Zuckermoleküle kettenförmig angeheftet sind) – es gibt N- und O-verknüpfte Zuckerketten. Die endständigen Zucker (Mannose) werden in N-Ketten mit alpha-1,2 Bindungen verknüpft und nicht mit potenziell allergenen alpha-1,3-Bindungen wie z. B. in der Bäckerhefe Saccharomyces cerevisiae.
Pichia angusta bietet eine exzellente Plattform für die gentechnische Produktion von Proteinen, insbesondere von Pharmazeutika wie Insulin für Diabetiker, für Hepatitis-B-Impfstoffe oder für IFN alpha-2a zur Behandlung von Hepatitis C.
Die zuvor beschriebenen ungewöhnlichen Eigenschaften machen „H. polymorpha“ zu einer attraktiven Plattform für die gentechnische Produktion von Proteinen. Basierend auf gentechnisch veränderten „H. polymorpha“ Hefen gibt es bereits zahlreiche Produkte und Produktionsverfahren (in der Überschrift sind einige wichtige Beispiele genannt). Abkömmlinge der Stämme CBS4732 und DL-1 werden dafür eingesetzt. Weitere Hefearten für diese Anwendung sind unter anderem K. phaffii (Pichia pastoris), Arxula adeninivorans, Saccharomyces cerevisiae und andere.
Hefen sind Mikroorganismen, die sich in großen Fermentern in kurzer Zeit zu großen Zelldichten kultivieren lassen. „H. polymorpha“ ist ein sicherer Organismus, da er keine pyrogenen oder pathogenen Substanzen enthält. Hefen können Proteine ins Medium entlassen (sezernieren), da sie die dazu notwendigen Strukturen höherer Zellen, wie die des Menschen, aufweisen. Das Darmbakterium kann dies zum Beispiel nicht. „H. polymorpha“ stellt attraktive genetische Elemente für die effiziente Produktion von Proteinen zur Verfügung.
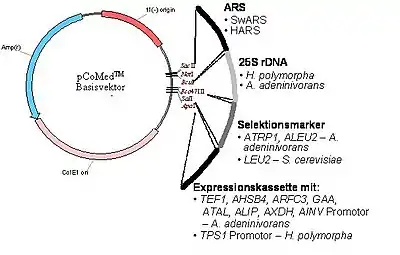
In Abb. 3 ist das generelle Schema eines Vektors dargestellt (ein ringförmiges DNA-Molekül für Verwandlung eines Hefestammes in einen genetisch veränderten Proteinproduzenten). Ein solcher Vektor (oder auch als Plasmid bezeichnet) muss verschiedene genetische Elemente enthalten:
- Einen „Selektionsmarker“, der notwendig ist, um einen transformierten Stamm von einem nicht-transformierten Hintergrund zu erkennen und zu selektionieren – dies kann z. B. erreicht werden, wenn ein derartiges genetische Element einen ursprünglich defizienten Stamm in die Lage zurückversetzt, unter Kulturbedingungen zu wachsen, in dem eine überlebensnotwendige Substanz, wie eine bestimmte Aminosäure fehlt, die dieser Stamm aber selbst nicht mehr produzieren kann.
- Bestimmte genetische Elemente für die Propagierung der DNA und für das zielgerichtete Einschleusen der fremden DNA an eine bestimmte Stelle der Chromosomen der aufnehmenden Hefezelle (ARS und/oder rDNA Sequenz).
- Ein Segment, das für die Produktion des erwünschten Proteins notwendig ist – eine Expressionskassette. Eine solche Kassette besteht aus einer Abfolge regulatorischer Elemente: zunächst enthält sie einen Promotor, der kontrolliert, wie viel und unter welchen Umständen eine nachfolgende Sequenz transkribiert (eine mRNA Kopie produziert) wird, und damit letztlich, wie viel und unter welchen Umständen ein erwünschtes Protein hergestellt wird. Das nachfolgende Segment ist variabel in Abhängigkeit von dem zu produzierenden Protein – es kann eine Gensequenz mit der Festlegung der Aminosäureabfolge für Insulin, Hepatitis B Antigene oder Interferon sein. Die Expressionskassette wird durch einen Terminator abgeschlossen, der einen korrekten Abschluss des Transkriptionsvorganges bewirkt. Die Promoterelemente des H. polymorpha Systems stammen von Genen, die stark exprimiert werden, z. B. von den bereits erwähnten MOX, FMD oder TPS1 Genen. Die Eigenschaften dieser Promotoren wie Stärke und Regulierbarkeit durch bestimmte Kohlenstoffquellen bleiben erhalten.
Anwendung
Die Plattform Pichia angusta wird von verschiedenen Biotechnologiefirmen genutzt, um Produktionsverfahren für wichtige Proteine zu entwickeln, unter anderem von den Unternehmen ARTES Biotechnology GmbH in Langenfeld, PharmedArtis in Aachen und dem Leibniz-Institut für Pflanzengenetik und Kulturpflanzenforschung (IPK).[3]
HPWN (Hansenula polymorpha worldwide network)
Viele akademische Forschungsgruppen weltweit studieren diesen Organismus. Im Jahr 2000 wurde eine wissenschaftliche Gesellschaft unter dem Namen HPWN (Hansenula polymorpha worldwide network) von Prof. Dr. Marten Veenhuis, Groningen, und Prof, Dr. Gerd Gellissen, Düsseldorf, gegründet. Alle zwei Jahre werden wissenschaftliche Treffen organisiert.[4]
Einzelnachweise
- Cletus Kurtzman, J.W. Fell, Teun Boekhout (Hrsg.): „The Yeasts: A Taxonomic Study, Volumen 1“; Elsevier, B.V. 2010: S. 685 ff.; ISBN 978-0-444-52149-1.
- Gerhard Steinborn, Erik Böer, Anja Scholz, Kristina Tag, Gotthard Kunze, Gerd Gellissen: Application of a wide-range yeast vector (CoMed™) system to recombinant protein production in dimorphic Arxula adeninivorans, methylotrophic Hansenula polymorpha and other yeasts. In: Microbial Cell Factories. Band 5, 2006, ISSN 1475-2859, S. 33, doi:10.1186/1475-2859-5-33.
- Arbeitsgruppe Hefegenetik. IPK, archiviert vom Original am 24. Februar 2015; abgerufen am 22. Mai 2013.
- Im Herbst 2008 ist dieses Treffen in Estland. Informationen zu dieser Gesellschaft und den Konferenzen können ab Sommer 2007 unter www.hansenula-network.com abgerufen werden.
Literatur
- Gellissen G (Ed) (2002) Hansenula polymorpha - biology and applications. Wiley-VCH, Weinheim ISBN 978-3-527-60235-3
- Gellissen G (Ed) (2005) Production of recombinant proteins - novel microbial and eukaryotic expression systems. Wiley-VCH, Weinheim ISBN 978-3-527-31036-4