Malacidine
Malacidine sind eine Gruppe von Lipopeptiden, die experimentell antibiotische Wirksamkeit zeigen. Der Name Malacidin wird abgeleitet von metagenomic acidic lipopeptide antibiotic (= „metagenomes saures Lipopeptid-Antibiotikum“) und der Endung „-cidin“.[1]
Vorkommen
Malacidine werden von Bakterien produziert, die man im Erdboden findet.
Chemische Struktur
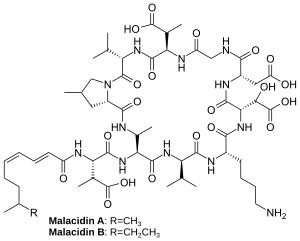
Malacidine sind eine Gruppe makrocyclischer Lipopeptide, die sich jeweils in der Kettenlänge der Lipid-Seitenkette unterscheiden.[2] Die Peptideinheit besteht aus vier nicht-proteinogenen Aminosäuren.[2] Malacidin A besitzt an der Acylgruppe (ein Lipid) eine Methylgruppe, während Malacidin B eine Ethylgruppe trägt.
Wirkung
Malacidine wirken gegen Gram-positive, nicht aber gegen Gram-negative Bakterien.[2][3] Ihre Wirkung bedingt die Anwesenheit von Calciumionen.
Im Laborversuch konnten mit Malacidinen resistente Krankheitserreger bekämpft werden, beispielsweise Vancomycin-resistente Bakterien und der Methicillin-resistente Bakterienstamm Staphylococcus aureus (MRSA) bei Hautinfektionen von Wunden.[2][3]
Durch Bindung an Calciumionen werden die Malacidine aktiviert. Der resultierende Komplex bindet an ein Lipid und wird zum bakteriellen Precursor einer Zellmembran, was zur Zerstörung der Zellmembran und schließlich zum Absterben des Bakteriums führt.[2][4] Die Malacidine gehören daher zur Klasse der calcium-abhängigen Antibiotika.[2][5] Die Entdeckung der Malacidine unterstützt die These, dass die Substanzklasse der calciumabhängigen Antibiotika größer ist, als bislang angenommen.[2]
Ob die Eigenschaften der Malacidine zu neuen antibiotischen Arzneimitteln führen können, ist derzeit noch ungewiss.[5]
Geschichte
Verschiedene Bodenproben wurden auf potentiell nützliche Wirkstoffe gescreent, eine geeignete Methode, um Primär- und Sekundärmetaboliten von Mikroorganismen zu untersuchen.[5][6]
Die Entdeckung der Malacidine wurde 2018 von Wissenschaftlern der Rockefeller University aus der Gruppe von Brad Hover und Sean Brady publiziert.[2] Die Gruppe hatte Antibiotika untersucht, die den calciumabhängigen Wirkstoff Daptomycin enthalten, aber sie fanden heraus, dass Variationen der Zellkultur unter Laborbedingungen ungünstig waren.[1] Stattdessen verwendete das Forscherteam einen genetischen Ansatz, der besser durchführbar war. Sie konzentrierten sich auf die Suche neuer biosynthetischer Gencluster (BGCs) und verwendeten Bakterien, um Sekundärmetaboliten zu erzeugen. Dafür extrahierten sie DNA von über 2000 Bodenproben, um eine Genbibliothek anzulegen, die die genetische Vielfalt der Umgebung des Mikrobioms wiedergibt. Sie produzierten einen Primer, um die Gene zu amplifizieren (vervielfachen), und stellten unter Verwendung der Polymerase-Kettenreaktion synthetisch Gencluster her, die dem Antibiotikum Daptomycin ähnlich sind. Anschließend testeten sie metagenomisch, dass diese Gene tatsächlich den erwarteten BGCs entsprechen. Einer der neuen BGCs war in 19 % der untersuchten Bodenproben, aber nicht oft in mikrobiologischen Sammlungen vorhanden. Dessen BGC wurden in einen anderen Bakterienstamm transformiert und kultiviert. Daraus wurden die Sekundärmetaboliten isoliert und analysiert.[2][1]
Einzelnachweise
- Melissa Healy: In soil-dwelling bacteria, scientists find a new weapon to fight drug-resistant superbugs. In: Los Angeles Times. 13. Februar 2018. Abgerufen am 13. Februar 2018.
- Bradley M. Hover et al.: Culture-independent discovery of the malacidins as calcium-dependent antibiotics with activity against multidrug-resistant Gram-positive pathogens. In: Nature Microbiology. 12. Februar 2018. doi:10.1038/s41564-018-0110-1. PMID 29434326.
- New antibiotic family discovered in dirt. In: BBC. 13. Februar 2018. Abgerufen am 13. Februar 2018.
- Anthony King: Soil search unearths new class of antibiotics. In: Chemistry World, 14. Februar 2018.
- Sarah Kaplan: A potentially powerful new antibiotic is discovered in dirt. In: The Washington Post. 13. Februar 2018. Abgerufen am 13. Februar 2018.
- Robert Lee Hotz: Scientists Unearth Hope for New Antibiotics. In: Wall Street Journal, 12. Februar 2018.