MHETase
Das Enzym MHETase ist eine Hydrolase, welche im Jahr 2016 entdeckt wurde und die Eigenschaft hat, vom Enzym PETase produzierte Mono-(2-hydroxyethyl)-terephthalsäure in Ethylenglycol und Terephthalsäure zu zerlegen.[1] Das Enzym entstand durch natürliche Evolution und kommt im ebenfalls 2016 entdeckten Bakterium Ideonella sakaiensis vor.[2]
| MHETase (Ideonella sakaiensis) | ||
|---|---|---|
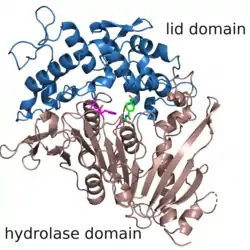
| ||
| Struktur der MHETase PDB 6QGA mit dem nicht hydrolysierbaren Substratanalog MHETA | ||
|
Vorhandene Strukturdaten: siehe UniProt | ||
| Masse/Länge Primärstruktur | 63 kDa / 596 Aminosäuren | |
| Sekundär- bis Quartärstruktur | α/β Hydrolase, Familie der Tannasen | |
| Bezeichner | ||
| Externe IDs | ||
| Enzymklassifikation | ||
| EC, Kategorie | 3.1.1.102, Esterase | |
| Reaktionsart | Hydrolyse aromatischer Ester | |
| Substrat | MHET (Mono-(2-hydroxyethyl)-terephthalsäure, Mono-hydroxyethyl-terephthalat) | |
| Produkte | Terephthalsäure und Ethylenglycol | |
| Vorkommen | ||
| Übergeordnetes Taxon | Bacteria › Proteobacteria › Betaproteobacteria › Burkholderiales › Ideonella | |
Chemische Reaktion
Das erste Enzym beim PET-Abbau, die PETase, spaltet den Kunststoff in das Zwischenprodukt MHET (Mono-(2-hydroxyethyl)-terephthalsäure) und geringe Mengen BHET (Bis-(2-hydroxyethyl)-terephthalsäure). MHETase hydrolysiert die Esterbindung von MHET unter Bildung von Terephthalsäure und Ethylenglycol.[1]
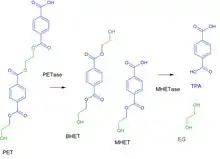
Neben dem natürlichen Substrat MHET wird das chromogene Substrat MpNPT, Mono-p-nitrophenyl-terephthalat, gut umgesetzt, was zur Bestimmung der enzymatischen Aktivität genutzt werden kann.[3] Ferulasäureester und Gallussäureester, die Substrate der nächsten Verwandten aus der Tannasefamilie, werden nicht umgesetzt. p-Nitrophenylester aliphatischer Monocarbonsäuren wie das meistverwendete Esterasesubstrat p-Nitrophenylacetat werden ebenfalls nicht umgesetzt.[1]
Struktur
Die Struktur wurde 2019 aufgeklärt[3] und zeigt die weit verbreitete Faltung einer α/β-Hydrolase. Nach Einteilung in der ESTHER Datenbank[4] gehört die MHETase im Block X zur Familie der Tannasen, die hauptsächlich Tannasen und Feruloylesterasen enthält. Außer der Hydrolasedomäne, die allen α/β-Hydrolase Enzymen gemein ist, besitzt die MHETase eine große Lid-Domäne ('Deckel'), die für die Substratspezifität verantwortlich ist. Die Reste der katalytischen Triade, Ser225, His528 und Asp492, liegen in der Hydrolasedomäne, die Reste Arg411, Ser416 und Ser419 in der Liddomäne sind mit ihren Wasserstoffbrückenbindungen zur Carboxylatgruppe essentiell für die Spezifität auf Terephthalsäureester.[3]
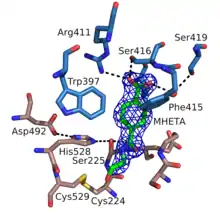
Weblinks
Einzelnachweise
- S Yoshida, K Hiraga, K Takehana, I Taniguchi, H Yamaji, Y Maeda, K Toyohara, K Miyamoto, Y Kimura, K Oda: A bacterium that degrades and assimilates poly(ethylene terephthalate). In: Science. 351, Nr. 6278, März 2016, S. 1196–9. doi:10.1126/science.aad6359. PMID 26965627.
- Michael: Erstes kunststoffabbauendes Bakterium entdeckt. Spektrum.de, 16. März 2016, abgerufen am 8. April 2016.
- Gottfried J. Palm, Lukas Reisky, Dominique Böttcher, Henrik Müller, Emil A.P. Michels, Miriam C. Walczak, Leona Berndt, Manfred Weiss, Uwe Bornscheuer, Gert Weber: Structure of the plastic-degrading Ideonella sakaiensis MHETase bound to a substrate. In: Nature Communications. 10, Nr. 1, April 2019, ISSN 2041-1723, S. 1717-. doi:10.1038/s41467-019-09326-3.
- L Renault, V Nègre, T Hotelier, X Cousin, P Marchot, A Chatonnet: New friendly tools for users of ESTHER, the database of the alpha/beta-hydrolase fold superfamily of proteins. In: Chem. Biol. Interact.. 157–158, Dezember 2005, S. 339–43. doi:10.1016/j.cbi.2005.10.100. PMID 16297901.