3D-SIM-Mikroskop
Das 3D-SIM-Mikroskop (engl. 3D structured illumination microscope) realisiert eine weiterentwickelte Form der Lichtmikroskopie, die Auflösungen jenseits der von Ernst Abbe beschriebenen Auflösungsgrenze ermöglicht. Das Konzept der 3D-SIM-Mikroskopie wurde erstmals von Lukosz und Marchand 1963 vorgestellt[1][2] und von einem Team um Mats G. L. Gustafsson und John W. Sedat an der University of California, San Francisco weiter entwickelt.[3] Kommerzielle Versionen werden von Applied Precision als „OMX“[4], von Carl Zeiss als „ELYRA S.1 bzw. PS.1“[5], sowie von Nikon als „N-SIM“[6] angeboten.
Funktionsprinzip
Die 3D-SIM Mikroskopie nutzt eine räumlich modulierte strukturierte Beleuchtung (meist in Form eines Liniengitters) zur Fluoreszenzanregung. Hierbei müssen mehrere Bilder der Probe aufgenommen werden, wobei das Beleuchtungsmuster in der Probe von einem zum nächsten aufgenommenen Bild verschoben werden muss. Dies wird für mehrere Ebenen eines 3D Volumens wiederholt. Ein Bild des Objekts (mit gesteigerter Auflösung) kann dann aus diesen gespeicherten Rohbildern berechnet werden. Die Auflösungssteigerung basiert hierbei auf dem Prinzip des Moiré-Effekts, wobei die detektierten Bilder als Überlagerung des (bekannten) Beleuchtungsmusters und der (unbekannten) Objektfrequenzen interpretiert werden. Im Fall eines Streifenmusters wird die Phasenlage des Musters über eine Periode in mindestens 5 Schritten verschoben. Da Streifenmuster nur in einer Richtung moduliert sind, müssen Rohbilder für mindestens drei Orientierungen des Musters aufgenommen werden. Im Gegensatz zu 2D-SIM Varianten werden bei 3D-SIM Rohbilder für ein ganzes Volumen abgespeichert und die Bilder des gemessenen Volumens zur Berechnung eines Volumens mit gesteigerter Auflösung benutzt. Damit kann die mikroskopische Auflösung in allen drei Raumrichtungen verdoppelt werden.
Leistung
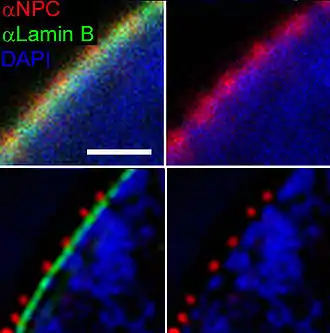
Beim OMX reicht die erzielbare Auflösung von 105 nm bei einer Beleuchtung mit Licht der Wellenlänge von 405 nm und bis 165 nm bei einer Wellenlänge von 593 nm. Dies entspricht etwa einer Halbierung der bisherigen Auflösungsgrenze von ca. 200 nm.[7] Mit Hilfe der 3D-SIM-Mikroskopie konnten Forscher erstmals Teile der Zellkern-Hülle wie Membranen und Poren sehen, die im konventionellen Lichtmikroskop nicht erkennbar waren; ebenso waren auf der Oberfläche von Chromosomen bisher nicht sichtbare Details erkennbar.[8][9]
Vorteile im Vergleich zu anderen Verfahren
Zwar ist mit Hilfe der Elektronenmikroskopie eine weit höhere Auflösung erzielbar, Lebendzell-Mikroskopie und mehrfarbige Abbildungen sind mit der Methode jedoch nicht möglich. Ein Vorteil der 3D-SIM-Mikroskopie im Vergleich zu einigen anderen neuartigen lichtmikroskopischen Verfahren jenseits der klassischen Auflösungsgrenze liegt darin, dass normale fluoreszenzmikroskopische Präparate verwendet werden können.
- Bilder von Zellkernen und Mitose-Stadien, die mit 3D-SIM Mikroskopie aufgenommen wurden.
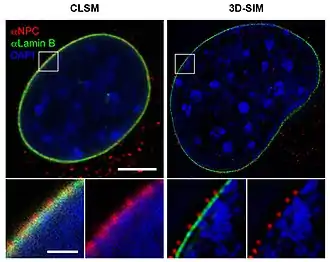 Vergleich Konfokalmikroskopie mit 3D-SIM
Vergleich Konfokalmikroskopie mit 3D-SIM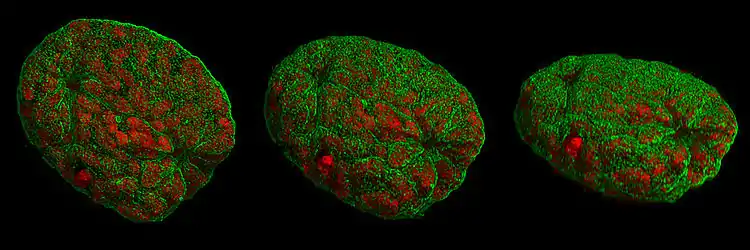
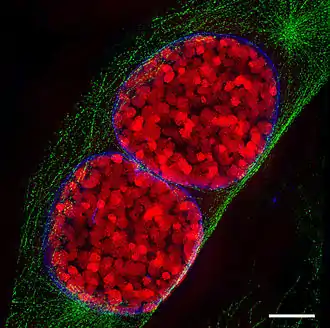 Zwei Mauszellkerne im Prophase-Stadium
Zwei Mauszellkerne im Prophase-Stadium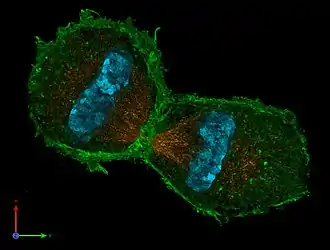 Mauszelle in der Telophase
Mauszelle in der Telophase
Weblinks
- Luise Dirscherl: Panoramablick in die Zelle - Mikroskopieren in 3D, mehrfarbig und in hoher Auflösung. In: idw-online.de. Ludwig-Maximilians-Universität München, 5. Juni 2008.
Einzelnachweise
- W. Lukosz, M. Marchand: Optischen Abbildung Unter Überschreitung der Beugungsbedingten Auflösungsgrenze. In: Optica Acta. 10, Nr. 3, 1963, S. 241–255. doi:10.1080/713817795.
- Carl Zeiss MicroImaging GmbH: Superresolution Structured Illumination Microscopy (SR-SIM). White Paper, Carl Zeiss BioSciences, Jena Location 2010 (PDF).
- M. G. Gustafsson, L. Shao, P. M. Carlton et al: Three-dimensional resolution doubling in wide-field fluorescence microscopy by structured illumination. In: Biophysical Journal. 94, Nr. 12, Juni 2008, S. 4957–4970. doi:10.1529/biophysj.107.120345. PMID 18326650.
- API DeltaVision OMX. Appliedprecision.com. Abgerufen am 23. Juni 2010.
- Carl Zeiss MicroImaging GmbH: ELYRA Enter the World of Superresolution. Carl Zeiss BioSciences, Jena Location 2011 (PDF).
- Nikon N-SIM (Memento vom 4. März 2016 im Internet Archive).
- I. M. Dobbie, E. King, R. M. Parton, P. M. Carlton, J. W. Sedat, J. R. Swedlow, I. Davis: OMX: A New Platform for Multimodal, Multichannel Wide-Field Imaging. In: Cold Spring Harbor Protocols. August 2011, S. 899–909, doi:10.1101/pdb.top121, PMID 21807861.
- L. Schermelleh, P. M. Carlton, S. Haase et al: Subdiffraction multicolor imaging of the nuclear periphery with 3D structured illumination microscopy. In: Science. 320, Nr. 5881, Juni 2008, S. 1332–1336. doi:10.1126/science.1156947. PMID 18535242.
- Carlton PM: Three-dimensional structured illumination microscopy and its application to chromosome structure. In: Chromosome research : an international journal on the molecular, supramolecular and evolutionary aspects of chromosome biology. 16, Nr. 3, 2008, S. 351–365. doi:10.1007/s10577-008-1231-9. PMID 18461477.