Single Base Extension
Unter Single Base Extension (SBE) wird eine biochemische Reaktion verstanden, die der Genotypisierung von SNPs (Single Nucleotide Polymorphisms) dient.[1] Der SBE geht im Regelfall eine PCR (Polymerase-Kettenreaktion) voraus.
Funktionsprinzip der SBE
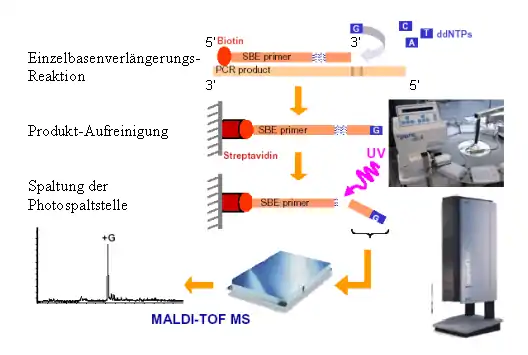
Nach Durchführung von PCR und Verdau liegt amplifizierte (vervielfältigte) DNA der interessierenden Regionen des Genoms vor. Unterscheidungsmerkmale zwischen den Genomen verschiedener Personen sind sogenannte Polymorphismen. Polymorphismen, von denen nur eine einzelne Base betroffen ist, werden Einzel-Basen-Mutationen oder SNPs genannt. Unterschiedliche Basen können zu unterschiedlichen Genprodukten oder veränderter Genexpression führen. Dies kann einen Risiko- oder auch Protektivfaktor, insbesondere für komplexe Erkrankungen, darstellen. Die SBE dient dazu zu bestimmen, welche Base bei der jeweils untersuchten Person vorliegt. Mittels SBE lässt sich also der Genotyp bestimmen. Hierzu werden die verdauten PCR-Produkte mit einem speziellen SBE-Primer versetzt. Die Gesamtreaktion der SBE ist so konzipiert, dass der SBE-Primer nur um eine einzige Base (die komplementär zum interessierenden SNP ist) verlängert wird.[2] Dies geschieht, indem man dem Reaktionsansatz ddNTPs (Didesoxynukleosidtriphosphate) statt dNTPs wie bei der PCR zusetzt. An ein ddNTP kann auf Grund seiner biochemischen Struktur keine weitere Base durch Polymerase angelagert werden. Die Elongationsphase, die analog zur PCR erfolgt, beschränkt sich also auf ein einziges Nukleotid. Mittels anschließender Messung im Massenspektrometer ist es möglich, festzustellen, um welche Base der SBE-Primer verlängert wurde, da sich die vier Basen in ihrer Masse unterscheiden. Die entsprechend komplementäre Base entspricht dann dem gesuchten SNP-Typ. Auch bei der SBE kommt dabei Multiplexing, d. h. die gleichzeitige Untersuchung verschiedener SNPs, zum Einsatz. Je SNP wird ein spezifischer SBE-Primer eingesetzt.
Aufbau des SBE-Primers
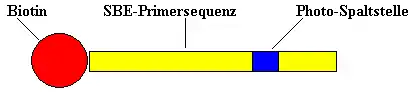
Der SBE-Primer besteht aus einem Biotin-Rest, der SBE-Primersequenz und einer Photo-Spaltstelle. Die Primersequenz wird so konstruiert, dass sie sich an einen spezifischen Bereich der DNA anlagert und genau eine Base vor dem SNP endet. Die Photo-Spaltstelle ist ein Bereich, der besonders empfindlich auf UV-Licht reagiert.
Durchführung der SBE
Der Beginn der SBE erfolgt analog zur PCR: Es wird ein Reaktionsgemisch hergestellt. Das Gemisch besteht aus dem SBE-Primer, ddNTPs, Magnesiumchlorid, einer Polymerase und zweifach destilliertem Wasser. Dieses Gemisch wird auf das verdaute PCR-Produkt gegeben. Die eigentliche Reaktion findet in einem Thermocycler statt. Im Thermocycler findet die Einzelbasenverlängerung statt. Am Ende dieses Schrittes liegen im Reaktionsgemisch der um eine Base verlängerte SBE-Primer, das unveränderte PCR-Produkt, verbliebene ddNTPs, sowie die übrigen Bestandteile des ursprünglichen Gemisches vor. Mit Ausnahme des verlängerten SBE-Primers stellt all dies für die nachfolgende Messung im Massenspektrometer eine störende Verunreinigung dar. Entsprechend folgt eine Aufreinigung.
Aufreinigung der SBE-Produkte
Hierzu wird das Reaktionsgemisch in die Kavitäten einer Streptavidin-Platte gegeben, wo der Biotin-Rest des SBE-Primers eine starke nichtkovalente Bindung mit dem Streptavidin eingeht und an diesem haftenbleibt. Die übrigen Bestandteile des Reaktionsgemisches werden mittels verschiedener Waschpuffer entfernt. Im letzten Schritt befinden sich nur noch hochreines Wasser und der am Streptavidin haftende SBE-Primer in der Kavität, die nun mit UV-Licht bestrahlt wird. Hierdurch bricht der SBE-Primer an der Photo-Spaltstelle. Die Masse des um eine Base verlängerten Primer-Bruchstückes kann nun im Massenspektrometer bestimmt werden.
Einzelnachweise
- G. A. Denomme: Single base extension in multiplex blood group genotyping. In: Methods in molecular biology (Clifton, N.J.). Band 496, 2009, S. 15–24, ISSN 1064-3745. doi:10.1007/978-1-59745-553-4_2. PMID 18839101.
- L. Kaderali: Primer design for multiplexed genotyping. In: Methods in molecular biology (Clifton, N.J.). Band 402, 2007, S. 269–286, ISSN 1064-3745. doi:10.1007/978-1-59745-528-2_13. PMID 17951800.